Genética e Arqueogenética do Sul da Ásia - Genetics and archaeogenetics of South Asia
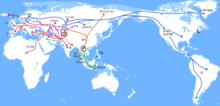
Genética e Arqueogenética do Sul da Ásia é o estudo da genética e da arqueogenética dos grupos étnicos do Sul da Ásia . O objetivo é desvendar a história genética desses grupos . A posição geográfica do Sul da Ásia torna sua biodiversidade importante para o estudo da dispersão inicial de humanos anatomicamente modernos pela Ásia .
Estudos baseados em variações do DNA mitocondrial ( mtDNA ) relataram unidade genética em várias subpopulações do sul da Ásia, mostrando que a maioria dos nós ancestrais da árvore filogenética de todos os tipos de mtDNA se originou no sul da Ásia. As conclusões dos estudos baseados na variação do cromossomo Y e na variação do DNA autossômico foram variadas.
Estudos recentes do genoma mostram que os sul-asiáticos são descendentes de um componente indígena do sul da Ásia (denominado antigos ancestrais índios sul , "AASI", distantemente relacionados com a Andamanese , papuas , e nativos australianos), e mais tarde, chegar West-euro-asiático (/ Oriente Europeia Relacionados ao Leste) e componentes da Eurásia do Leste (relacionados ao Leste Asiático), respectivamente, em vários graus.
Estudos genômicos descreveram a paisagem genética do Sul da Ásia como um composto de componentes exógenos da Eurásia Ocidental e Eurásia Oriental que se misturaram aos grupos indígenas do Sul da Ásia para criar os modernos sul-asiáticos. O componente de ancestralidade relacionado ao Leste Asiático está formando a principal ancestralidade entre populações específicas no sopé do Himalaia e no nordeste da Índia e é geralmente distribuído por todo o subcontinente indiano , com pico entre os grupos de língua austro-asiática , bem como entre cingaleses e bengalis .
Foi descoberto que o nó ancestral da árvore filogenética de todos os tipos de mtDNA ( haplogrupos de DNA mitocondrial ) tipicamente encontrados na Ásia Central, Ásia Ocidental e Europa também podem ser encontrados no Sul da Ásia em frequências relativamente altas. Estima-se que a divergência inferida desse nó ancestral comum tenha ocorrido pouco menos de 50.000 anos atrás, apoiando um evento "Fora da Índia" para as populações da Eurásia Ocidental, como os europeus, durante o início do Paleolítico. Na índia, as grandes linhagens maternas são vários M subclades, seguido por R e U sublinhagens. Os tempos de coalescência desses haplogrupos mitocondriais foram estimados até hoje em 50.000 BP.
As principais linhagens paternas de índios, representadas pelos cromossomos Y , são os haplogrupos R1a1 , R2 , H , L , J2 e O2a . Alguns pesquisadores argumentaram que o Haplogrupo R1a1 (M17) do Y-DNA é de origem autóctone do sul da Ásia. No entanto, propostas para uma origem de estepe da Ásia Central / Eurásia para R1a1 também são bastante comuns e apoiadas por vários estudos mais recentes.
Visão geral
Sugere-se que a população indígena do Sul da Ásia, às vezes denominada "AASI", tenha se originado de uma população inicial da Dispersão do Sul , que também deu origem aos povos Andamaneses , povos indígenas da Nova Guiné (papuas) e indígenas australianos . A população indígena do sul da Ásia tornou-se geneticamente isolada de outras populações desde aproximadamente 45.000 anos antes de Cristo . A ancestralidade indígena do sul da Ásia (AASI) nos modernos sul-asiáticos é combinada, em vários graus, com os componentes mais recentes da Eurásia Ocidental e Oriental do período Neolítico e da Idade do Bronze . O povo tribal Paniya é, ao lado do povo Irula e do povo Soliga , o melhor representante da ancestralidade indígena do sul da Ásia.
Propõe-se que a ancestralidade mais antiga da Eurásia Ocidental tenha chegado durante o período Neolítico ou já durante o Holoceno , em conjunto com a chegada de componentes relacionados ao Leste Asiático durante o início do período Neolítico com grupos Austroasiático e Tibeto-Birmanês do Sudeste Asiático e do Leste Asiático, respectivamente .
mtDNA
Os haplogrupos de mtDNA mais frequentes no Sul da Ásia são M , R e U (onde U é descendente de R). Defendendo o "modelo rival do cromossomo Y" de longo prazo, Stephen Oppenheimer acredita que é altamente sugestivo que a Índia seja a origem dos haplogrupos eurasianos do mtDNA que ele chama de "Eves Eurasianas". De acordo com Oppenheimer, é altamente provável que quase todas as linhagens maternas humanas na Ásia Central, Oriente Médio e Europa descendam de apenas quatro linhagens de mtDNA que se originaram no Sul da Ásia 50.000–100.000 anos atrás.
Macrohaplogrupo M
O macrohaplogrupo M , considerado um aglomerado de linhagens maternas proto-asiáticas, representa mais de 60% do MtDNA do sul da Ásia.
O macrohaplótipo M na Índia inclui muitos subgrupos que diferem profundamente de outras sub-linhagens no Leste Asiático, especialmente as populações mongolóides. As raízes profundas da filogenia M determinam claramente a relíquia das linhagens do sul da Ásia em comparação com outras sub-linhagens M (no leste da Ásia e em outros lugares), sugerindo origem 'in situ' desses sub-haplogrupos no sul da Ásia, mais provavelmente na Índia. Essas linhagens de enraizamento profundo não são específicas de um idioma e estão espalhadas por todos os grupos linguísticos da Índia.
Praticamente todas as linhagens modernas de MtDNA M da Ásia Central parecem pertencer aos subtipos da Ásia Central ( Mongol ), e não aos subtipos do haplogrupo M da Ásia Meridional, o que indica que nenhuma migração em grande escala das atuais populações de língua turca da Ásia Central ocorreu para a Índia. A ausência do haplogrupo M nos europeus, em comparação com sua frequência igualmente alta entre sul-asiáticos, asiáticos do leste e em algumas populações da Ásia Central, contrasta com as tendências da Eurásia Ocidental das linhagens paternas do sul da Ásia.
A maioria das fronteiras do mtDNA existentes no sul e no sudoeste da Ásia provavelmente foram moldadas durante a colonização inicial da Eurásia por humanos anatomicamente modernos.
| Haplogrupo | Subclados importantes | Populações |
|---|---|---|
| M2 | M2a, M2b | Em todo o continente, com baixa presença no pico do noroeste em Bangladesh, Andhra Pradesh, na costa de Tamil Nadu e no Sri Lanka |
| M3 | M3a | Concentrado no noroeste da Índia, o mais alto entre os parses de Mumbai |
| M4 | M4a | Picos no Paquistão, Caxemira e Andhra Pradesh |
| M6 | M6a, M6b | Caxemira e perto da costa da Baía de Bengala, Sri Lanka |
| M18 | Em todo o Sul da Ásia, com pico em Rajasthan e Andhra Pradesh |
|
| M25 | Moderadamente frequente em Kerala e Maharashtra, mas pouco frequente em outras partes da Índia |
Macrohaplogrupo R
O macrohaplogrupo R (uma subdivisão muito grande e antiga do macrohaplogrupo N ) também é amplamente representado e é responsável pelos outros 40% do MtDNA do sul da Ásia. Uma subdivisão muito antiga e mais importante dela é o haplogrupo U que, embora também esteja presente na Eurásia Ocidental , possui vários subclados específicos do Sul da Ásia.
Os haplogrupos do sul da Ásia mais importantes dentro de R:
| Haplogrupo | Populações |
|---|---|
| R2 | Distribuído amplamente em todo o subcontinente |
| R5 | amplamente distribuído na maior parte da Índia. Picos na costa sudoeste da Índia |
| R6 | generalizado a taxas baixas em toda a Índia. Picos entre tâmeis e caxemires |
| C | Encontrado em estados do noroeste. Picos em Gujarat, Punjab e Caxemira, a frequência é baixa em outros lugares. |
Haplogrupo U
O haplogrupo U é um sub-haplogrupo do macrohaplogrupo R. A distribuição do haplogrupo U é uma imagem espelhada daquela do haplogrupo M: o primeiro não foi descrito até agora entre os asiáticos orientais, mas é frequente nas populações europeias e também entre os sul-asiáticos. As linhagens U do sul da Ásia diferem substancialmente daquelas da Europa e sua coalescência a um ancestral comum também remonta a cerca de 50.000 anos.
| Haplogrupo | Populações |
|---|---|
| U2 * | (um parahaplogrupo ) é esparsamente distribuído, especialmente na metade norte do Sul da Ásia.
Também é encontrado no sudoeste da Arábia. |
| U2a | mostra densidade relativamente alta no Paquistão e no noroeste da Índia, mas também em Karnataka, onde atinge sua densidade mais alta. |
| U2b | tem a maior concentração em Uttar Pradesh, mas também é encontrada em muitos outros lugares, especialmente em Kerala e Sri Lanka.
Também é encontrado em Omã. |
| U2c | é especialmente importante em Bangladesh e em Bengala Ocidental. |
| U2l | é talvez o mais importante numericamente entre os subclados U no Sul da Ásia, atingindo concentrações especialmente altas (mais de 10%) em Uttar Pradesh, Sri Lanka, Sindh e partes de Karnataka. Também tem alguma importância em Omã. O haplogrupo U2i do mtDNA é apelidado de "Western Eurasian" em Bamshad et al. estudo, mas "Leste da Eurásia (principalmente da Índia)" em Kivisild et al. estude. |
| U7 | este haplogrupo tem uma presença significativa em Gujarat, Punjab e Paquistão. A possível pátria deste haplogrupo abrange Gujarat (frequência mais alta, 12%) e o Irã porque a partir daí sua frequência diminui abruptamente para o leste e para o oeste. |
Cromossomo Y
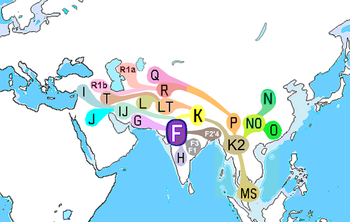
Os principais haplogrupos de DNA do cromossomo Y do sul da Ásia são H , J2 , L , R1a1 , R2 e O-K18 . Suas origens geográficas são listadas a seguir, de acordo com a bolsa mais recente:
| Principais linhagens cromossômicas Y do sul da Ásia: | H | J2 | eu | R1a | R2 |
|---|---|---|---|---|---|
| Basu et al. (2003) | sem comentários | sem comentários | sem comentários | Ásia Central | sem comentários |
| Kivisild et al. (2003) | Índia | Ásia Ocidental | Índia | Ásia Meridional e Ocidental | Centro-Sul da Ásia |
| Cordaux et al. (2004) | Índia | Ásia Ocidental ou Central | Oriente médio | Ásia Central | Centro-Sul da Ásia |
| Sengupta et al. (2006) | Índia | Oriente Médio e Ásia Central | Sul da Índia | Norte da Índia | Norte da Índia |
| Thanseem et al. (2006) | Índia | O levante | O Oriente Médio | Ásia Meridional e Central | Ásia Meridional e Central |
| Sahoo et al. (2006) | sul da Asia | O oriente próximo | sul da Asia | Sul ou oeste da ásia | sul da Asia |
| Mirabal et al. (2009) | sem comentários | sem comentários | sem comentários | Noroeste da Índia ou Ásia Central | sem comentários |
| Zhao et al. (2009) | Índia | O Oriente Médio | O Oriente Médio | Ásia Central ou Eurásia Ocidental | Ásia Central ou Eurásia Ocidental |
| Sharma et al. (2009) | sem comentários | sem comentários | sem comentários | sul da Asia | sem comentários |
| Thangaraj et al. (2010) | sul da Asia | O oriente próximo | O oriente próximo | sul da Asia | sul da Asia |
Haplogrupo H
O haplogrupo H (Y-DNA) é encontrado com alta frequência no sul da Ásia e é considerado a principal linhagem paterna indígena. H é hoje raramente encontrado fora do sul da Ásia, mas é comum entre as populações descendentes do sul da Ásia, como os Romanis , particularmente o subgrupo H-M82. H também foi encontrado em algumas amostras antigas da Europa e ainda hoje é encontrado com uma frequência baixa em certos europeus do sudeste e árabes do Levante . O Haplogrupo H é freqüentemente encontrado entre as populações da Índia , Sri Lanka , Nepal , Paquistão e Maldivas . Todos os três ramos do Haplogrupo H (Y-DNA) são encontrados no sul da Ásia.
Acredita-se que o Haplogrupo H tenha surgido no sul da Ásia entre 30.000 e 40.000 anos atrás. Seu provável local de introdução é o sul da Ásia, uma vez que ali está concentrado. Parece representar o principal haplogrupo do cromossomo Y dos habitantes paleolíticos do sul da Ásia e da Europa, respectivamente. Alguns indivíduos no sul da Ásia também mostraram pertencer ao muito mais raro subclado H3 (Z5857). O haplogrupo H não é de forma alguma restrito a populações específicas. Por exemplo, H é possuído por cerca de 28,8% das castas indo-arianas. e nas tribos cerca de 25–35%.
Haplogrupo J2
O haplogrupo J2 está presente no Sul da Ásia principalmente como J2a-M410 e J2b-M102, desde os tempos neolíticos (9500 YBP). Os clados J2 atingem frequências de pico no noroeste e sul da Índia e são encontrados em 19% nas castas do sul da Índia, 11% nas castas do norte da Índia e 12% no Paquistão. No sul da Índia , a presença de J2 é maior entre as castas médias com 21%, seguida pelas castas superiores com 18,6% e pelas castas inferiores com 14%. Entre os grupos de castas, a maior frequência de J2-M172 é observada entre Tamil Vellalars do sul da Índia, com 38,7%. J2 também está presente nas tribos e tem uma frequência de 11% nas tribos austro-asiáticas. Entre os tribais austro-asiáticos, o J2 predominante ocorre em Lodha (35%). J2 também está presente na tribo das colinas do sul da Índia Toda com 38,46%, na tribo Andh de Telangana com 35,19% e na tribo Kol de Uttar Pradesh com uma frequência de 33,34%. O haplogrupo J-P209 foi considerado mais comum em muçulmanos xiitas da Índia , dos quais 28,7% pertencem ao haplogrupo J, com 13,7% em J-M410, 10,6% em J-M267 e 4,4% em J2b ( Eaaswarkhanth 2009 ) .
Em Paquistão , foram observadas as maiores frequências de J2-M172 entre o pársis a 38,89%, o dravidiano -Falando Brahuis a 28,18% e as Makrani Balochs a 24%. Também ocorre em 18,18% em Makrani Siddis e em 3% em Karnataka Siddis .
J2-M172 é encontrado com uma frequência geral de 10,3% entre o povo cingalês do Sri Lanka . Nas Maldivas , constatou-se que 20,6% da população das Maldivas tinha haplogrupo J2 positivo.
Haplogrupo L
De acordo com o Dr. Spencer Wells , o L-M20 se originou na região de Pamir Knot no Tajiquistão e migrou para o Paquistão e Índia ca. 30.000 anos atrás. No entanto, a maioria dos outros estudos propôs uma origem do sul da Ásia para L-M20 e associou sua expansão com o vale do Indo (~ 7.000 YBP). Existem três sub-ramos do haplogrupo L: L1-M76 (L1a1), L2-M317 (L1b) e L3-M357 (L1a2), encontrados em vários níveis no Sul da Ásia.
Índia
O haplogrupo L mostra o tempo de expansão neolítica. O clado está presente na população indiana com uma frequência geral de ca. 7–15%. O haplogrupo L tem uma frequência mais alta entre as castas do sul da Índia (cerca de 17–19%) e atinge 68% em algumas castas em Karnataka, mas é um pouco mais raro nas castas do norte da Índia (cerca de 5–6%). A presença do haplogrupo L é bastante rara entre os grupos tribais (ca. 5,6–7%), no entanto 14,6% foram observados entre os Chenchus .
Entre os grupos regionais e sociais, frequências moderadas a altas foram observadas em Konkanastha Brahmins (18,6%), Punjabis (12,1%), Gujaratis (10,4%), Lambadis (17,1%) e Jats (36,8%).
Paquistão
No Paquistão, os subclados L1-M76 e L3-M357 de L-M20 atingem frequências gerais de 5,1% e 6,8%, respectivamente. O haplogrupo L3 (M357) é freqüentemente encontrado entre burusho (aprox. 12%) e pashtuns (aprox. 7%). Sua maior frequência pode ser encontrada no sudoeste da província de Baluchistão , ao longo da costa de Makran (28%) até o delta do rio Indo . L3a (PK3) é encontrado em aproximadamente 23% de Nuristani no noroeste do Paquistão .
O clado está presente em distribuição moderada entre a população geral do Paquistão (14% aproximadamente).
Sri Lanka
Em um estudo, 16% dos cingaleses foram considerados positivos para o Haplogrupo L-M20. Em outro estudo, 18% pertenciam a L1.
Haplogrupo R1a1
No sul da Ásia, R1a1 foi observado frequentemente com alta frequência em vários grupos demográficos, bem como com a maior diversidade de STR , o que leva alguns a vê-lo como o locus de origem.
Enquanto R1a originou ca. 22.000 a 25.000 anos atrás, seu subclado M417 (R1a1a1) diversificou ca. 5.800 anos atrás. A distribuição de M417-subclados R1-Z282 (incluindo R1-Z280) na Europa Central e Oriental e R1-Z93 na Ásia sugere que R1a1a se diversificou nas estepes da Eurásia ou no Oriente Médio e região do Cáucaso . O lugar de origem desses subclados desempenha um papel no debate sobre as origens dos indo-europeus . Embora o haplogrupo R seja comumente encontrado entre os eurasianos ocidentais, estudos recentes sugerem que sua linhagem ancestral ( Haplogrupo P ) descendia de uma população sul-euro-asiática no sudeste da Ásia . Hoje é comumente encontrado entre os povos Aeta , Jahai e alguns povos Andamaneses .
Índia
Na Índia , uma alta porcentagem deste haplogrupo é observada nos Brahmins de Bengala Ocidental (72%) a leste, Gujarat Lohanas (60%) a oeste, Khatris (67%) no norte e Iyengar Brahmins (31%) em o sul. Ele também foi encontrado em vários sul da Índia Dravidian -Falando tribais , incluindo o Kotas (41%) de Tamil Nadu, Chenchu (26%) e Valmikis de Andhra Pradesh , bem como o Yadav e Kallar de Tamil Nadu sugerindo que M17 é generalizada essas tribos de índios do sul. Além desses, estudos mostram porcentagens elevadas em grupos regionalmente diversos, como Manipuris (50%) ao extremo nordeste e entre Punjabis (47%) ao extremo noroeste.
Paquistão
No Paquistão, é encontrada em 71% entre os Mohanna da província de Sindh ao sul e 46% entre os Baltis de Gilgit-Baltistan ao norte.
Sri Lanka
23% dos cingaleses de uma amostra de 87 indivíduos foram considerados R1a1a (R-SRY1532) positivos de acordo com uma pesquisa de 2003.
Maldivas
Nas Maldivas , 23,8% da população das Maldivas tinha R1a1a (M17) positivo.
Nepal
Pessoas na região de Terai , Nepal mostram R1a1a em 69%.
Haplogrupo R2
No Sul da Ásia, a frequência da linhagem R2 e R2a é de cerca de 10–15% na Índia e Sri Lanka e 7–8% no Paquistão. Pelo menos 90% dos indivíduos com R-M124 estão localizados no Sul da Ásia. Também é relatado no Cáucaso e na Ásia Central com uma frequência menor. Um estudo genético de Mondal et al. em 2017 concluiu que o Haplogroup R2 se originou no norte da Índia e já estava presente antes da migração das estepes.
Índia
Entre os grupos regionais, é encontrada entre West Bengalis (23%), Hindus de Nova Delhi (20%), Punjabis (5%) e Gujaratis (3%). Entre os grupos tribais, Karmalis da Bengala Ocidental apresentou o maior índice com 100%, seguido por Lodhas (43%) para o leste, enquanto Bhil de Gujarat no oeste ficou com 18%, Tharus do norte mostrou isso com 17%, o Chenchu e Pallan do sul estavam em 20% e 14%, respectivamente. Entre os grupos de castas, altas porcentagens são mostradas por Jaunpur Kshatriyas (87%), Kamma (73%), Bihar Yadav (50%), Khandayat (46%) e Kallar (44%).
Também é significativamente elevada em muitos Brahmin grupos incluindo panjabi Brâmanes (25%), Bengala Brâmanes (22%), Konkanastha Brâmanes (20%), Chaturvedis (32%), Bhargavas (32%), caxemire Pandits (14%) e Brahmins Lingayat (30%).
Os muçulmanos do norte da Índia têm uma frequência de 19% ( sunitas ) e 13% ( xiitas ), enquanto Dawoodi Bohra Muslim no estado ocidental de Gujarat tem uma frequência de 16% e os muçulmanos Mappila do sul da Índia têm uma frequência de 5%.
Paquistão
O haplogrupo R2 é encontrado em 71% da população de Burusho . Entre o povo Hunza é encontrada em 18% enquanto os parsis mostram em 20%.
Sri Lanka
38% dos cingaleses do Sri Lanka foram considerados R2 positivos, de acordo com uma pesquisa de 2003.
Maldivas
12% da população das Maldivas tem R2.
Nepal
No Nepal, as porcentagens de R2 variam de 2% a 26% em diferentes grupos em vários estudos. Newars mostram uma frequência significativamente alta de 26%, enquanto as pessoas de Kathmandu mostram isso em 10%.
Haplogrupo O2a (O-K18)
No sul da Ásia, a frequência média de O2a ou O-K18 é 5% —15% na Índia e 15% —35% no Sri Lanka e Bangladesh, respectivamente. Entre os grupos regionais, especialmente entre os grupos austroasíacos, atinge um pico de cerca de 75%. O haplogrupo O2a (O-K18) é freqüentemente encontrado entre os asiáticos do sudeste e leste, especialmente entre os arrozeiros austro-asiáticos. É sugerido que O2a (O-K18) chegou do Sudeste Asiático à Índia após o último máximo glacial, por volta de 10.000 AC.
Reconstruindo a história da população do sul da Ásia
The Indian Genome Variation Consortium (2008) , divide a população do Sul da Ásia em quatro grupos etnolinguísticos (não genéticos): indo-europeu , dravidiano , tibeto-birmanês e austro-asiático . Os estudos de antropologia molecular usam três tipos diferentes de marcadores: variação do DNA mitocondrial (mtDNA) que é herdada da mãe e altamente polimórfica, variação do cromossomo Y que envolve a transmissão uniparental ao longo das linhagens masculinas e variação do DNA autossômico.
variação do mtDNA
A maioria dos estudos baseados na variação do mtDNA relatou a unidade genética das populações do sul da Ásia em vários idiomas, castas e grupos tribais. É provável que o haplogrupo M tenha sido trazido da África Oriental para a Ásia ao longo da rota sul pela primeira onda de migração de 78.000 anos atrás.
De acordo com Kivisild et al. (1999), "Pequenas sobreposições com linhagens descritas em outras populações da Eurásia demonstram claramente que as imigrações recentes tiveram muito pouco impacto na estrutura inata do pool genético materno dos asiáticos do sul. Apesar das variações encontradas na Índia, essas populações derivam de uma forma limitada número de linhagens fundadoras. Essas linhagens foram provavelmente introduzidas no Sul da Ásia durante o Paleolítico Médio, antes do povoamento da Europa há 48.000 anos e talvez do Velho Mundo em geral. " Basu et al. (2003) também enfatiza a unidade subjacente das linhagens femininas na Índia.
Variação do cromossomo Y
As conclusões baseadas na variação do cromossomo Y têm sido mais variadas do que aquelas baseadas na variação do mtDNA. Enquanto Kivisild et al. (2003) propõe uma herança genética antiga e compartilhada de linhagens masculinas no sul da Ásia, Bamshad et al. (2001) sugere uma afinidade entre as linhagens masculinas do sul da Ásia e os modernos eurasianos ocidentais proporcional à classe de casta superior e coloca as populações de casta superior dos estados do sul da Índia mais perto dos europeus orientais.
Basu et al. (2003) conclui que as populações tribais austro-asiáticas entraram na Índia primeiro pelo corredor noroeste e muito mais tarde algumas delas pelo corredor nordeste. Enquanto Kumar et al. (2007) analisaram 25 tribos austro-asiáticas do sul da Ásia e encontraram uma forte ligação genética paterna entre os grupos sublinguísticos das populações austro-asiáticas do sul da Ásia. Mukherjee et al. (2001) coloca paquistaneses e indianos do norte entre as populações da Ásia Ocidental e da Ásia Central, enquanto Cordaux et al. (2004) argumenta que as populações de castas indianas estão mais próximas das populações da Ásia Central. Sahoo et al. (2006) e Sengupta et al. (2006) sugerem que as populações de castas indianas não foram sujeitas a quaisquer misturas recentes. Sanghamitra Sahoo conclui seu estudo com:
Não é necessário, com base nas evidências atuais, olhar além do Sul da Ásia para as origens da herança paterna da maioria dos índios na época do início da agricultura sedentária. O conceito perene de pessoas, língua e agricultura chegando juntas à Índia através do corredor noroeste não resiste a um exame minucioso. Reclamações recentes para uma ligação dos haplogrupos J2, L, R1a e R2 com uma origem contemporânea para a maioria das linhagens paternas das castas indianas de fora do Sul da Ásia são rejeitadas, embora nossos resultados apóiem uma origem local dos haplogrupos F * e H. Dos outros, apenas J2 indica uma contribuição externa recente inequívoca, da Ásia Ocidental em vez da Ásia Central. As distribuições atuais de frequências de haplogrupos são, com exceção das linhagens, predominantemente conduzidas por determinantes geográficos, ao invés de culturais. Ironicamente, é no nordeste da Índia, entre os grupos de TB, que há evidências claras de difusão dêmica em grande escala rastreável por genes, cultura e idioma, mas aparentemente não pela agricultura.
A análise do vizinho mais próximo feita por Mondal et al. em 2017 concluiu que as linhagens Y indianas estão próximas às populações do sul da Europa e ao tempo de divergência entre as duas migrações estepárias anteriores:
Esses resultados sugerem que a ancestralidade relacionada à Europa nas populações indígenas pode ser muito mais antiga e mais complexa do que o previsto, e pode se originar da primeira onda de agricultores ou até mesmo antes
- Mondal et al. 2017
Variação autossômica de DNA
AASI-ANI-ASI
Os resultados dos estudos baseados na variação autossômica do DNA também foram variados. Em um grande estudo (2009) usando mais de 500.000 marcadores autossômicos bialélicos, Reich formulou a hipótese de que a população moderna do sul da Ásia era o resultado da mistura entre duas populações ancestrais geneticamente divergentes datando da era pós-Holoceno. Essas duas populações antigas "reconstruídas" ele denominou "Índios Ancestrais do Sul" (ASI) e "Índios Ancestrais do Norte" (ANI). De acordo com Reich: "A ancestralidade ANI é significativamente maior em falantes indo-europeus do que em falantes dravidianos, sugerindo que o ASI ancestral pode ter falado uma língua dravidiana antes de se misturar com o ANI." Embora o ANI seja geneticamente próximo aos do Oriente Médio, centro-asiáticos e europeus, o ASI não está intimamente relacionado a grupos fora do subcontinente. Como nenhum DNA antigo "ASI" está disponível, os indígenas Andamanese Onge são usados como um proxy (imperfeito) do ASI (de acordo com Reich et al., Os Andamaneses, embora distintos deles, são a população viva mais próxima do ASI). De acordo com Reich et al., Tanto a ancestralidade ANI quanto a ASI são encontradas em todo o subcontinente (no norte e no sul da Índia) em proporções variadas, e que "a ancestralidade ANI varia de 39-71% na Índia, e é maior no tradicionalmente superior casta e falantes indo-europeus. "
Moorjani et al. 2013 afirmam que os ASI, embora não sejam intimamente relacionados a qualquer grupo vivo, são "aparentados (distantemente) com os indígenas das ilhas de Andaman". Moorjani et al. no entanto, sugere um possível fluxo gênico para o Andamanês de uma população relacionada ao ASI, causando a relação modelada. O estudo concluiu que "quase todos os grupos que falam línguas indo-europeias ou dravidianas estão ao longo de um gradiente de parentesco variável com os eurasianos ocidentais no PCA (referido como" cline indiano ")".
Um estudo de Chaubey de 2013 usando o polimorfismo de nucleotídeo único (SNP) mostra que o genoma do povo andamanês (Onge) está mais próximo do genoma de outros grupos Negrito Oceânicos do que dos sul-asiáticos.
De acordo com Basu et al. 2016, uma análise mais aprofundada revelou que a estrutura genômica das populações indígenas do continente é melhor explicada por contribuições de quatro componentes ancestrais. Além do ANI e ASI, Basu et. al (2016) identificou dois componentes ancestrais do Leste Asiático na Índia continental que são importantes para os tribais de língua austro-asiática e os falantes de tibeto-birmanês, que eles designaram como AAA (para "Ancestral Austro-Asiático") e ATB (para " Tibeto-birmanês ancestral "), respectivamente. O estudo também infere que as populações do arquipélago das Ilhas Andaman formam uma ancestralidade distinta, que "foi considerada coancestral às populações oceânicas ", mas mais distante dos sul-asiáticos.
O cline de mistura entre as linhagens ANI e ASI é datado do período de c. 4,2-1,9 kya por Moorjani et al. (2013), correspondente à Idade do Bronze Indiana, e associada pelos autores ao processo de desurbanização da Civilização do Vale do Indo e à mudança da população para o sistema Gangético na incipiente Idade do Ferro Indiana. Basu et al. (2003) sugere que "falantes dravidianos estavam possivelmente espalhados por toda a Índia antes da chegada dos nômades de língua indo-europeia" e que "a formação de populações por fissão que resultou em efeitos fundadores e de deriva deixaram suas marcas nas estruturas genéticas contemporâneas populações ". O geneticista PP Majumder (2010) argumentou recentemente que os achados de Reich et al. (2009) estão em notável concordância com pesquisas anteriores usando mtDNA e Y-DNA:
Supõe-se que as populações da Ásia Central tenham contribuído significativamente para o pool genético indiano, particularmente para o pool genético do norte da Índia, e os migrantes supostamente se mudaram para a Índia através do que hoje é o Afeganistão e o Paquistão. Usando dados de variação de DNA mitocondrial coletados de vários estudos, mostramos que as populações da Ásia Central e do Paquistão apresentam o menor coeficiente de diferenciação genética com as populações do norte da Índia, uma maior diferenciação com as populações do sul da Índia e a maior com as populações do nordeste da Índia. . As populações do norte da Índia são geneticamente mais próximas dos centro-asiáticos do que as populações de outras regiões geográficas da Índia .... Consistente com as descobertas acima, um estudo recente usando mais de 500.000 marcadores autossômicos bialélicos encontrou um gradiente norte a sul de proximidade genética das populações indianas aos eurasianos ocidentais. Esta característica está provavelmente relacionada às proporções de ancestralidade derivada do pool de genes da Eurásia ocidental, que, como este estudo mostrou, é maior em populações que habitam o norte da Índia do que aquelas que habitam o sul da Índia.
Chaubey et al. 2015 detectou um componente ancestral distinto do Leste Asiático, principalmente restrito a populações específicas no sopé do Himalaia e na parte nordeste da Índia. A frequência mais alta do componente é observada entre os grupos de língua tibeto-birmanesa do nordeste da Índia e também foi detectada em populações de Andamanese em 32%, com presença substancial também entre falantes austro-asiáticos. Encontra-se amplamente ausente em falantes indo-europeus e dravidianos, exceto em alguns grupos étnicos específicos que vivem no sopé do Himalaia e no centro-sul da Índia. Os pesquisadores, entretanto, sugeriram que a ancestralidade do Leste Asiático (representada pelo Han) medida nos grupos andamaneses estudados pode realmente refletir a captura da afinidade dos Andamaneses com os Negritos da Malásia e Melanésios (ao invés da verdadeira mistura do Leste Asiático), como um estudo anterior por Chaubey et al. sugeriu "uma ancestralidade comum profunda" entre Andamanese, Melanésios e outros grupos Negrito, e uma afinidade entre Negritos do Sudeste Asiático e Melanésios (assim como os Andamaneses) com os Asiáticos Orientais.
Lazaridis et al. (2016) observa "O impacto demográfico das populações relacionadas às estepes no Sul da Ásia foi substancial, já que os Mala , uma população dalit do sul da Índia com ancestralidade ancestral mínima do norte da Índia (ANI), ainda tem ancestralidade Yamnaya de ~ 18% das estepes , enquanto os Kalash do Paquistão, infere-se que tem cerca de 50% de ancestralidade Yamnaya estepe, uma das maiores percentagens a seguir aos europeus orientais, com 51%. " O estudo de Lazaridis et al. De 2016 estimou (6,5–50,2%) a mistura relacionada às estepes em sul-asiáticos. Lazaridis et al. observa ainda que "Uma direção útil para pesquisas futuras é uma amostragem mais abrangente de DNA antigo de populações de estepe, bem como populações da Ásia Central (leste do Irã e sul da estepe), o que pode revelar fontes mais próximas do ANI do que os considerados aqui, e do Sul da Ásia para determinar a trajetória da mudança populacional na área diretamente.
Pathak et al. 2018 concluiu que os falantes indo-europeus das planícies gangéticas e os falantes dravidianos têm ancestralidade Yamnaya da Idade do Bronze Médio (Steppe_EMBA) significativa, mas nenhuma ancestralidade das Estepes da Idade do Bronze Médio-Final (Steppe_MLBA). Por outro lado, as populações do "Noroeste da Índia e Paquistão" (PNWI) mostraram ancestralidade Steppe_MLBA significativa junto com ancestralidade Yamnaya (Steppe_EMBA). O estudo também observou que as amostras antigas do sul da Ásia tinham Steppe_MLBA significativamente maior do que Steppe_EMBA (ou Yamnaya). O estudo também sugeriu que os Rors poderiam ser usados como um proxy para o ANI.
David Reich em seu livro de 2018 Quem Somos e Como Chegamos Aqui afirma que as análises de 2016 descobriram que o ASI tem quantidades significativas de um componente de ancestralidade derivado de agricultores iranianos (cerca de 25% de sua ancestralidade), com os restantes 75% de seus ancestralidade derivada de caçadores-coletores nativos do sul da Ásia. Ele acrescenta que a ASI era improvável que os caçadores-coletores locais do Sul da Ásia como estabelecido anteriormente, mas uma população responsável por espalhar a agricultura por todo o Sul da Ásia. No caso do ANI, a ancestralidade dos agricultores iranianos é de 50%, com o restante sendo de grupos de estepes relacionados ao Yamnaya.
Narasimhan et al. (2018) , da mesma forma, concluem que ANI e ASI foram formados no segundo milênio AEC. Eles foram precedidos por uma mistura de AASI (ancestrais ancestrais do sul da Índia, isto é, caçadores-coletores compartilhando uma raiz distante com os andamaneses, aborígenes australianos e asiáticos); e os agricultores iranianos que chegaram à Índia ca. 4700-3000 AC, e "deve ter alcançado o Vale do Indo por volta do 4º milênio AC". De acordo com Narasimhan et al., Esta população mista, que provavelmente era nativa da Civilização do Vale do Indo, "contribuiu em grandes proporções tanto para o ANI quanto para o ASI", que tomou forma durante o segundo milênio aC. ANI formou-se de uma mistura de " grupos relacionados à Periferia Indus " e migrantes da estepe, enquanto ASI foi formado de " grupos relacionados à Periferia Indus " que se mudaram para o sul e se misturaram ainda mais com caçadores-coletores locais. A ancestralidade da população ASI é sugerida como tendo uma média de 73% do AASI e 27% dos fazendeiros iranianos. Narasimhan et al. observe que as amostras do grupo da periferia do Indo são sempre misturas das mesmas duas fontes proximais de AASI e ancestralidade relacionada à agricultura iraniana; com "um dos indivíduos da Periferia Indus tendo ~ 42% de ancestralidade AASI e os outros dois indivíduos tendo ~ 14-18% de ancestralidade AASI" (com o restante de sua ancestralidade sendo da população ligada a agricultores iranianos). Os autores propõem que os caçadores-coletores indígenas AASI representam um ramo divergente que se separou na mesma época em que os ancestrais aborígenes do Leste Asiático, Onge (Andamanês) e australianos se separaram. Ele inferiu que "essencialmente toda a ancestralidade dos atuais asiáticos do leste e do sul (antes da mistura relacionada com a Eurásia Ocidental nos asiáticos do sul) deriva de uma única propagação para o leste, que deu origem em um curto espaço de tempo às linhagens que levaram ao AASI , Asiáticos do leste, Onge e australianos. "
Um estudo genético de Yelmen et al. (2019) no entanto, mostra que as populações modernas do sul da Ásia são geralmente mais próximas umas das outras e bastante distintas das populações fora do sul da Ásia. Eles concluíram que os modernos sul-asiáticos são basicamente uma mistura de um componente nativo do sul da Ásia (AASI) e um componente ocidental-eurasiano que chegou mais tarde (derivado da Ásia Ocidental e das estepes ocidentais). Os autores também revelaram que o componente indígena do sul da Ásia (AASI) é bastante distinto dos andamaneses ou asiáticos do leste, e que os andamaneses são, portanto, um substituto imperfeito. Eles propõem que o povo Paniya tribal do sul da Índia (um grupo de ascendência predominantemente ASI) serviria como um substituto melhor do que os Andamaneses (Onge) para o componente "indígena do Sul da Ásia" nos sul-asiáticos modernos, já que os Paniya são derivados diretamente do nativos do sul da Ásia, enquanto os Onge podem ter recebido fluxo genético de outros grupos. Eles descobriram que a população indígena do sul da Ásia estava isolada de outras populações desde pelo menos 45.000 aC.
Dois estudos genéticos (Shinde et al. 2019 e Narasimhan et al. 2019) analisando vestígios da civilização do Vale do Indo (de partes da Idade do Bronze no noroeste da Índia e Paquistão Oriental), descobriram que eles tinham uma mistura de ancestrais, ambos do sul nativo Caçadores-coletores asiáticos que compartilham uma raiz distante com os andamaneses e de um grupo relacionado aos fazendeiros iranianos. As amostras analisadas por Shinde derivaram cerca de 50-98% de seu genoma de povos relacionados ao Irã e de 2-50% de caçadores-coletores nativos do sul da Ásia. As amostras analisadas por Narasimhan et al. tinha 45–82% de ancestralidade ligada a fazendeiros iranianos e 11–50% de origem caçadora-coletora do sul da Ásia. As amostras analisadas de ambos os estudos têm pouco ou nenhum componente da " ancestralidade das estepes " associado às migrações indo-europeias posteriores para a Índia. Os autores descobriram que as respectivas quantidades dessas linhagens variaram significativamente entre os indivíduos e concluíram que mais amostras são necessárias para obter uma imagem completa da história da população indiana.
Distância genética entre grupos de castas e tribos
Estudos de Watkins et al. (2005) e Kivisild et al. (2003) com base em marcadores autossômicos concluem que a casta indiana e as populações tribais têm uma ancestralidade comum. Reddy et al. (2005) encontraram distribuições de frequência de alelos bastante uniformes em grupos de castas do sul de Andhra Pradesh , mas distância genética significativamente maior entre grupos de castas e tribos, indicando isolamento genético das tribos e castas.
Viswanathan et al. (2004) em um estudo sobre a estrutura genética e afinidades entre populações tribais do sul da Índia conclui: " A diferenciação genética era alta e as distâncias genéticas não estavam significativamente correlacionadas com as distâncias geográficas. A deriva genética, portanto, provavelmente desempenhou um papel significativo na formação dos padrões de variação genética observada em populações tribais da Índia do Sul. Caso contrário, as análises das relações populacionais mostraram que todas as populações da Índia e do Sul da Ásia ainda são semelhantes entre si, independentemente das características fenotípicas, e não mostram nenhuma afinidade particular com os africanos. Concluímos que as semelhanças fenotípicas de alguns grupos de índios para africanos não refletem uma relação estreita entre esses grupos, mas são melhor explicados pela convergência . "
Um estudo de 2011 publicado no American Journal of Human Genetics indica que os componentes ancestrais dos índios são o resultado de uma história demográfica mais complexa do que se pensava anteriormente. De acordo com os pesquisadores, o Sul da Ásia abriga dois componentes ancestrais principais, um dos quais se espalha com frequência e diversidade genética comparáveis em populações da Ásia Central, Ásia Ocidental e Europa; o outro componente é mais restrito ao sul da Ásia. No entanto, se alguém fosse descartar a possibilidade de uma migração indo-ariana em grande escala, essas descobertas sugerem que as afinidades genéticas de ambos os componentes ancestrais indianos são o resultado de múltiplos fluxos gênicos ao longo de milhares de anos.
A modelagem das diversidades de haplótipos observados sugere que ambos os componentes da ancestralidade indiana são mais antigos do que a suposta invasão indo-ariana de 3.500 YBP. Consistente com os resultados das distâncias genéticas entre pares entre as regiões do mundo, os indianos compartilham mais sinais de ancestralidade com o oeste do que com os eurasianos do leste.
Narashimhan et al 2019 descobriram que os tribais Munda de língua austro-asiática não podiam ser modelados simplesmente como uma mistura de ancestrais ASI, AASI e ANI, ao contrário de outros sul-asiáticos, mas exigiam um componente de ancestralidade adicional do Sudeste Asiático. Eles foram modelados como uma mistura de 88% AASI e 12% relacionado ao Leste Asiático com Nicobarese , portanto, o perfil de ancestralidade dos Mundas fornece uma linha independente de ancestralidade do Sudeste Asiático por volta do terceiro milênio aC. Lipson et al. 2018 encontrou resultados de mistura semelhantes em relação aos tribais Munda, afirmando que "obtivemos um bom ajuste com três componentes de ancestralidade: um eurasiano ocidental, um eurasiano oriental profundo (interpretado como uma linhagem indígena do sul da Ásia) e um do clado austroasiático" . Lipson et al. 2018 descobriu ainda que o revestimento de origem austro - asiática (proporção 35%) em tribos Munda foi inferido como sendo o mais próximo de Mlabri . Singh e cols. 2020, de forma semelhante, descobriram que falantes austro-asiáticos no sul da Ásia caem fora do cline do sul da Ásia devido à sua afinidade genética do sudeste asiático.
Veja também
- Arqueogenética
- Grupos étnicos do Sul da Ásia
- Lista de regiões etnolinguísticas do Sul da Ásia
- Povoamento da Índia
- Haplogrupos Y-DNA em populações do Sul da Ásia
- Estudos genéticos no povo Gujarati
- História genética da Europa
- História genética do Oriente Médio
Notas
Referências
Leitura adicional
- Allikas A, Ord D, Kurg R, Kivi S, Ustav M (junho de 2001). "Papéis da região de dobradiça e do domínio de ligação ao DNA da proteína E2 do papilomavírus bovino tipo 1 na iniciação da replicação do DNA". Pesquisa de vírus . 75 (2): 95–106. doi : 10.1016 / S0168-1702 (01) 00219-2 . PMID 11325464 .
- Behar DM, Garrigan D, Kaplan ME, Mobasher Z, Rosengarten D, Karafet TM, et al. (Março de 2004). "Padrões contrastantes de variação do cromossomo Y em populações de judeus Ashkenazi e não-judeus europeus". Genética Humana . 114 (4): 354–65. doi : 10.1007 / s00439-003-1073-7 . PMID 14740294 . S2CID 10310338 .
- Bhattacharyya NP, Basu P, Das M, Pramanik S, Banerjee R, Roy B, et al. (Agosto de 1999). "Fluxo insignificante de genes masculinos através das fronteiras étnicas na Índia, revelado pela análise de polimorfismos de DNA do cromossomo Y" . Genome Research . 9 (8): 711–9. doi : 10.1101 / gr.9.8.711 (inativo em 6 de maio de 2021). PMID 10447506 .Manutenção do CS1: DOI inativo em maio de 2021 ( link )
- Cann RL (março de 2001). "Pistas genéticas para a dispersão em populações humanas: reconstituindo o passado do presente". Ciência . 291 (5509): 1742–8. Bibcode : 2001Sci ... 291.1742C . doi : 10.1126 / science.1058948 . PMID 11249820 . S2CID 19367408 .
- Cinnioğlu C, King R, Kivisild T, Kalfoğlu E, Atasoy S, Cavalleri GL, et al. (Janeiro de 2004). "Escavando estratos de haplótipos do cromossomo Y na Anatólia". Genética Humana . 114 (2): 127–48. doi : 10.1007 / s00439-003-1031-4 . PMID 14586639 . S2CID 10763736 .
- Das B, Chauhan PS, Seshadri M (outubro de 2004). "Compartilhamento mínimo de haplótipos STR do cromossomo Y entre cinco grupos populacionais endógamos do oeste e sudoeste da Índia". Biologia Humana . 76 (5): 743–63. doi : 10.1353 / hub.2005.0003 . PMID 15757245 . S2CID 12835244 .
- Hemphill BE, Christensen AF (3 de novembro de 1994). A civilização de Oxus como um elo entre o Oriente e o Ocidente: uma análise não métrica das afinidades biológicas do Bactrain da Idade do Bronze . Madison, Wisconsin. p. 13 (artigo lido na Conferência da Ásia do Sul)
- Jobling MA, Tyler-Smith C (agosto de 2003). "O cromossomo Y humano: um marcador evolutivo chega à maioridade". Nature Reviews. Genética . 4 (8): 598–612. doi : 10.1038 / nrg1124 . PMID 12897772 . S2CID 13508130 .
- Kivisild T , Rootsi S, Metspalu M, Mastana S, Kaldma K, Parik J, et al. (Fevereiro de 2003). "A herança genética dos primeiros colonos persiste tanto nas populações indígenas tribais como nas de castas" . American Journal of Human Genetics . 72 (2): 313–32. doi : 10.1086 / 346068 . PMC 379225 . PMID 12536373 .
- Metspalu M, Kivisild T, Metspalu E, Parik J, Hudjashov G, Kaldma K, et al. (Agosto de 2004). "A maioria das fronteiras do mtDNA existentes no sul e sudoeste da Ásia foram provavelmente moldadas durante a colonização inicial da Eurásia por humanos anatomicamente modernos" . BMC Genetics . 5 : 26. doi : 10.1186 / 1471-2156-5-26 . PMC 516768 . PMID 15339343 .
- Patowary A, Purkanti R, Singh M, Chauhan RK, Bhartiya D, Dwivedi OP, et al. (Julho de 2012). "Análise sistemática e anotação funcional de variações no genoma de um indivíduo indiano". Mutação Humana . 33 (7): 1133–40. doi : 10.1002 / humu.22091 . PMID 22461382 . S2CID 11466942 .
- Rootsi S, Magri C, Kivisild T, Benuzzi G, Help H, Bermisheva M, et al. (Julho de 2004). "A filogeografia do haplogrupo I do cromossomo Y revela domínios distintos do fluxo gênico pré-histórico na Europa" . American Journal of Human Genetics . 75 (1): 128–37. doi : 10.1086 / 422196 . PMC 1181996 . PMID 15162323 .
- Qamar R, Ayub Q, Mohyuddin A, Helgason A, Mazhar K, Mansoor A, et al. (Maio de 2002). "Variação do DNA cromossômico Y no Paquistão" . American Journal of Human Genetics . 70 (5): 1107–24. doi : 10.1086 / 339929 . PMC 447589 . PMID 11898125 .
- Semino O, Magri C, Benuzzi G, Lin AA, Al-Zahery N, Battaglia V, et al. (Maio de 2004). "Origem, difusão e diferenciação dos haplogrupos E e J do cromossomo Y: inferências sobre a neolitização da Europa e eventos migratórios posteriores na área do Mediterrâneo" . American Journal of Human Genetics . 74 (5): 1023–34. doi : 10.1086 / 386295 . PMC 1181965 . PMID 15069642 .
Fontes
- Indian Genome Variation Consortium (abril de 2008). "Paisagem genética do povo da Índia: uma tela para a exploração de genes de doenças". Journal of Genetics . 87 (1): 3–20. doi : 10.1007 / s12041-008-0002-x . PMID 18560169 . S2CID 21473349 .
- Endicott P, Metspalu M, Kivisild T (2007). "Evidência genética em dispersões humanas modernas no Sul da Ásia: cromose Y e perspectivas de DNA mitocondrial" . Em Michael D. Petraglia, Bridget Allchin (eds.). A evolução e a história das populações humanas no sul da Ásia . Springer. pp. 201–228. ISBN 978-1-4020-5561-4.
- Hemphill B, Lukacs J, Kennedy K (1991). "Adaptações biológicas e afinidades dos Harappans da Idade do Bronze". Em Meadow RH (ed.). Escavações Harappa 1986-1990: uma abordagem multidisciplinar para o urbanismo do terceiro milênio . pp. 137–82. ISBN 978-0-9629110-1-9.
- Kennedy KA (1984). "Uma reavaliação das teorias das origens raciais do povo da civilização do Vale do Indo a partir de dados antropológicos recentes". Em Kennedy KA, Possehl GL (eds.). Estudos em Arqueologia e Paleoantropologia do Sul da Ásia . Atlantic Highlands, NJ : Humanities Press. pp. 99–107.
- Kennedy KA (1995). "Os arianos foram identificados no registro esquelético pré-histórico do sul da Ásia?" . Em George Erdosy (ed.). Os indo-arianos do antigo sul da Ásia . Walter de Gruyter. pp. 49–54. ISBN 978-3-11-014447-5.
- Kivisild T (2000b). As origens das populações do sul e oeste da Eurásia: um estudo de mtDNA (PDF) . Universidade de Tartu, Estônia. (PhD)
- Mascarenhas DD, Raina A, Aston CE, Sanghera DK (2015). "Reconstrução genética e cultural da migração de uma linhagem antiga" . BioMed Research International . 2015 : 651415. doi : 10.1155 / 2015/651415 . PMC 4605215 . PMID 26491681 .
- Narasimhan VM, Anthony D, Mallory J, Reich D (2018). A formação genômica da Ásia do Sul e Central . bioRxiv 10.1101 / 292581 . doi : 10.1101 / 292581 . hdl : 21.11116 / 0000-0001-E7B3-0 . S2CID 89658279 .
- Oppenheimer S (2003). The Real Eve: Modern Man's Journey out of Africa . Nova York: Carroll and Graf Publishers. ISBN 978-0-7867-1192-5.
- Pamjav (dezembro de 2012). "Breve comunicação: Novos marcadores binários do cromossomo Y melhoram a resolução filogenética no haplogrupo R1a1". American Journal of Physical Anthropology . 149 (4): 611–615. doi : 10.1002 / ajpa.22167 . PMID 23115110 . S2CID 4820868 .
- Renfrew C, Boyle K, eds. (2000a). Um ancestral indiano: uma chave para a compreensão da diversidade humana na Europa e além (PDF) . ISBN 978-1-902937-08-3.
- Silva M, Oliveira M, Vieira D, Brandão A, Rito T, Pereira JB, et al. (Março de 2017). "Uma cronologia genética para o subcontinente indiano aponta para dispersões fortemente enviesadas pelo sexo" . BMC Evolutionary Biology . 17 (1): 88. doi : 10.1186 / s12862-017-0936-9 . PMC 5364613 . PMID 28335724 .
- Underhill PA (2003). "Inferindo história humana: pistas dos haplótipos do cromossomo Y". Simpósio Cold Spring Harbor sobre Biologia Quantitativa . 68 : 487–93. doi : 10.1101 / sqb.2003.68.487 . PMID 15338652 . S2CID 15527457 .
- Underhill PA, Poznik GD, Rootsi S, Järve M, Lin AA, Wang J, et al. (Janeiro de 2015). "A estrutura filogenética e geográfica do haplogrupo R1a do cromossomo Y" . European Journal of Human Genetics . 23 (1): 124–31. doi : 10.1038 / ejhg.2014.50 . PMC 4266736 . PMID 24667786 .
- Wells S (2003). The Journey of Man: A Genetic Odyssey . Princeton University Press. ISBN 9780691115320.
links externos
- Introdução aos Haplogrupos e Haplótipos , Mark A. Jobling, University of Leicester. [1]
- Journey of Man: Peopling of the World , Fundação Bradshaw, em associação com Stephen Oppenheimer .
- Instituto Indiano de Banco de Dados de Variação do Genoma de Genômica e Biologia Integrativa
- Lista de frequência R2
- Maldivas