Modelos moleculares de DNA - Molecular models of DNA
Modelos moleculares de estruturas de DNA são representações da geometria molecular e topologia de moléculas de ácido desoxirribonucléico ( DNA ) usando um de vários meios, com o objetivo de simplificar e apresentar as propriedades essenciais, físicas e químicas, de estruturas moleculares de DNA in vivo ou em vitro . Essas representações incluem esferas compactadas ( modelos CPK ) feitas de plástico, fios de metal para modelos esqueléticos , cálculos gráficos e animações por computadores, renderização artística. Modelos moleculares de computador também permitem animações e simulações de dinâmica molecular que são muito importantes para entender como o DNA funciona in vivo .
Os modelos moleculares de DNA mais avançados e baseados em computador envolvem simulações de dinâmica molecular e cálculos de mecânica quântica de vibro-rotações, orbitais moleculares deslocalizados (MOs), momentos de dipolo elétrico , ligações de hidrogênio e assim por diante. A modelagem da dinâmica molecular do DNA envolve a simulação da geometria molecular do ácido desoxirribonucléico ( DNA ) e mudanças na topologia com o tempo como resultado de interações intra e inter-moleculares do DNA. Enquanto os modelos moleculares de moléculas de DNA, como esferas compactadas (modelos CPK) feitos de plástico ou fios de metal para modelos esqueléticos, são representações úteis de estruturas estáticas de DNA, sua utilidade é muito limitada para representar a dinâmica complexa do DNA. A modelagem molecular por computador permite animações e simulações de dinâmica molecular que são muito importantes para entender como o DNA funciona in vivo .
História
Desde os estágios iniciais dos estudos estruturais de DNA por difração de raios-X e meios bioquímicos, modelos moleculares como o modelo de dupla hélice de ácido nucleico de Watson-Crick foram empregados com sucesso para resolver o 'quebra-cabeça' da estrutura do DNA e também descobrir como o a última relaciona-se com suas funções-chave nas células vivas. Os primeiros padrões de difração de raios-X de alta qualidade do A-DNA foram relatados por Rosalind Franklin e Raymond Gosling em 1953. Rosalind Franklin fez a observação crítica de que o DNA existe em duas formas distintas, A e B, e produziu as imagens mais nítidas de ambos através Técnica de difração de raios-X. Os primeiros cálculos da transformada de Fourier de uma hélice atômica foram relatados um ano antes por Cochran, Crick e Vand, e foram seguidos em 1953 pelo cálculo da transformada de Fourier de uma bobina enrolada por Crick.
A informação estrutural é gerada a partir de estudos de difração de raios-X de fibras de DNA orientadas com a ajuda de modelos moleculares de DNA que são combinados com análises cristalográficas e matemáticas dos padrões de raios-X.
Os primeiros relatos de um modelo molecular em dupla hélice do DNA estrutura-B foram feitas por James Watson e Francis Crick em 1953. Nesse mesmo ano, Maurice F. Wilkins, A. Stokes e HR Wilson, relatou os primeiros padrões de raios-X de em vivo B-DNA em cabeças de esperma de salmão parcialmente orientadas.
O desenvolvimento do primeiro modelo molecular de dupla hélice correto de DNA por Crick e Watson pode não ter sido possível sem a evidência bioquímica para o pareamento de bases de nucleotídeos ([A --- T]; [C --- G]), ou Regras de Chargaff . Embora esses estudos iniciais das estruturas do DNA com a ajuda de modelos moleculares fossem essencialmente estáticos, suas consequências para explicar as funções in vivo do DNA foram significativas nas áreas de biossíntese de proteínas e na quase universalidade do código genético. Os estudos de transformação epigenética do DNA in vivo foram, entretanto, muito mais lentos para se desenvolver, apesar de sua importância para a embriologia, morfogênese e pesquisa do câncer. Essa dinâmica química e reações bioquímicas do DNA são muito mais complexas do que a dinâmica molecular das interações físicas do DNA com água, íons e proteínas / enzimas em células vivas.
Importância
Um velho problema dinâmico é como a "autorreplicação" do DNA ocorre em células vivas, o que deve envolver o desenrolamento transitório de fibras superenroladas de DNA. Embora o DNA consista em moléculas de biopolímero alongadas relativamente rígidas e muito grandes chamadas de fibras ou cadeias (que são feitas de unidades de nucleotídeo repetidas de quatro tipos básicos, ligadas a grupos de desoxirribose e fosfato), sua estrutura molecular in vivo sofre alterações de configuração dinâmica que envolvem anexação dinâmica moléculas de água e íons. Superenrolamento, empacotamento com histonas em estruturas cromossômicas e outros aspectos supramoleculares também envolvem a topologia do DNA in vivo, que é ainda mais complexa do que a geometria molecular do DNA, tornando a modelagem molecular do DNA um problema especialmente desafiador para biólogos moleculares e biotecnologistas. Como outras moléculas grandes e biopolímeros, o DNA frequentemente existe em múltiplas geometrias estáveis (ou seja, exibe isomerismo conformacional ) e estados quânticos configuracionais que estão próximos uns dos outros em energia na superfície de energia potencial da molécula de DNA.
Essas geometrias moleculares variáveis também podem ser calculadas, pelo menos em princípio, empregando métodos de química quântica ab initio que podem atingir alta precisão para moléculas pequenas, embora alegações de que uma precisão aceitável também possa ser alcançada para polinuclelotídeos e conformações de DNA tenham sido feitas recentemente em a base de dados espectrais de dicroísmo circular vibracional (VCD). Tais geometrias quânticas definem uma importante classe de modelos moleculares ab initio de DNA cuja exploração mal começou, especialmente relacionada aos resultados obtidos por VCD em soluções. Comparações mais detalhadas com tais cálculos quânticos ab initio são, em princípio, obtidos por meio de espectroscopia 2D-FT NMR e estudos de relaxamento de soluções de polinucleotídeos ou DNA especificamente marcado, como por exemplo com marcadores de deutério.
Em uma interessante reviravolta de papéis, a molécula de DNA foi proposta para ser usada na computação quântica via DNA. Ambas as nanoestruturas de DNA e biochips de computação de DNA foram construídos.
Conceitos fundamentais
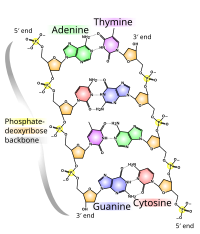
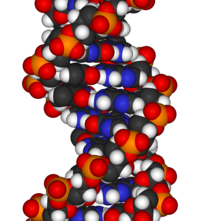
A estrutura química do DNA é insuficiente para entender a complexidade das estruturas 3D do DNA. Em contraste, os modelos moleculares animados permitem explorar visualmente a estrutura tridimensional (3D) do DNA. O modelo de DNA mostrado (extrema direita) é um modelo de preenchimento de espaço, ou CPK , da dupla hélice do DNA. Modelos moleculares animados, como o tipo wire, ou esquelético, mostrado no início deste artigo, permitem explorar visualmente a estrutura tridimensional (3D) do DNA. Outro tipo de modelo de DNA é o modelo de preenchimento de espaço, ou CPK.
A dinâmica da ligação de hidrogênio e a troca de prótons são muito diferentes em muitas ordens de magnitude entre os dois sistemas de DNA totalmente hidratado e moléculas de água no gelo. Assim, a dinâmica do DNA é complexa, envolvendo escalas de tempo de nanossegundos e várias dezenas de picossegundos, enquanto a do gelo líquido está na escala de picossegundos e a da troca de prótons no gelo está na escala de milissegundos. As taxas de troca de prótons no DNA e proteínas anexadas podem variar de picossegundo a nanossegundo, minutos ou anos, dependendo da localização exata dos prótons trocados nos grandes biopolímeros.
Um oscilador harmônico simples 'vibração' é apenas uma representação dinâmica supersimplificada das vibrações longitudinais das hélices entrelaçadas do DNA que foram consideradas anarmônicas ao invés de harmônicas como freqüentemente assumido em simulações dinâmicas quânticas de DNA.
Estrutura do DNA
A estrutura do DNA mostra uma variedade de formas, tanto de fita dupla quanto de fita simples. As propriedades mecânicas do DNA, que estão diretamente relacionadas à sua estrutura, são um problema significativo para as células . Todo processo que liga ou lê o DNA é capaz de usar ou modificar as propriedades mecânicas do DNA para fins de reconhecimento, empacotamento e modificação. O comprimento extremo (um cromossomo pode conter uma fita de DNA de 10 cm de comprimento), a rigidez relativa e a estrutura helicoidal do DNA levaram à evolução das histonas e de enzimas como topoisomerases e helicases para gerenciar o DNA de uma célula. As propriedades do DNA estão intimamente relacionadas à sua estrutura e sequência molecular, particularmente a fraqueza das ligações de hidrogênio e interações eletrônicas que mantêm as fitas de DNA juntas em comparação com a força das ligações dentro de cada fita.
Os métodos experimentais que podem medir diretamente as propriedades mecânicas do DNA são relativamente novos, e a visualização de alta resolução em solução costuma ser difícil. No entanto, os cientistas descobriram uma grande quantidade de dados sobre as propriedades mecânicas deste polímero, e as implicações das propriedades mecânicas do DNA nos processos celulares é um tópico de pesquisa atual ativa.
O DNA encontrado em muitas células pode ter comprimento macroscópico: alguns centímetros de comprimento para cada cromossomo humano. Conseqüentemente, as células devem compactar ou empacotar o DNA para carregá-lo dentro delas. Nos eucariotos, isso é transportado por proteínas semelhantes a carretéis chamadas histonas , em torno das quais o DNA se enrola. É a compactação posterior desse complexo DNA-proteína que produz os cromossomos eucarióticos mitóticos bem conhecidos .
No final dos anos 1970, modelos alternativos não helicoidais da estrutura do DNA foram brevemente considerados como uma solução potencial para problemas na replicação do DNA em plasmídeos e cromatina . No entanto, os modelos foram deixados de lado em favor do modelo de dupla hélice devido aos avanços experimentais subsequentes, como a cristalografia de raios X de duplexes de DNA e, posteriormente, a partícula central do nucleossomo e a descoberta de topoisomerases . Esses modelos não-helicoidais não são atualmente aceitos pela comunidade científica convencional.
Determinação da estrutura do DNA usando modelagem molecular e padrões de raios-X de DNA
Depois que o DNA foi separado e purificado por métodos bioquímicos padrão, uma amostra foi colocada em um frasco, muito parecido com a figura no topo deste artigo. Abaixo estão as principais etapas envolvidas na geração de informações estruturais a partir de estudos de difração de raios-X de fibras de DNA orientadas que são extraídas da amostra de DNA hidratado com a ajuda de modelos moleculares de DNA que são combinados com análises cristalográficas e matemáticas dos padrões de raios-X .
Modelos de rede paracristalina de estruturas de B-DNA
Uma rede paracristalina , ou paracristal, é uma rede molecular ou atômica com quantidades significativas (por exemplo, maiores do que alguns por cento) de desordem parcial de arranjos moleculares. Casos limites do modelo paracristal são nanoestruturas , como vidros , líquidos , etc., que podem possuir apenas ordenação local e nenhuma ordem global. Um exemplo simples de uma rede paracristalina é mostrado na figura a seguir para um vidro de sílica:
Os cristais líquidos também têm estruturas paracristalinas em vez de estruturas cristalinas .
B-DNA altamente hidratado ocorre naturalmente em células vivas em um estado paracristalino, que é dinâmico, apesar da dupla hélice de DNA relativamente rígida estabilizada por ligações de hidrogênio paralelas entre os pares de bases de nucleotídeos nas duas cadeias de DNA helicoidais complementares (ver figuras ) Para simplificar, a maioria dos modelos moleculares de DNA omite água e íons dinamicamente ligados ao B-DNA e, portanto, são menos úteis para a compreensão dos comportamentos dinâmicos do B-DNA in vivo . A análise física e matemática de dados de raios-X e espectroscópicos para B-DNA paracristalino é, portanto, muito mais complexa do que os padrões de difração de raios-X A-DNA cristalinos. O modelo paracristal também é importante para aplicações tecnológicas de DNA, como nanotecnologia de DNA . Novos métodos que combinam difração de raios-X de DNA com microscopia de raios-X em células vivas hidratadas também estão sendo desenvolvidos.
Aplicações genômicas e biotecnológicas da modelagem molecular de DNA

Existem vários usos de modelagem molecular de DNA em aplicações de pesquisa em Genômica e Biotecnologia, de reparo de DNA a PCR e nanoestruturas de DNA . Matrizes de junção de DNA bidimensional foram visualizadas por microscopia de força atômica .
A modelagem molecular de DNA tem vários usos em genômica e biotecnologia , com aplicações de pesquisa que variam de reparo de DNA a PCR e nanoestruturas de DNA. Estes incluem modelos moleculares de computador de moléculas tão variadas quanto RNA polimerase, uma E. coli, modelo de DNA primase bacteriano sugerindo uma dinâmica muito complexa nas interfaces entre as enzimas e o modelo de DNA e modelos moleculares da interação química mutagênica de moléculas potentes de carcinógeno com DNA. Todos eles estão representados na galeria abaixo.
A aplicação tecnológica inclui um biochip de DNA e nanoestruturas de DNA projetados para computação de DNA e outras aplicações dinâmicas de nanotecnologia de DNA . A imagem à direita é de nanoestruturas de DNA automontadas. A estrutura de "ladrilhos" do DNA nesta imagem consiste em quatro junções ramificadas orientadas em ângulos de 90 °. Cada bloco consiste em nove oligonucleotídeos de DNA, conforme mostrado; tais ladrilhos servem como o "bloco de construção" primário para a montagem das nanogrelhas de DNA mostradas na micrografia AFM.
O DNA quádruplo pode estar envolvido em certos tipos de câncer. Imagens do DNA quádruplo estão na galeria abaixo.
Galeria de modelos de DNA
Modelo de DNA de 15 m de comprimento, Naturalis Biodiversity Center
Veja também
- G-quadruplex
- Cristalografia
- Redes de cristal
- Banco de dados de propriedades de dinucleotídeos ( DiProDB ), projetado para coletar e analisar características termodinâmicas, estruturais e outras características de dinucleotídeos
- Microscopia de raios X
- Espalhamento de raios-x
- Dispersão de nêutrons
- Sequência de ácido nucléico
- Dicroísmo circular vibracional (VCD)
- Espectroscopia Raman -microscopia e espectroscopia Raman anti-Stokes coerente (CARS)
- Sir Lawrence Bragg , FRS
- Comparação de software de simulação de ácido nucleico
- ÂMBAR
- CHARMM
- Abalone (mecânica molecular)
- Software de visualização Sirius
- QMC @ Home
- Espectroscopia de NMR (FT-NMR)
- Microscopia de imagem NMR
- Espectroscopia de micro-ondas
- FT- infravermelho (IR)
- FT- espectroscopia de infravermelho próximo (NIR)
- Imagens espectrais , imagens hiperespectrais , imagens químicas
- Espectroscopia de correlação de fluorescência
- Espectroscopia de correlação cruzada de fluorescência e transferência de energia de ressonância de Förster (FRET)
- Microscopia confocal
- Modelos obsoletos da estrutura do DNA
Referências
Leitura adicional
- Aplicações de novas técnicas para alimentos saudáveis, biotecnologia médica e agrícola . (Junho de 2004) IC Baianu, PR Lozano, VI Prisecaru e HC Lin., Q-bio / 0406047.
- F. Bessel, Untersuchung des Theils der planetarischen Störungen , Berlin Abhandlungen (1824), artigo 14.
- Sir Lawrence Bragg, FRS. O estado cristalino, um levantamento geral. Londres: G. Bells and Sons, Ltd., vols. 1 e 2., 1966., 2024 páginas.
- Cantor, CR e Schimmel, PR Biophysical Chemistry, Parts I e II ., San Francisco: WH Freeman and Co. 1980. 1.800 páginas.
- Voet, D. e JG Voet. Biochemistry , 2nd Edn., New York, Toronto, Singapore: John Wiley & Sons, Inc., 1995, ISBN 0-471-58651-X ., 1361 páginas.
- Watson, GN A Treatise on the Theory of Bessel Functions ., (1995) Cambridge University Press. ISBN 0-521-48391-3 .
- Watson, James D. Molecular Biology of the Gene. New York e Amsterdam: WA Benjamin, Inc. 1965., 494 páginas.
- Wentworth, WE Physical Chemistry. Um pequeno curso. , Malden (Mass.): Blackwell Science, Inc. 2000.
- Herbert R. Wilson, FRS. Diffraction of X-rays by protein, Nucleic Acids and Viruses ., London: Edward Arnold (Publishers) Ltd. 1966.
- Kurt Wuthrich. RMN de proteínas e ácidos nucléicos. , New York, Brisbane, Chicester, Toronto, Singapore: J. Wiley & Sons. 1986., 292 páginas.
- Hallin PF, David Ussery D (2004). "CBS Genome Atlas Database: Um armazenamento dinâmico para resultados bioinformáticos e dados de sequência de DNA" . Bioinformática . 20 (18): 3682–6. doi : 10.1093 / bioinformática / bth423 . PMID 15256401 .
- Zhang CT, Zhang R, Ou HY (2003). "O banco de dados da curva Z: uma representação gráfica das sequências do genoma" . Bioinformática . 19 (5): 593–599. doi : 10.1093 / bioinformática / btg041 . PMID 12651717 .
links externos
- DNA the Double Helix Game Do site oficial do Prêmio Nobel
- MDDNA: Bioinformática Estrutural do DNA
- Double Helix 1953–2003 National Center for Biotechnology Education
- DNAlive: uma interface web para calcular as propriedades físicas do DNA . Também permite a ligação cruzada dos resultados com o navegador do genoma UCSC e a dinâmica do DNA.
- Mais detalhes da análise matemática e molecular da estrutura do DNA com base em dados de raios-X
- Funções de Bessel correspondentes às transformadas de Fourier de hélices atômicas ou moleculares.
- visão geral dos princípios STM / AFM / SNOM com vídeos educativos
Bancos de dados para sequências e modelos moleculares de DNA
- Difração de raios X
- ID do NDB: banco de dados UD0017
- Atlas de raios-X - banco de dados
- Arquivos PDB de coordenadas para estruturas de ácido nucléico de difração de raios-X por cristais de NA (incl. DNA)
- Fatores de estrutura de arquivos para download em formato CIF
- Dispersão de nêutrons
- Fonte de nêutrons ISIS : fonte de nêutrons pulsados ISIS: Um centro mundial para a ciência com nêutrons e múons em Harwell, perto de Oxford, Reino Unido.
- Microscopia de raios X
- Microscópio eletrônico
- Bancos de dados NMR
- Atlas NMR - banco de dados
- Arquivos de coordenadas mmcif para download de ácidos nucléicos em solução a partir de dados 2D-FT NMR
- Arquivos de restrições de NMR para NAs em formato PDB
- Bancos de dados genômicos e estruturais
- CBS Genome Atlas Database - contém exemplos de inclinações de base.
- O banco de dados da curva Z de genomas - uma ferramenta de visualização e análise tridimensional de genomas .
- Modelos moleculares de DNA e outros ácidos nucleicos: arquivos de coordenadas de modelos de estrutura molecular de ácidos nucleicos nos formatos PDB e CIF
- Força atômica microscópica