Análogo de ácido nucléico - Nucleic acid analogue
Os análogos de ácido nucléico são compostos análogos (estruturalmente semelhantes) ao RNA e DNA de ocorrência natural , usados na medicina e na pesquisa em biologia molecular. Os ácidos nucléicos são cadeias de nucleotídeos, que são compostas de três partes: uma estrutura de fosfato , um açúcar pentose, ribose ou desoxirribose , e uma de quatro nucleobases . Um análogo pode ter qualquer um deles alterado. Normalmente, as nucleobases analógicas conferem, entre outras coisas, diferentes propriedades de emparelhamento e empilhamento de bases. Os exemplos incluem bases universais, que podem emparelhar com todas as quatro bases canônicas, e análogos de estrutura fosfato-açúcar, como PNA , que afetam as propriedades da cadeia (PNA pode até formar uma hélice tripla ). Os análogos do ácido nucléico também são chamados de Ácido Nucleico Xeno e representam um dos principais pilares da xenobiologia , o design de formas de vida novas para a natureza com base em bioquímicas alternativas.
Os ácidos nucléicos artificiais incluem ácido nucléico peptídico (PNA), Morfolino e ácido nucléico bloqueado (LNA), bem como ácido nucléico glicol (GNA), ácido nucléico treose (TNA) e ácidos nucléicos hexitol (HNA). Cada um deles se distingue do DNA ou RNA de ocorrência natural por mudanças na estrutura da molécula.
Em maio de 2014, os pesquisadores anunciaram que haviam introduzido com sucesso dois novos nucleotídeos artificiais no DNA bacteriano e, ao incluir nucleotídeos artificiais individuais no meio de cultura, conseguiram passar as bactérias 24 vezes; eles não criaram mRNA ou proteínas capazes de usar os nucleotídeos artificiais. Os nucleotídeos artificiais apresentavam 2 anéis aromáticos fundidos.
Medicina
Vários análogos de nucleosídeos são usados como agentes antivirais ou anticâncer. A polimerase viral incorpora esses compostos com bases não canônicas. Esses compostos são ativados nas células ao serem convertidos em nucleotídeos, eles são administrados como nucleosídeos, uma vez que nucleotídeos carregados não podem atravessar facilmente as membranas celulares.
Biologia molecular
Análogos de ácido nucléico são usados em biologia molecular para vários propósitos: Investigação de possíveis cenários da origem da vida: testando diferentes análogos, os pesquisadores tentam responder se o uso de DNA e RNA na vida foi selecionado ao longo do tempo devido às suas vantagens, ou se foram escolhidos por acaso arbitrário; Como uma ferramenta para detectar sequências particulares: XNA pode ser usado para marcar e identificar uma ampla gama de componentes de DNA e RNA com alta especificidade e precisão; Como uma enzima que atua sobre substratos de DNA, RNA e XNA - XNA demonstrou ter a capacidade de clivar e ligar DNA, RNA e outras moléculas de XNA semelhantes às ações de ribozimas de RNA ; Como ferramenta com resistência à hidrólise de RNA ; Investigação dos mecanismos usados pela enzima; Investigação das características estruturais dos ácidos nucléicos.
Análogos de backbone
Análogos de RNA resistentes à hidrólise
Para superar o fato de que o grupo hidroxi 2 'da ribose que reage com o grupo hidroxi 3' ligado ao fosfato (o RNA é muito instável para ser usado ou sintetizado de forma confiável), um análogo da ribose é usado. Os análogos de RNA mais comuns são RNA substituído por 2'-O-metil, ácido nucleico bloqueado (LNA) ou ácido nucleico em ponte (BNA), morfolino e ácido nucleico peptídico ( PNA ). Embora esses oligonucleotídeos tenham uma estrutura de açúcar diferente ou, no caso do PNA, um resíduo de aminoácido no lugar do fosfato de ribose, eles ainda se ligam ao RNA ou DNA de acordo com o emparelhamento de Watson e Crick, mas são imunes à atividade de nuclease. Eles não podem ser sintetizados enzimaticamente e só podem ser obtidos sinteticamente usando a estratégia de fosforamidita ou, para PNA, métodos de síntese de peptídeos .
Outros análogos notáveis usados como ferramentas
Os didesoxinucleotídeos são usados no sequenciamento . Esses trifosfatos de nucleosídeos possuem um açúcar não canônico, a didesoxirribose, que não possui o grupo hidroxila 3 'normalmente presente no DNA e, portanto, não pode se ligar à próxima base. A falta do grupo hidroxila 3 'termina a reação em cadeia, pois as DNA polimerases a confundem com um desoxirribonucleotídeo regular. Outro análogo de terminação de cadeia que carece de um hidroxila 3 'e imita a adenosina é chamado cordicepina . Cordycepin é uma droga anticâncer que tem como alvo a replicação do RNA . Outro análogo no sequenciamento é um análogo de nucleobase, 7-deaza-GTP e é usado para sequenciar regiões ricas em CG, em vez de 7-deaza-ATP é chamado de tubercidina , um antibiótico.
Precursores do mundo do RNA
O RNA pode ser muito complexo para ser o primeiro ácido nucléico, portanto, antes do mundo do RNA, vários ácidos nucléicos mais simples que diferem na estrutura, como TNA e GNA e PNA , foram oferecidos como candidatos para os primeiros ácidos nucléicos.
Análogos de base
Estrutura e nomenclatura da nucleobase
As bases que ocorrem naturalmente podem ser divididas em duas classes de acordo com sua estrutura:
- as pirimidinas são heterocíclicas de seis membros com átomos de nitrogênio nas posições 1 e 3.
- as purinas são bicíclicas, consistindo de uma pirimidina fundida a um anel imidazol.
Nucleotídeos artificiais ( pares de bases não naturais (UBPs) denominados d5SICS UBP e dNaM UBP ) foram inseridos no DNA bacteriano, mas esses genes não moldaram mRNA ou induziram a síntese de proteínas. Os nucleotídeos artificiais apresentavam dois anéis aromáticos fundidos que formaram um complexo (d5SICS – dNaM) imitando o par de bases natural (dG – dC).
Mutagênicos
Um dos análogos de base mais comuns é o 5-bromouracil (5BU), a base anormal encontrada no análogo de nucleotídeo mutagênico BrdU. Quando um nucleotídeo contendo 5-bromouracil é incorporado ao DNA, é mais provável que ele pareie com a adenina; no entanto, ele pode mudar espontaneamente para outro isômero que emparelha com uma nucleobase diferente , a guanina . Se isso acontecer durante a replicação do DNA, uma guanina será inserida como o análogo da base oposta e, na próxima replicação do DNA, essa guanina irá emparelhar com uma citosina. Isso resulta em uma mudança em um par de bases de DNA, especificamente uma mutação de transição .
Além disso, o HNO2, ou ácido nitroso, é um potente mutagênico que atua na replicação e não replicação do DNA. Pode causar desaminação dos grupos amino da Adenina, Guanina e Citosina. A adenina é desaminada em hipoxantina , que forma pares de bases em citosina em vez de timina. A citosina é desaminada em uracila, que forma pares de bases com adenina em vez de guanina. A desaminação da Guanina não é mutagênica. Mutações induzidas por ácido nitroso também são induzidas a sofrer mutação de volta ao tipo selvagem usando ácido nitroso.
Fluoróforos
Comumente os fluoróforos (como a rodamina ou a fluoresceína ) estão ligados ao anel ligado ao açúcar (em para) por meio de um braço flexível, presumivelmente extrudando do sulco principal da hélice. Devido à baixa processabilidade dos nucleotídeos ligados a adutos volumosos, como floróforos por taq polimerases, a sequência é tipicamente copiada usando um nucleotídeo com um braço e posteriormente acoplado com um fluoróforo reativo (marcação indireta):
- amina reativa: O nucleotídeo aminoalil contém um grupo amina primária em um ligante que reage com o corante amino reativo, como cianina ou corantes Alexa Fluor , que contêm um grupo abandonante reativo, como um éster succinimidílico (NHS). (os grupos amino de emparelhamento de bases não são afetados).
- tiol reativo: os nucleotídeos contendo tiol reagem com o fluoróforo ligado a um grupo abandonante reativo, como uma maleimida.
- os nucleotídeos ligados à biotina contam com o mesmo princípio de marcação indireta (+ estreptavidina fluorescente) e são usados em chips de DNA da Affymetrix .
Os fluoróforos encontram uma variedade de usos na medicina e na bioquímica.
Análogos de base fluorescente
O análogo de base fluorescente mais comumente usado e comercialmente disponível, 2-aminopurina (2-AP), tem um rendimento quântico de alta fluorescência livre em solução (0,68) que é consideravelmente reduzido (aprox. 100 vezes, mas altamente dependente da sequência de base) quando incorporado em ácidos nucléicos. A sensibilidade de emissão de 2-AP ao ambiente imediato é compartilhada por outros análogos de base fluorescente promissores e úteis como 3-MI, 6-MI, 6-MAP, pirrolo-dC (também disponível comercialmente), derivados modificados e melhorados de pirrolo-dC , bases modificadas com furano e muitos outros (ver análises recentes). Essa sensibilidade ao microambiente tem sido utilizada em estudos de, por exemplo, estrutura e dinâmica dentro do DNA e do RNA, dinâmica e cinética da interação DNA-proteína e transferência de elétrons dentro do DNA.
Um grupo recentemente desenvolvido e muito interessante de análogos de base fluorescentes que tem um rendimento quântico de fluorescência que é quase insensível ao seu entorno imediato é a família da citosina tricíclica. 1,3-Diaza-2-oxofenotiazina, tC, tem um rendimento quântico de fluorescência de aproximadamente 0,2 tanto em fita simples quanto em fita dupla, independentemente das bases circundantes. Além disso, o oxo-homólogo de tC denominado tC O (ambos disponíveis comercialmente), 1,3-diaza-2-oxofenoxazina, tem um rendimento quântico de 0,2 em sistemas de fita dupla. No entanto, é um pouco sensível às bases circundantes em fitas simples (rendimentos quânticos de 0,14–0,41). Os rendimentos quânticos altos e estáveis desses análogos de base os tornam muito brilhantes e, em combinação com suas boas propriedades de análogos de base (deixa a estrutura e estabilidade do DNA quase imperturbável), eles são especialmente úteis em anisotropia de fluorescência e medições FRET, áreas onde outros análogos de base fluorescente são menos precisos. Além disso, na mesma família de análogos de citosina , foi desenvolvido um análogo de base aceptora de FRET, tC nitro . Juntamente com tC O como um doador de FRET, isso constitui o primeiro par de FRET de análogo de base de ácido nucleico já desenvolvido. A família tC tem, por exemplo, sido usada em estudos relacionados com a ligação ao DNA da polimerase e mecanismos de polimerização do DNA.
Bases naturais não canônicas
Em uma célula, existem várias bases não canônicas presentes: ilhas CpG no DNA (são frequentemente metiladas), todo mRNA eucariótico (coberto com uma metil-7-guanosina) e várias bases de rRNAs (são metiladas). Freqüentemente, os tRNAs são fortemente modificados pós-transcricionalmente a fim de melhorar sua conformação ou pareamento de bases, em particular no / próximo ao anticódon: a inosina pode parear com C, U e até mesmo com A, enquanto a tiouridina (com A) é mais específica do que o uracil (com uma purina). Outras modificações de base de tRNA comuns são pseudouridina (que dá seu nome ao loop TΨC), dihidrouridina (que não se acumula porque não é aromática), queuosina, wiosina e assim por diante. No entanto, todas essas são modificações em bases normais e não são colocadas por uma polimerase.
Emparelhamento de base
Bases canónicas pode ter quer um grupo carbonilo ou um grupo amina sobre os carbonos que envolvem o átomo de azoto mais afastado da ligação glicosídica, o que lhes permite ao par de bases (Watson-Crick), através de ligações de hidrogénio (amina com a cetona, purina com pirimidina) . A adenina e a 2-aminoadenina têm um / dois grupo (s) amina, enquanto a timina tem dois grupos carbonila, e a citosina e a guanina são amina e carbonila misturadas (invertidas uma em relação à outra).
| Pares de base naturais | |
|---|---|

|

|
| Um par de base GC: purina carbonil / amina forma três ligações de hidrogênio intermoleculares com pirimidina amina / carbonil | Um par de base AT: purina amina / - forma duas ligações de hidrogênio intermoleculares com pirimidina carbonil / carbonil |
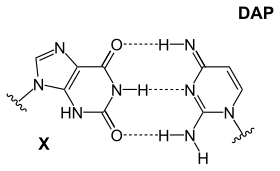
A razão exata pela qual existem apenas quatro nucleotídeos é debatida, mas existem várias possibilidades não utilizadas. Além disso, a adenina não é a escolha mais estável para o emparelhamento de bases: no Cyanophage S-2L, a diaminopurina (DAP) é usada em vez da adenina ( evasão do hospedeiro ). Pares de base da diaminopurina perfeitamente com timina, pois é idêntica à adenina, mas tem um grupo amina na posição 2 formando 3 ligações de hidrogênio intramoleculares, eliminando a principal diferença entre os dois tipos de pares de base (Fraco: AT e Forte: CG). Esta estabilidade melhorada afeta as interações de ligação de proteínas que dependem dessas diferenças. Outras combinações incluem,
- isoguanina e isocitosina, que têm sua amina e cetona invertidas em comparação com a guanina e citosina padrão (provavelmente não usados como tautômeros são problemáticos para o emparelhamento de bases, mas isoC e isoG podem ser amplificados corretamente com PCR, mesmo na presença das 4 bases canônicas)
- diaminopirimidina e uma xantina, que se ligam como 2-aminoadenina e timina, mas com estruturas invertidas (não usada porque a xantina é um produto de desaminação)
| Arranjos de pares de base não utilizados | ||
|---|---|---|

|
|

|
| Uma base DAP-T: purina amina / amina forma três ligações de hidrogênio intermoleculares com pirimidina cetona / cetona | Uma base X-DAP: purina cetona / cetona forma três ligações de hidrogênio intermoleculares com pirimidina amina / amina | Uma base iG-iC: purina amina / cetona forma três ligações de hidrogênio intermoleculares com pirimidina cetona / amina |
No entanto, a estrutura correta do DNA pode se formar mesmo quando as bases não estão emparelhadas por meio de ligações de hidrogênio; ou seja, o par de bases graças à hidrofobicidade, como os estudos mostraram usando isósteros de DNA (análogos com o mesmo número de átomos), como o análogo de timina 2,4-difluorotolueno (F) ou o análogo de adenina 4-metilbenzimidazol (Z). Um par hidrofóbico alternativo poderia ser a isoquinolina e a pirrolo [2,3-b] piridina
Outros pares de base dignos de nota:
- Também foram feitas várias bases fluorescentes, como o par de bases 2-amino-6- (2-tienil) purina e pirrol-2-carbaldeído.
- Bases coordenadas por metal, como o emparelhamento entre um piridina-2,6-dicarboxilato (ligante tridentado) e uma piridina (ligante monodentado) por meio de coordenação quadrada plana a um íon de cobre central.
- As bases universais podem emparelhar indiscriminadamente com qualquer outra base, mas, em geral, diminuem consideravelmente a temperatura de fusão da sequência; exemplos incluem derivados de 2'-desoxinucleotídeo (hipoxantina desoxinucleotídeo), análogos de nitroazol e bases hidrofóbicas aromáticas de não ligação de hidrogênio (fortes efeitos de empilhamento). São utilizados como prova de conceito e, em geral, não são utilizados em primers degenerados (que são uma mistura de primers).
- O número de pares de bases possíveis é duplicado quando o xDNA é considerado. O xDNA contém bases expandidas, nas quais foi adicionado um anel de benzeno, que pode emparelhar com bases canônicas, resultando em quatro pares de bases possíveis (8 bases: xA-T, xT-A, xC-G, xG-C, 16 bases se os arranjos não utilizados forem usados). Outra forma de bases adicionadas com benzeno é o yDNA, em que a base é alargada pelo benzeno.
| Pares de base novos com propriedades especiais | ||
|---|---|---|

|

|

|
| Uma base FZ: metilbenzimidazol não forma ligações de hidrogênio intermoleculares com tolueno F / F | Uma base S-Pa: purina tienil / amina forma três ligações de hidrogênio intermoleculares com pirrol - / carbaldeído | Uma base xA-T: mesma ligação que AT |
Pares de bases de metal
No pareamento de bases metálicas, as ligações de hidrogênio Watson-Crick são substituídas pela interação entre um íon metálico com nucleosídeos atuando como ligantes. As geometrias possíveis do metal que permitiriam a formação de duplex com dois nucleosídeos bidentados ao redor de um átomo de metal central são: tetraédrica , dodecaédrica e quadrada plana . A complexação do metal com o DNA pode ocorrer pela formação de pares de bases não canônicos de nucleobases naturais com a participação de íons metálicos e também pela troca dos átomos de hidrogênio que fazem parte do par de bases Watson-Crick por íons metálicos. A introdução de íons metálicos em um duplex de DNA demonstrou possuir potencial magnético, propriedades condutoras, bem como estabilidade aumentada.
Foi demonstrado que a complexação de metais ocorre entre nucleobases naturais . Um exemplo bem documentado é a formação de T-Hg-T, que envolve duas timina nucleobases desprotonadas que são reunidas por Hg 2+ e formam um par metal-base conectado. Este motivo não acomoda Hg 2+ empilhado em um duplex devido a um processo de formação de grampo de cabelo intracadeia que é favorecido em relação à formação de duplex. Duas timas uma em frente à outra em um duplex não formam um par de bases Watson-Crick em um duplex; este é um exemplo em que uma incompatibilidade de pares de base Watson-Crick é estabilizada pela formação do par metal-base. Outro exemplo de um metal complexado com nucleobases naturais é a formação de A-Zn-T e G-Zn-C em pH alto; Co +2 e Ni +2 também formam esses complexos. Estes são pares de bases Watson-Crick onde o cátion divalente está coordenado com as nucleobases. A ligação exata é debatida.
Uma grande variedade de nucleobases artificiais foi desenvolvida para uso como pares de bases de metal. Essas nucleobases modificadas exibem propriedades eletrônicas ajustáveis, tamanhos e afinidades de ligação que podem ser otimizadas para um metal específico. Por exemplo, um nucleosídeo modificado com uma piridina-2,6-dicarboxilato mostrou se ligar fortemente a Cu 2+ , enquanto outros íons divalentes estão apenas fracamente ligados. O personagem tridentado contribui para essa seletividade. O quarto sítio de coordenação no cobre é saturado por uma piridina nucleobase opostamente disposta. O sistema de pareamento de bases de metal assimétrico é ortogonal aos pares de bases Watson-Crick. Outro exemplo de nucleobase artificial é aquela com nucleobases de hidroxipiridona, que são capazes de se ligar a Cu 2+ dentro do duplex de DNA. Cinco pares de bases de cobre-hidroxipiridona consecutivos foram incorporados em uma fita dupla, que foram flanqueados por apenas uma nucleobase natural em ambas as extremidades. Os dados de EPR mostraram que a distância entre os centros de cobre foi estimada em 3,7 ± 0,1 Å, enquanto um duplex de DNA do tipo B natural é apenas ligeiramente maior (3,4 Å). O apelo para empilhar íons de metal dentro de um duplex de DNA é a esperança de obter fios de metal nanoscópicos com automontagem, embora isso ainda não tenha sido realizado.
Par de bases não naturais (UBP)
Um par de bases não naturais (UBP) é uma subunidade (ou nucleobase ) projetada de DNA que é criada em um laboratório e não ocorre na natureza. Em 2012, um grupo de cientistas americanos liderados por Floyd Romesberg, biólogo químico do Scripps Research Institute em San Diego, Califórnia, publicou que sua equipe projetou um par de bases não naturais (UBP). Os dois novos nucleotídeos artificiais ou Unnatural Base Pair (UBP) foram denominados d5SICS e dNaM . Mais tecnicamente, esses nucleotídeos artificiais com nucleobases hidrofóbicas apresentam dois anéis aromáticos fundidos que formam um complexo (d5SICS-dNaM) ou par de bases no DNA. Em 2014, a mesma equipe do Scripps Research Institute relatou que sintetizou um trecho de DNA circular conhecido como plasmídeo contendo pares de bases naturais de TA e CG, juntamente com o UBP de melhor desempenho que o laboratório de Romesberg havia projetado, e o inseriu em células do comum bactéria E. coli que replicou com sucesso os pares de bases não naturais ao longo de várias gerações. Este é o primeiro exemplo conhecido de um organismo vivo transmitindo um código genético expandido para as gerações subsequentes. Isso foi em parte conseguido pela adição de um gene de algas de suporte que expressa um transportador de trifosfato de nucleotídeo que importa eficientemente os trifosfatos de d5SICSTP e dNaMTP em bactérias E. coli . Em seguida, as vias de replicação bacteriana natural usam-nas para replicar com precisão o plasmídeo contendo d5SICS – dNaM.
A incorporação bem-sucedida de um terceiro par de bases é um avanço significativo em direção ao objetivo de expandir muito o número de aminoácidos que podem ser codificados pelo DNA, dos 20 aminoácidos existentes para um 172 teoricamente possível, expandindo assim o potencial dos organismos vivos para produzir novas proteínas . Anteriormente, as cadeias artificiais de DNA não codificavam para nada, mas os cientistas especularam que poderiam ser projetadas para fabricar novas proteínas que poderiam ter usos industriais ou farmacêuticos. A transcrição do DNA contendo o par de bases não naturais e a tradução do mRNA correspondente foram realmente alcançadas recentemente. Em novembro de 2017, a mesma equipe do Scripps Research Institute que introduziu duas nucleobases extras no DNA bacteriano, relatou ter construído uma bactéria E. coli semissintética capaz de fazer proteínas usando esse DNA. Seu DNA continha seis nucleobases diferentes : quatro canônicas e duas adicionadas artificialmente, dNaM e dTPT3 (as duas formam um par). Além disso, essa bactéria tinha duas bases de RNA adicionais correspondentes incluídas em dois novos códons, tRNAs adicionais que reconheciam esses novos códons (esses tRNAs também continham duas novas bases de RNA dentro de seus anticódons) e aminoácidos adicionais, tornando a bactéria capaz de sintetizar proteínas "não naturais" .
Outra demonstração de UBPs foi realizada pelo grupo de Ichiro Hirao no instituto RIKEN no Japão. Em 2002, eles desenvolveram um par de bases não natural entre 2-amino-8- (2-tienil) purina (s) e piridina-2-ona (y) que funciona in vitro na transcrição e tradução, para a incorporação específica do local de aminoácidos não padronizados em proteínas. Em 2006, eles criaram 7- (2-tienil) imidazo [4,5-b] piridina (Ds) e pirrol-2-carbaldeído (Pa) como um terceiro par de bases para replicação e transcrição. Posteriormente, Ds e 4- [3- (6-aminohexanamido) -1-propinil] -2-nitropirrol (Px) foi descoberto como um par de alta fidelidade na amplificação por PCR. Em 2013, eles aplicaram o par Ds-Px para geração de aptâmero de DNA por seleção in vitro (SELEX) e demonstraram que a expansão do alfabeto genético aumenta significativamente as afinidades de aptâmero de DNA para proteínas-alvo.
Sistema ortogonal
Foi proposta e estudada a possibilidade, tanto teórica como experimentalmente, de implementar um sistema ortogonal no interior das células independente do material genético celular de forma a constituir um sistema totalmente seguro, com o possível aumento dos potenciais de codificação. Vários grupos se concentraram em diferentes aspectos:
- novos backbones e pares de bases, conforme discutido acima
- Polimerases de replicação / transcrição artificial XNA ( Ácido Xeno Nucleico ) começando geralmente a partir da polimerase de RNA T7
- ribossomos ( sequências 16S com sequência alterada anti -Shine-Dalgarno, permitindo a tradução de apenas mRNA ortogonal com uma sequência Shine-Dalgarno alterada correspondente)
- novo tRNA que codifica aminoácidos não naturais. Veja código genético expandido
Veja também
- Biotina
- Quencher escuro
- Desoxirribozima
- Código genético expandido
- Fluoróforo
- Genética
- Biologia molecular
- Ácido nucleico
- Nucleobase
- Nucleosídeo
- Nucleotide
- Síntese de oligonucleotídeos
- Ribozima
- Biologia sintética
- Xenobiologia
- xDNA
- DNA de Hachimoji
- Sistema de Informação Genética Artificialmente Expandido (AEGIS)
- Ácido xeno nucléico