Nucleossomo - Nucleosome

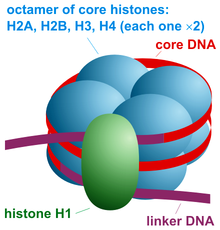
Um nucleossomo é a unidade estrutural básica de empacotamento de DNA em eucariotos . A estrutura de um nucleossomo consiste em um segmento de DNA enrolado em torno de oito proteínas histonas e lembra um fio enrolado em um carretel. O nucleossomo é a subunidade fundamental da cromatina . Cada nucleossomo é composto por um pouco menos de duas voltas de DNA envolvidas em um conjunto de oito proteínas chamadas histonas, conhecidas como octâmero de histona . Cada octâmero de histona é composto por duas cópias de cada uma das proteínas histonas H2A , H2B , H3 e H4 .
O DNA deve ser compactado em nucleossomos para caber no núcleo da célula . Além do envolvimento do nucleossomo, a cromatina eucariótica é ainda mais compactada ao ser dobrada em uma série de estruturas mais complexas, eventualmente formando um cromossomo . Cada célula humana contém cerca de 30 milhões de nucleossomos.
Acredita-se que os nucleossomos carreguem informações herdadas epigeneticamente na forma de modificações covalentes de suas histonas centrais . As posições dos nucleossomos no genoma não são aleatórias e é importante saber onde cada nucleossomo está localizado, pois isso determina a acessibilidade do DNA às proteínas regulatórias .
Os nucleossomos foram observados pela primeira vez como partículas no microscópio eletrônico por Don e Ada Olins em 1974, e sua existência e estrutura (como octâmeros de histona cercados por aproximadamente 200 pares de bases de DNA) foram propostas por Roger Kornberg . O papel do nucleossomo como um repressor genético geral foi demonstrado por Lorch et al. in vitro, e por Han e Grunstein in vivo em 1987 e 1988, respectivamente.
A partícula central do nucleossomo consiste em aproximadamente 146 pares de bases (pb) de DNA envolto em 1,67 voltas super - helicoidais canhotas em torno de um octâmero de histona , consistindo em 2 cópias de cada uma das histonas centrais H2A , H2B , H3 e H4 . As partículas centrais são conectadas por trechos de DNA ligante , que podem ter até cerca de 80 bp de comprimento. Tecnicamente, um nucleossomo é definido como a partícula central mais uma dessas regiões de ligação; entretanto, a palavra é freqüentemente sinônima com a partícula central. Mapas de posicionamento de nucleossomos de todo o genoma estão agora disponíveis para muitos organismos modelo, incluindo fígado e cérebro de camundongo.
Histonas ligantes, como H1 e suas isoformas, estão envolvidas na compactação da cromatina e ficam na base do nucleossomo próximo à entrada e saída do DNA, ligando-se à região ligante do DNA. Os nucleossomos não condensados sem a histona ligante se assemelham a "contas em uma cadeia de DNA" sob um microscópio eletrônico .
Em contraste com a maioria das células eucarióticas, os espermatozoides maduros usam amplamente protaminas para empacotar seu DNA genômico, provavelmente para atingir uma proporção de empacotamento ainda maior. Equivalentes de histonas e uma estrutura de cromatina simplificada também foram encontrados em Archaea , sugerindo que os eucariotos não são os únicos organismos que usam nucleossomos.
Estrutura
Estrutura da partícula central
Visão geral
Estudos estruturais pioneiros na década de 1980 pelo grupo de Aaron Klug forneceram a primeira evidência de que um octâmero de proteínas histonas envolve o DNA em torno de si mesmo em cerca de 1,7 voltas de uma superhélice canhota. Em 1997, a primeira estrutura cristalina de resolução quase atômica do nucleossomo foi resolvida pelo grupo de Richmond, mostrando os detalhes mais importantes da partícula. O DNA palindrômico do satélite alfa humano, crítico para alcançar a estrutura de cristal do nucleossomo de 1997, foi desenvolvido pelo grupo Bunick no Laboratório Nacional de Oak Ridge, no Tennessee. As estruturas de mais de 20 partículas centrais de nucleossomos diferentes foram resolvidas até o momento, incluindo aquelas contendo variantes de histonas e histonas de diferentes espécies. A estrutura da partícula central do nucleossomo é notavelmente conservada, e mesmo uma mudança de mais de 100 resíduos entre as histonas de rã e levedura resulta em mapas de densidade de elétrons com um desvio quadrático médio da raiz geral de apenas 1,6Å.
A partícula central do nucleossomo (NCP)
A partícula central do nucleossomo (mostrada na figura) consiste em cerca de 146 pares de bases de DNA envolvidos em 1,67 voltas super - helicoidais canhotas em torno do octâmero da histona , consistindo em 2 cópias de cada uma das histonas centrais H2A , H2B , H3 e H4 . Os nucleossomos adjacentes são unidos por um trecho de DNA livre denominado DNA ligante (que varia de 10 a 80 bp de comprimento dependendo da espécie e do tipo de tecido). Toda a estrutura gera um cilindro de diâmetro de 11 nm e altura de 5,5 nm.

As partículas do núcleo do nucleossomo são observadas quando a cromatina em interfase é tratada para fazer com que a cromatina se desdobre parcialmente. A imagem resultante, por meio de um microscópio eletrônico, é "contas em um cordão". O fio é o DNA, enquanto cada conta do nucleossomo é uma partícula central. A partícula central do nucleossomo é composta de proteínas de DNA e histonas.
A digestão parcial da cromatina com DNAse revela sua estrutura de nucleossomo. Como as porções de DNA das partículas do núcleo do nucleossomo são menos acessíveis para DNAse do que as seções de ligação, o DNA é digerido em fragmentos de comprimentos iguais à multiplicidade de distância entre os nucleossomos (180, 360, 540 pares de bases, etc.). Portanto, um padrão muito característico semelhante a uma escada é visível durante a eletroforese em gel desse DNA. Tal digestão pode ocorrer também em condições naturais durante a apoptose ("suicídio celular" ou morte celular programada), porque a autodestruição do DNA é tipicamente seu papel.
Interações de proteínas dentro do nucleossomo
As proteínas da histona central contêm um motivo estrutural característico denominado "dobra da histona", que consiste em três alfa-hélices (α1-3) separadas por duas voltas (L1-2). Em solução, as histonas formam heterodímeros H2A-H2B e heterotetrâmeros H3-H4. As histonas se dimerizam em torno de suas longas hélices α2 em uma orientação antiparalela e, no caso de H3 e H4, dois desses dímeros formam um feixe de 4 hélices estabilizado por extensa interação H3-H3 '. O dímero H2A / H2B se liga ao tetrâmero H3 / H4 devido às interações entre H4 e H2B, que incluem a formação de um cluster hidrofóbico. O octâmero de histona é formado por um tetrâmero H3 / H4 central imprensado entre dois dímeros H2A / H2B. Devido à carga altamente básica de todas as quatro histonas centrais, o octâmero da histona é estável apenas na presença de DNA ou concentrações de sal muito altas.
Histona - interações de DNA
O nucleossomo contém mais de 120 interações diretas de proteína-DNA e várias centenas de interações mediadas por água. As interações diretas de proteína - DNA não estão espalhadas uniformemente sobre a superfície do octâmero, mas sim localizadas em locais discretos. Estes são devidos à formação de dois tipos de sítios de ligação ao DNA dentro do octâmero; o local α1α1, que usa a hélice α1 de duas histonas adjacentes, e o local L1L2 formado pelas alças L1 e L2. Ligações de sal e ligações de hidrogênio entre os grupos básicos e hidroxila da cadeia lateral e as amidas da cadeia principal com os fosfatos do esqueleto do DNA formam a maior parte das interações com o DNA. Isso é importante, visto que a distribuição onipresente de nucleossomos ao longo dos genomas exige que ele seja um fator de ligação ao DNA não específico para a sequência. Embora os nucleossomos tendam a preferir algumas sequências de DNA a outras, eles são capazes de se ligar a praticamente qualquer sequência, o que se acredita ser devido à flexibilidade na formação dessas interações mediadas por água. Além disso, interações não polares são feitas entre as cadeias laterais de proteínas e os grupos de desoxirribose, e uma cadeia lateral de arginina intercala no sulco menor do DNA em todos os 14 locais onde está voltada para a superfície do octâmero. A distribuição e a força dos locais de ligação ao DNA sobre a superfície do octâmero distorcem o DNA dentro do núcleo do nucleossomo. O DNA não é uniformemente dobrado e também contém defeitos de torção. A torção do DNA na forma B livre em solução é de 10,5 bp por turno. No entanto, a torção geral do DNA nucleossômico é de apenas 10,2 bp por volta, variando de um valor de 9,4 a 10,9 bp por volta.
Domínios da cauda da histona
As extensões da cauda das histonas constituem até 30% da massa das histonas, mas não são visíveis nas estruturas cristalinas dos nucleossomos devido à sua alta flexibilidade intrínseca e foram consideradas amplamente não estruturadas. As caudas N-terminais das histonas H3 e H2B passam por um canal formado pelos sulcos menores das duas fitas de DNA, saindo do DNA a cada 20 bp. A cauda N-terminal da histona H4, por outro lado, possui uma região de aminoácidos altamente básicos (16-25), que, na estrutura cristalina, forma uma interação com a região de superfície altamente ácida de um dímero H2A-H2B de outro nucleossomo, sendo potencialmente relevante para a estrutura de ordem superior dos nucleossomos. Acredita-se que essa interação ocorra também em condições fisiológicas e sugere que a acetilação da cauda H4 distorce a estrutura de ordem superior da cromatina.
Estrutura de ordem superior
A organização do DNA que é alcançada pelo nucleossomo não pode explicar completamente o empacotamento de DNA observado no núcleo da célula. A compactação adicional da cromatina no núcleo da célula é necessária, mas ainda não é bem compreendida. O entendimento atual é que os nucleossomos repetidos com DNA "ligante" intermediário formam uma fibra de 10 nm , descrita como "contas em um cordão", e têm uma proporção de empacotamento de cerca de cinco a dez. Uma cadeia de nucleossomos pode ser organizada em uma fibra de 30 nm , uma estrutura compactada com uma taxa de empacotamento de ~ 50 e cuja formação depende da presença da histona H1 .
Uma estrutura cristalina de um tetranucleosome foi apresentada e usada para construir uma estrutura proposta da fibra de 30 nm como uma hélice de duas partidas. Ainda existe uma certa controvérsia em relação a este modelo, pois é incompatível com dados recentes de microscopia eletrônica . Além disso, a estrutura da cromatina é mal compreendida, mas é classicamente sugerido que a fibra de 30 nm é disposta em alças ao longo de uma estrutura protéica central para formar a eucromatina transcricionalmente ativa . A compactação posterior leva à heterocromatina transcricionalmente inativa .
Dinâmica
Embora o nucleossomo seja um complexo proteína-DNA muito estável, ele não é estático e foi demonstrado que sofre uma série de rearranjos estruturais diferentes, incluindo o deslizamento do nucleossomo e a exposição do local do DNA. Dependendo do contexto, os nucleossomos podem inibir ou facilitar a ligação do fator de transcrição. As posições dos nucleossomos são controladas por três contribuições principais: Primeiro, a afinidade de ligação intrínseca do octâmero da histona depende da sequência de DNA. Em segundo lugar, o nucleossomo pode ser deslocado ou recrutado pela ligação competitiva ou cooperativa de outros fatores proteicos. Terceiro, o nucleossomo pode ser translocado ativamente por complexos de remodelação dependentes de ATP.
Nucleossomo deslizante
Trabalho realizado no laboratório de Bradbury mostrou que os nucleossomos reconstituídos na sequência de posicionamento do DNA 5S foram capazes de se reposicionar translacionalmente nas sequências adjacentes quando incubados termicamente. Trabalhos posteriores mostraram que esse reposicionamento não exigia a interrupção do octâmero da histona, mas era consistente com o fato de os nucleossomos serem capazes de "deslizar" ao longo do DNA em cis . Em 2008, foi ainda revelado que os locais de ligação do CTCF atuam como âncoras de posicionamento de nucleossomos de modo que, quando usados para alinhar vários sinais genômicos, múltiplos nucleossomos de flanco podem ser facilmente identificados. Embora os nucleossomos sejam intrinsecamente móveis, os eucariotos desenvolveram uma grande família de enzimas de remodelação da cromatina dependentes de ATP para alterar a estrutura da cromatina, muitas das quais o fazem por meio do deslizamento do nucleossomo. Em 2012, o laboratório de Beena Pillai demonstrou que o deslizamento do nucleossoma é um dos possíveis mecanismos para a expressão de genes em tecidos específicos em larga escala. O trabalho mostra que o local de início da transcrição para genes expressos em um determinado tecido está depletado no nucleossomo, enquanto o mesmo conjunto de genes em outro tecido onde não são expressos está ligado ao nucleossomo.
Exposição do site de DNA
Trabalhos do laboratório Widom mostraram que o DNA nucleossômico está em equilíbrio entre um estado embrulhado e um não embalado. As medições dessas taxas usando FRET resolvido no tempo revelaram que o DNA dentro do nucleossomo permanece totalmente embrulhado por apenas 250 ms antes de ser desembrulhado por 10-50 ms e então rapidamente reembalado. Isso implica que o DNA não precisa ser dissociado ativamente do nucleossomo, mas que há uma fração significativa de tempo durante o qual ele está totalmente acessível. Na verdade, isso pode ser estendido à observação de que a introdução de uma sequência de ligação ao DNA dentro do nucleossomo aumenta a acessibilidade de regiões adjacentes do DNA quando ligado. Esta propensão para o DNA dentro do nucleossomo "respirar" tem consequências funcionais importantes para todas as proteínas de ligação ao DNA que operam em um ambiente de cromatina. Em particular, a respiração dinâmica de nucleossomos desempenha um papel importante na restrição do avanço da RNA polimerase II durante o alongamento da transcrição.
Região livre de nucleossomo
Os promotores de genes ativos têm regiões livres de nucleossomos (NFR). Isso permite a acessibilidade do DNA do promotor a várias proteínas, como fatores de transcrição. A região livre do nucleossomo normalmente se estende por 200 nucleotídeos em S. cerevisae Nucleossomos bem posicionados formam os limites de NFR. Esses nucleossomos são chamados de + 1-nucleossomo e −1-nucleossomo e estão localizados a distâncias canônicas a jusante e a montante, respectivamente, do local de início da transcrição. + 1-nucleossomo e vários nucleossomos a jusante também tendem a incorporar a variante da histona H2A.Z.
Modulando a estrutura do nucleossomo
Os genomas eucarióticos estão associados de forma ubíqua à cromatina; no entanto, as células devem regular espacial e temporalmente loci específicos, independentemente da cromatina em massa. A fim de alcançar o alto nível de controle necessário para coordenar os processos nucleares, como replicação, reparo e transcrição de DNA, as células desenvolveram uma variedade de meios para modular localmente e especificamente a estrutura e função da cromatina. Isso pode envolver a modificação covalente das histonas, a incorporação de variantes das histonas e a remodelação não covalente por enzimas de remodelação dependentes de ATP.
Modificações pós-tradução de histonas
Desde que foram descobertos em meados da década de 1960, as modificações nas histonas afetaram a transcrição. O fato de que a maioria das primeiras modificações pós-traducionais encontradas estavam concentradas nas extensões da cauda que se projetam do núcleo do nucleossomo levou a duas teorias principais a respeito do mecanismo de modificação das histonas. A primeira das teorias sugeriu que eles podem afetar as interações eletrostáticas entre as caudas das histonas e o DNA para "afrouxar" a estrutura da cromatina. Mais tarde, foi proposto que as combinações dessas modificações podem criar epítopos de ligação com os quais recrutar outras proteínas. Recentemente, dado que mais modificações foram encontradas nas regiões estruturadas das histonas, foi proposto que essas modificações podem afetar as interações histona-DNA e histona-histona dentro do núcleo do nucleossomo. Modificações (como acetilação ou fosforilação) que diminuem a carga do núcleo da histona globular são previstas para "afrouxar" a associação núcleo-DNA; a força do efeito depende da localização da modificação dentro do núcleo. Foi demonstrado que algumas modificações estão relacionadas ao silenciamento de genes; outros parecem estar correlacionados com a ativação do gene. As modificações comuns incluem acetilação , metilação ou ubiquitinação de lisina ; metilação de arginina ; e fosforilação de serina . A informação armazenada desta forma é considerada epigenética , uma vez que não é codificada no DNA, mas ainda é herdada de células-filhas. A manutenção de um status reprimido ou ativado de um gene é freqüentemente necessária para a diferenciação celular .
Variantes de histonas
Embora as histonas sejam notavelmente conservadas ao longo da evolução, várias formas variantes foram identificadas. Esta diversificação da função das histonas é restrita a H2A e H3, com H2B e H4 sendo principalmente invariantes. H2A pode ser substituído por H2AZ (que leva à estabilidade reduzida do nucleossomo) ou H2AX (que está associado ao reparo de DNA e diferenciação de células T ), enquanto os cromossomos X inativos em mamíferos são enriquecidos em macroH2A. H3 pode ser substituído por H3.3 (que se correlaciona com genes ativos e elementos reguladores) e nos centrômeros H3 é substituído por CENPA .
Remodelação de nucleossomos dependentes de ATP
Várias reações distintas estão associadas ao termo remodelação da cromatina dependente de ATP . Foi demonstrado que as enzimas de remodelação deslizam os nucleossomos ao longo do DNA, interrompem os contatos histona-DNA a ponto de desestabilizar o dímero H2A / H2B e geram torção super-helicoidal negativa no DNA e na cromatina. Recentemente, foi demonstrado que a enzima de remodelação Swr1 introduz a histona variante H2A.Z nos nucleossomos. No momento, não está claro se tudo isso representa reações distintas ou meramente resultados alternativos de um mecanismo comum. O que é compartilhado entre todos, e de fato a marca registrada da remodelação da cromatina dependente de ATP, é que todos eles resultam em acessibilidade alterada ao DNA.
Estudos observando a ativação do gene in vivo e, mais surpreendentemente, a remodelação in vitro revelaram que os eventos de remodelação da cromatina e a ligação do fator de transcrição são cíclicos e periódicos por natureza. Embora as consequências disso para o mecanismo de reação da remodelação da cromatina não sejam conhecidas, a natureza dinâmica do sistema pode permitir que ele responda mais rapidamente a estímulos externos. Um estudo recente indica que as posições dos nucleossomos mudam significativamente durante o desenvolvimento de células-tronco embrionárias de camundongo, e essas mudanças estão relacionadas à ligação de fatores de transcrição do desenvolvimento.
Remodelação de nucleossomos dinâmicos em todo o genoma de levedura
Estudos em 2007 catalogaram as posições dos nucleossomos em leveduras e mostraram que os nucleossomos estão esgotados nas regiões promotoras e nas origens de replicação . Cerca de 80% do genoma da levedura parece estar coberto por nucleossomos e o padrão de posicionamento dos nucleossomos está claramente relacionado às regiões do DNA que regulam a transcrição , regiões que são transcritas e regiões que iniciam a replicação do DNA. Mais recentemente, um novo estudo examinou mudanças dinâmicas no reposicionamento do nucleossomo durante um evento de reprogramação transcricional global para elucidar os efeitos no deslocamento do nucleossomo durante as mudanças transcricionais do genoma em leveduras ( Saccharomyces cerevisiae ). Os resultados sugeriram que os nucleossomos localizados nas regiões promotoras são deslocados em resposta ao estresse (como o choque térmico ). Além disso, a remoção de nucleossomos geralmente correspondia à ativação transcricional e a substituição de nucleossomos geralmente correspondia à repressão transcricional, presumivelmente porque os sítios de ligação do fator de transcrição tornaram-se mais ou menos acessíveis, respectivamente. Em geral, apenas um ou dois nucleossomos foram reposicionados no promotor para efetuar essas alterações transcricionais. No entanto, mesmo em regiões cromossômicas que não foram associadas a alterações transcricionais, foi observado reposicionamento de nucleossomos, sugerindo que a cobertura e a revelação do DNA transcricional não necessariamente produzem um evento transcricional. Após a transcrição, a região do rDNA deve ser protegida de qualquer dano, sugerindo que as proteínas HMGB desempenham um papel importante na proteção da região livre do nucleossomo.
Montagem do nucleossomo in vitro
Os nucleossomos podem ser montados in vitro usando histonas nativas ou recombinantes purificadas. Uma técnica padrão de carregar o DNA ao redor das histonas envolve o uso de diálise com sal . Uma reação que consiste nos octâmeros de histona e um molde de DNA nu podem ser incubados juntos em uma concentração de sal de 2 M. Ao diminuir continuamente a concentração de sal, o DNA se equilibrará para uma posição onde é enrolado em torno dos octâmeros de histona, formando nucleossomos. Em condições apropriadas, este processo de reconstituição permite que a afinidade de posicionamento do nucleossomo de uma dada sequência seja mapeada experimentalmente.
Partículas de núcleo de nucleossomo reticuladas por dissulfeto
Um avanço recente na produção de partículas de núcleo de nucleossomo com estabilidade aprimorada envolve reticulações de dissulfeto específicas do local . Duas ligações cruzadas diferentes podem ser introduzidas na partícula central do nucleossomo. Um primeiro reticula as duas cópias de H2A por meio de uma cisteína introduzida (N38C), resultando em octâmero de histona que é estável contra a perda de dímero H2A / H2B durante a reconstituição do nucleossomo. Uma segunda reticulação pode ser introduzida entre a cauda da histona H3 N-terminal e as extremidades do DNA do nucleossomo por meio de um nucleotídeo conversível incorporado. A reticulação de octâmero de DNA-histona estabiliza a partícula do núcleo do nucleossomo contra a dissociação do DNA em concentrações de partículas muito baixas e em concentrações elevadas de sal.
Montagem do nucleossomo in vivo
Os nucleossomos são a unidade básica de empacotamento do DNA construída a partir de proteínas histonas em torno das quais o DNA é enrolado. Eles servem como um arcabouço para a formação da estrutura da cromatina de ordem superior, bem como para uma camada de controle regulatório da expressão gênica. Os nucleossomos são rapidamente montados no DNA recém-sintetizado atrás da forquilha de replicação.
H3 e H4
As histonas H3 e H4 de nucleossomos antigos desmontados são mantidas nas proximidades e distribuídas aleatoriamente no DNA recém-sintetizado. Eles são montados pelo complexo fator-1 de montagem da cromatina (CAF-1), que consiste em três subunidades (p150, p60 e p48). H3 e H4 recentemente sintetizados são montados pelo fator de montagem de acoplamento de replicação (RCAF). O RCAF contém a subunidade Asf1, que se liga às proteínas H3 e H4 recentemente sintetizadas. As antigas proteínas H3 e H4 retêm suas modificações químicas que contribuem para a passagem da assinatura epigenética. As proteínas H3 e H4 recentemente sintetizadas são gradualmente acetiladas em diferentes resíduos de lisina como parte do processo de maturação da cromatina. Também se pensa que as antigas proteínas H3 e H4 nos novos nucleossomos recrutam enzimas modificadoras de histonas que marcam as novas histonas, contribuindo para a memória epigenética.
H2A e H2B
Em contraste com H3 e H4 antigos, as proteínas histonas H2A e H2B antigas são liberadas e degradadas; portanto, as proteínas H2A e H2B recém-montadas são incorporadas a novos nucleossomos. H2A e H2B são montados em dímeros que são carregados nos nucleossomos pela proteína-1 de montagem do nucleossomo (NAP-1), que também auxilia no deslizamento do nucleossomo. Os nucleossomos também são espaçados por complexos de remodelação de nucleossomos dependentes de ATP contendo enzimas como Isw1, Ino80 e Chd1, e subsequentemente montados em uma estrutura de ordem superior.
Galeria
A estrutura cristalina da partícula nuclear do nucleossomo ( PDB : 1EQZ ) - diferentes visualizações mostrando detalhes de dobramento e organização das histonas. As histonas H2A , H2B , H3 , H4 e DNA são coloridas.
Veja também
Referências
links externos
- MBInfo - O que são nucleossomos
- Nucleosomes no site do Richmond Lab
- Proteopedia Nucleosomes
- Nucleossomo no PDB
- Remodelação dinâmica de nucleossomos individuais em um genoma eucariótico em resposta à perturbação transcricional
- Dados de posicionamento de nucleossomos e ferramentas online (lista anotada, constantemente atualizada)
- Estrutura da proteína histona
- HistoneDB 2.0 - Banco de dados de histonas e variantes no NCBI