Mutação pontual - Point mutation
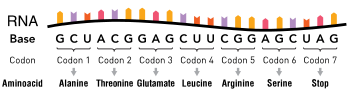
Uma mutação ou substituição pontual é uma mutação genética em que uma única base de nucleotídeo é alterada, inserida ou deletada de uma sequência de DNA ou RNA do genoma de um organismo. As mutações pontuais têm uma variedade de efeitos no produto da proteína downstream - consequências que são moderadamente previsíveis com base nas especificidades da mutação. Essas consequências podem variar de nenhum efeito (por exemplo, mutações sinônimas ) a efeitos deletérios (por exemplo, mutações de frameshift ), no que diz respeito à produção, composição e função de proteínas.
Causas
As mutações pontuais geralmente ocorrem durante a replicação do DNA . A replicação do DNA ocorre quando uma molécula de DNA de fita dupla cria duas fitas simples de DNA, cada uma das quais é um modelo para a criação da fita complementar. Uma única mutação pontual pode alterar toda a sequência de DNA. Mudar uma purina ou pirimidina pode mudar o aminoácido para o qual os nucleotídeos codificam.
As mutações pontuais podem surgir de mutações espontâneas que ocorrem durante a replicação do DNA . A taxa de mutação pode ser aumentada por agentes mutagênicos . Os mutagênicos podem ser físicos, como radiação de raios ultravioleta , raios-X ou calor extremo, ou químicos (moléculas que perdem os pares de bases ou interrompem a forma helicoidal do DNA). Os mutagênicos associados ao câncer são frequentemente estudados para aprender sobre o câncer e sua prevenção.
Existem várias maneiras de ocorrerem mutações pontuais. Primeiro, a luz ultravioleta (UV) e a luz de alta frequência são capazes de ionizar elétrons, o que por sua vez pode afetar o DNA. Moléculas reativas de oxigênio com radicais livres, que são um subproduto do metabolismo celular, também podem ser muito prejudiciais ao DNA. Esses reagentes podem levar a quebras de DNA de fita simples e quebras de DNA de fita dupla. Terceiro, as ligações no DNA eventualmente se degradam, o que cria outro problema para manter a integridade do DNA em um alto padrão. Também pode haver erros de replicação que levam a mutações de substituição, inserção ou exclusão.
Categorização
Categorização de transição / transversão

Em 1959, Ernst Freese cunhou os termos "transições" ou "transversões" para categorizar diferentes tipos de mutações pontuais. As transições são a substituição de uma base de purina por outra purina ou a substituição de uma pirimidina por outra pirimidina. As conversões são a substituição de uma purina por uma pirimidina ou vice-versa. Há uma diferença sistemática nas taxas de mutação para transições (Alfa) e transversões (Beta). Mutações de transição são cerca de dez vezes mais comuns do que transversões.
Categorização funcional
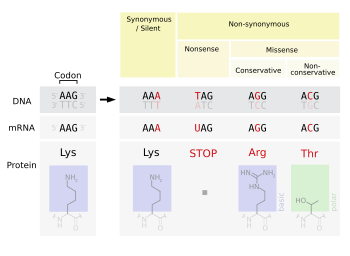
Mutações sem sentido incluem stop-gain e start-loss. O ganho de parada é uma mutação que resulta em um códon de terminação prematuro ( uma parada foi obtida ), que sinaliza o fim da tradução. Essa interrupção faz com que a proteína seja anormalmente encurtada. O número de aminoácidos perdidos medeia o impacto na funcionalidade da proteína e se ela funcionará de alguma forma. A perda de parada é uma mutação no códon de terminação original ( uma parada foi perdida ), resultando em extensão anormal do terminal carboxila de uma proteína. O ganho inicial cria um códon de início AUG a montante do local de início original. Se o novo AUG estiver próximo ao local de início original, no quadro do transcrito processado e a jusante para um local de ligação ribossômica, ele pode ser usado para iniciar a tradução. O efeito provável são aminoácidos adicionais adicionados ao terminal amino da proteína original. Mutações frame-shift também são possíveis em mutações de ganho inicial, mas normalmente não afetam a tradução da proteína original. A perda inicial é uma mutação pontual no códon inicial AUG de um transcrito, resultando na redução ou eliminação da produção de proteína.
Mutações missense codificam para um aminoácido diferente. Uma mutação sem sentido muda um códon para que uma proteína diferente seja criada, uma mudança não sinônima. As mutações conservadoras resultam em uma mudança de aminoácidos. No entanto, as propriedades do aminoácido permanecem as mesmas (por exemplo, hidrofóbico, hidrofílico, etc.). Às vezes, a alteração de um aminoácido da proteína não é prejudicial ao organismo como um todo. A maioria das proteínas pode resistir a uma ou duas mutações pontuais antes que suas funções mudem. Mutações não conservativas resultam em uma mudança de aminoácido que possui propriedades diferentes do tipo selvagem . A proteína pode perder sua função, o que pode resultar em uma doença no organismo. Por exemplo, a doença falciforme é causada por uma mutação de ponto único (uma mutação missense) no gene da beta- hemoglobina que converte um códon GAG em GUG, que codifica o aminoácido valina em vez do ácido glutâmico . A proteína também pode exibir um "ganho de função" ou tornar-se ativada, como é o caso da mutação que transforma uma valina em ácido glutâmico no gene BRAF ; isso leva a uma ativação da proteína RAF que causa sinalização proliferativa ilimitada nas células cancerosas. Ambos são exemplos de uma mutação não conservadora (missense).
Mutações silenciosas codificam para o mesmo aminoácido (uma " substituição sinônima "). Uma mutação silenciosa não afeta o funcionamento da proteína . Um único nucleotídeo pode mudar, mas o novo códon especifica o mesmo aminoácido, resultando em uma proteína não mutada. Esse tipo de mudança é chamado de mudança sinônima, uma vez que o código do códon antigo e do novo para o mesmo aminoácido. Isso é possível porque 64 códons especificam apenas 20 aminoácidos. No entanto, diferentes códons podem levar a níveis diferenciais de expressão de proteínas.
Inserções e exclusões de par de base único
Às vezes, o termo mutação de ponto é usado para descrever inserções ou deleções de um único par de bases (que tem mais um efeito adverso na proteína sintetizada devido aos nucleotídeos ainda serem lidos em tripletos, mas em quadros diferentes: uma mutação chamada frameshift mutação ).
Consequências gerais
As mutações pontuais que ocorrem em sequências não codificantes são, na maioria das vezes, sem consequências, embora haja exceções. Se o par de bases com mutação estiver na sequência do promotor de um gene, a expressão do gene pode mudar. Além disso, se a mutação ocorrer no local de splicing de um íntron , isso pode interferir no splicing correto do pré-mRNA transcrito .
Ao alterar apenas um aminoácido, todo o peptídeo pode mudar, mudando assim toda a proteína. A nova proteína é chamada de variante da proteína . Se a proteína original funciona na reprodução celular, essa mutação de ponto único pode alterar todo o processo de reprodução celular desse organismo.
Ponto de mutações germinativas pode levar a benéfico, bem como traços prejudiciais ou doenças. Isso leva a adaptações baseadas no ambiente onde o organismo vive. Uma mutação vantajosa pode criar uma vantagem para aquele organismo e fazer com que o traço seja passado de geração em geração, melhorando e beneficiando toda a população. A teoria científica da evolução é muito dependente de mutações pontuais nas células . A teoria explica a diversidade e a história dos organismos vivos na Terra. Em relação às mutações pontuais, afirma que as mutações benéficas permitem que o organismo se desenvolva e se reproduza, passando assim seus genes mutados positivamente afetados para a próxima geração. Por outro lado, mutações prejudiciais fazem com que o organismo morra ou tenha menos probabilidade de se reproduzir em um fenômeno conhecido como seleção natural .
Existem diferentes efeitos de curto e longo prazo que podem surgir de mutações. Os menores seriam uma interrupção do ciclo celular em vários pontos. Isso significa que um códon que codifica para o aminoácido glicina pode ser alterado para um códon de parada, fazendo com que as proteínas que deveriam ter sido produzidas sejam deformadas e incapazes de completar as tarefas pretendidas. Como as mutações podem afetar o DNA e, portanto, a cromatina , ela pode impedir a ocorrência de mitose devido à falta de um cromossomo completo. Problemas também podem surgir durante os processos de transcrição e replicação do DNA. Todos eles proíbem a reprodução da célula e, portanto, levam à morte da célula. Os efeitos de longo prazo podem ser uma mudança permanente de um cromossomo, o que pode levar a uma mutação. Essas mutações podem ser benéficas ou prejudiciais. O câncer é um exemplo de como eles podem ser prejudiciais.
Outros efeitos de mutações pontuais, ou polimorfismos de nucleotídeo único no DNA, dependem da localização da mutação dentro do gene. Por exemplo, se a mutação ocorrer na região do gene responsável pela codificação, a sequência de aminoácidos da proteína codificada pode ser alterada, causando uma alteração na função, localização da proteína, estabilidade da proteína ou complexo proteico. Muitos métodos foram propostos para prever os efeitos de mutações missense em proteínas. Os algoritmos de aprendizado de máquina treinam seus modelos para distinguir mutações neutras e associadas a doenças, enquanto outros métodos não treinam explicitamente seus modelos, mas quase todos os métodos exploram a conservação evolucionária, assumindo que as mudanças em posições conservadas tendem a ser mais deletérias. Embora a maioria dos métodos forneça uma classificação binária dos efeitos das mutações em prejudiciais e benignos, um novo nível de anotação é necessário para oferecer uma explicação de por que e como essas mutações danificam as proteínas.
Além disso, se a mutação ocorrer na região do gene onde a maquinaria de transcrição se liga à proteína, a mutação pode afetar a ligação dos fatores de transcrição porque as sequências de nucleotídeos curtas reconhecidas pelos fatores de transcrição serão alteradas. Mutações nessa região podem afetar a taxa de eficiência da transcrição gênica, que por sua vez pode alterar os níveis de mRNA e, portanto, os níveis de proteínas em geral.
As mutações pontuais podem ter vários efeitos no comportamento e na reprodução de uma proteína, dependendo de onde a mutação ocorre na sequência de aminoácidos da proteína. Se a mutação ocorrer na região do gene responsável pela codificação da proteína, o aminoácido pode ser alterado. Esta ligeira mudança na sequência de aminoácidos pode causar uma mudança na função, ativação da proteína significando como ela se liga a uma determinada enzima, onde a proteína estará localizada dentro da célula, ou a quantidade de energia livre armazenada dentro da proteína .
Se a mutação ocorrer na região do gene onde a maquinaria transcricional se liga à proteína, a mutação pode afetar a maneira como os fatores de transcrição se ligam à proteína. Os mecanismos de transcrição ligam-se a uma proteína por meio do reconhecimento de sequências curtas de nucleotídeos. Uma mutação nessa região pode alterar essas sequências e, assim, mudar a forma como os fatores de transcrição se ligam à proteína. Mutações nesta região podem afetar a eficiência da transcrição do gene, que controla tanto os níveis de mRNA quanto os níveis gerais de proteína.
Doenças específicas causadas por mutações pontuais
Câncer
Mutações pontuais em várias proteínas supressoras de tumor causam câncer . Por exemplo, mutações pontuais na polipose adenomatosa coli promovem a tumorigênese. Um novo ensaio, a proteólise paralela rápida (FASTpp) , pode ajudar a triagem rápida de defeitos de estabilidade específicos em pacientes com câncer individuais.
Neurofibromatose
A neurofibromatose é causada por mutações pontuais no gene Neurofibromina 1 ou Neurofibromina 2 .
Anemia falciforme
A anemia falciforme é causada por uma mutação pontual na cadeia β-globina da hemoglobina, fazendo com que o aminoácido hidrofílico ácido glutâmico seja substituído pelo aminoácido hidrofóbico valina na sexta posição.
O gene da β-globina é encontrado no braço curto do cromossomo 11. A associação de duas subunidades de α-globina de tipo selvagem com duas subunidades de β-globina mutantes forma a hemoglobina S (HbS). Em condições de baixo oxigênio (estando em alta altitude, por exemplo), a ausência de um aminoácido polar na posição seis da cadeia β-globina promove a polimerização não covalente (agregação) da hemoglobina, que distorce os glóbulos vermelhos em um forma de foice e diminui sua elasticidade.
A hemoglobina é uma proteína encontrada nas células vermelhas do sangue e é responsável pelo transporte de oxigênio pelo corpo. Existem duas subunidades que constituem a proteína da hemoglobina: beta-globinas e alfa-globinas . A beta-hemoglobina é criada a partir da informação genética do HBB, ou gene "hemoglobina beta" encontrado no cromossomo 11p15.5. Uma mutação de ponto único nesta cadeia polipeptídica, que tem 147 aminoácidos de comprimento, resulta na doença conhecida como anemia falciforme. A anemia falciforme é uma doença autossômica recessiva que afeta 1 em cada 500 afro-americanos e é uma das doenças do sangue mais comuns nos Estados Unidos. A única substituição do sexto aminoácido na beta-globina, o ácido glutâmico, por valina resulta em glóbulos vermelhos deformados. Essas células em forma de foice não podem transportar tanto oxigênio quanto os glóbulos vermelhos normais e são capturadas com mais facilidade pelos capilares, interrompendo o suprimento de sangue aos órgãos vitais. A alteração de um único nucleotídeo na beta-globina significa que mesmo o menor dos esforços por parte do portador resulta em dor intensa e até ataque cardíaco. Abaixo está um gráfico que descreve os primeiros treze aminoácidos na cadeia polipeptídica da célula falciforme normal e anormal .
| AGO | GUG | CAC | CUG | ACU | CCU | G A G | MORDAÇA | AAG | UCU | GCC | GUU | ACU |
| COMEÇAR | Val | Seu | Leu | Thr | Pró | Glu | Glu | Lys | Ser | Ala | Val | Thr |
| AGO | GUG | CAC | CUG | ACU | CCU | G U G | MORDAÇA | AAG | UCU | GCC | GUU | ACU |
| COMEÇAR | Val | Seu | Leu | Thr | Pró | Val | Glu | Lys | Ser | Ala | Val | Thr |
Doença de Tay-Sachs
A causa da doença de Tay-Sachs é um defeito genético transmitido de pais para filhos. Esse defeito genético está localizado no gene HEXA, que é encontrado no cromossomo 15.
O gene HEXA faz parte de uma enzima chamada beta-hexosaminidase A, que desempenha um papel crítico no sistema nervoso. Esta enzima ajuda a quebrar uma substância gordurosa chamada gangliosídeo GM2 nas células nervosas. Mutações no gene HEXA interrompem a atividade da beta-hexosaminidase A, evitando a quebra das substâncias gordurosas. Como resultado, as substâncias gordurosas se acumulam em níveis fatais no cérebro e na medula espinhal. O acúmulo de gangliosídeo GM2 causa danos progressivos às células nervosas. Essa é a causa dos sinais e sintomas da doença de Tay-Sachs.
Daltonismo
Pessoas daltônicas têm mutações em seus genes que causam a perda de cones vermelhos ou verdes e, portanto, têm dificuldade em distinguir entre as cores. Existem três tipos de cones no olho humano: vermelho, verde e azul. Agora, os pesquisadores descobriram que algumas pessoas com a mutação do gene que causa o daltonismo perdem todo um conjunto de cones "coloridos" sem alterar a clareza de sua visão em geral.
Mutação pontual induzida por repetição
Em biologia molecular , a mutação pontual induzida por repetição ou RIP é um processo pelo qual o DNA acumula mutações de transição G : C para A : T. A evidência genômica indica que o RIP ocorre ou ocorreu em uma variedade de fungos, enquanto a evidência experimental indica que o RIP é ativo em Neurospora crassa , Podospora anserina , Magnaporthe grisea , Leptosphaeria maculans , Gibberella zeae e Nectria haematococca . Em Neurospora crassa , as sequências mutadas por RIP são frequentemente metiladas de novo .
RIP ocorre durante o estágio sexual em núcleos haplóides após a fertilização, mas antes da replicação do DNA meiótico . No Neurospora crassa , as sequências repetidas de pelo menos 400 pares de bases de comprimento são vulneráveis ao RIP. Repetições com apenas 80% de identidade de nucleotídeos também podem estar sujeitas a RIP. Embora o mecanismo exato de reconhecimento de repetição e mutagênese sejam mal compreendidos, o RIP resulta em sequências repetidas passando por múltiplas mutações de transição .
As mutações RIP não parecem estar limitadas a sequências repetidas. De fato, por exemplo, no fungo fitopatogênico L. maculans , as mutações RIP são encontradas em regiões de cópia única, adjacentes aos elementos repetidos. Essas regiões são regiões não codificantes ou genes que codificam pequenas proteínas segregadas, incluindo genes de avirulência. O grau de RIP dentro dessas regiões de cópia única foi proporcional à sua proximidade a elementos repetitivos.
Rep e Kistler especularam que a presença de regiões altamente repetitivas contendo transposons pode promover a mutação de genes efetores residentes. Portanto, sugere-se que a presença de genes efetores nessas regiões promova sua adaptação e diversificação quando expostos a forte pressão de seleção.
Como a mutação RIP é tradicionalmente observada como restrita a regiões repetitivas e não a regiões de cópia única, Fudal et al. sugeriram que o vazamento da mutação RIP pode ocorrer dentro de uma distância relativamente curta de uma repetição afetada por RIP. Na verdade, isso foi relatado em N. crassa, em que o vazamento de RIP foi detectado em sequências de cópia única a pelo menos 930 pb da fronteira de sequências duplicadas vizinhas. Elucidar o mecanismo de detecção de sequências repetidas que levam ao RIP pode permitir entender como as sequências de flanqueamento também podem ser afetadas.
Mecanismo
O RIP causa mutações de transição de G : C para A : T nas repetições; no entanto, o mecanismo que detecta as sequências repetidas é desconhecido. RID é a única proteína conhecida essencial para RIP. É uma proteína semelhante à metiltransferease de DNA, que quando sofre mutação ou nocaute resulta em perda de RIP. A exclusão do homólogo de rid em Aspergillus nidulans , dmtA , resulta em perda de fertilidade, enquanto a exclusão do homólogo de rid em Ascobolus immersens , masc1 , resulta em defeitos de fertilidade e perda de metilação induzida por pré-biota (MIP) .
Consequências
Acredita-se que o RIP tenha evoluído como um mecanismo de defesa contra elementos transponíveis , que se assemelham a parasitas invadindo e se multiplicando dentro do genoma. O RIP cria múltiplas mutações sem sentido e sem sentido na sequência de codificação. Essa hipermutação de GC para AT em sequências repetitivas elimina produtos de genes funcionais da sequência (se é que havia algum). Além disso, muitos dos nucleotídeos portadores de C tornam-se metilados , diminuindo assim a transcrição.
Uso em biologia molecular
Como o RIP é tão eficiente na detecção e na mutação de repetições, os biólogos fúngicos costumam usá-lo como uma ferramenta para mutagênese . Uma segunda cópia de um gene de cópia única é primeiro transformada no genoma . O fungo deve então acasalar e passar por seu ciclo sexual para ativar a máquina RIP. Muitas mutações diferentes dentro do gene duplicado são obtidas até mesmo de um único evento de fertilização, de modo que alelos inativados, geralmente devido a mutações sem sentido , bem como alelos contendo mutações sem sentido, podem ser obtidos.
História
O processo de reprodução celular da meiose foi descoberto por Oscar Hertwig em 1876. A mitose foi descoberta vários anos depois, em 1882, por Walther Flemming .
Hertwig estudou ouriços-do-mar e notou que cada ovo continha um núcleo antes da fertilização e dois núcleos depois. Esta descoberta provou que um espermatozóide pode fertilizar um óvulo e, portanto, provou o processo de meiose. Hermann Fol continuou a pesquisa de Hertwig testando os efeitos da injeção de vários espermatozóides em um óvulo e descobriu que o processo não funcionava com mais de um espermatozóide.
Flemming começou sua pesquisa sobre a divisão celular em 1868. O estudo das células era um tópico cada vez mais popular nessa época. Em 1873, Schneider já havia começado a descrever as etapas da divisão celular. Flemming ampliou essa descrição em 1874 e 1875, ao explicar as etapas com mais detalhes. Ele também argumentou com as descobertas de Schneider de que o núcleo se separava em estruturas semelhantes a bastonetes, sugerindo que o núcleo na verdade se separava em fios que, por sua vez, se separavam. Flemming concluiu que as células se replicam por meio da divisão celular, para ser uma mitose mais específica.
Matthew Meselson e Franklin Stahl são creditados com a descoberta da replicação do DNA . Watson e Crick reconheceram que a estrutura do DNA indica que existe alguma forma de processo de replicação. No entanto, não havia muitas pesquisas feitas sobre esse aspecto do DNA até depois de Watson e Crick. As pessoas consideraram todos os métodos possíveis para determinar o processo de replicação do DNA, mas nenhum teve sucesso até Meselson e Stahl. Meselson e Stahl introduziram um isótopo pesado em algum DNA e traçaram sua distribuição. Por meio desse experimento, Meselson e Stahl foram capazes de provar que o DNA se reproduz de forma semi-conservadora.
Veja também
Referências
links externos
- Point + Mutation nos cabeçalhos de assuntos médicos da Biblioteca Nacional de Medicina dos EUA (MeSH)