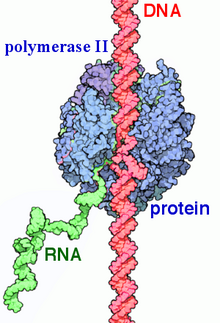
RNA polimerase II - RNA polymerase II
A RNA polimerase II ( RNAP II e Pol II ) é um complexo multiproteico que transcreve o DNA em precursores do RNA mensageiro (mRNA) e da maioria dos pequenos RNAs nucleares (snRNA) e microRNA . É uma das três enzimas RNAP encontradas no núcleo das células eucarióticas . Um complexo de 12 subunidades de 550 kDa , RNAP II é o tipo de RNA polimerase mais estudado . Uma ampla gama de fatores de transcrição é necessária para que ele se ligue aos promotores do gene a montante e comece a transcrição.
Descoberta

Os primeiros estudos sugeriram um mínimo de dois RNAPs: um que sintetizou rRNA no nucléolo e outro que sintetizou outro RNA no nucleoplasma , parte do núcleo, mas fora do nucléolo. Em 1969, os experimentalistas científicos Robert Roeder e William Rutter descobriram definitivamente um RNAP adicional que era responsável pela transcrição de algum tipo de RNA no nucleoplasma. A descoberta foi obtida pelo uso de cromatografia de troca iônica via pérolas Sephadex revestidas com DEAE . A técnica separou as enzimas pela ordem das eluições correspondentes, Ι, ΙΙ, ΙΙΙ, aumentando a concentração de sulfato de amônio. As enzimas foram nomeadas de acordo com a ordem das eluições, RNAP I , RNAP II, RNAP IΙI . Essa descoberta demonstrou que havia uma enzima adicional presente no nucleoplasma, o que permitiu a diferenciação entre RNAP II e RNAP III.
Subunidades

O núcleo eucariótico de RNA polimerase II foi primeiro purificado usando ensaios de transcrição. A enzima purificada tem tipicamente 10-12 subunidades (12 em humanos e leveduras) e é incapaz de reconhecimento de promotor específico. Muitas interações subunidade-subunidade são conhecidas.
- Subunidade RPB1 de RNA polimerase II dirigida por DNA - uma enzima que em humanos é codificada pelo gene POLR2A e em leveduras é codificada por RPO21. RPB1 é a maior subunidade da RNA polimerase II. Ele contém um domínio carboxi terminal (CTD) composto de até 52 repetições de heptapeptídeo (YSPTSPS) que são essenciais para a atividade da polimerase. O CTD foi descoberto pela primeira vez no laboratório de CJ Ingles na Universidade de Toronto e por JL Corden na Universidade Johns Hopkins . Em combinação com várias outras subunidades de polimerase, a subunidade RPB1 forma o domínio de ligação ao DNA da polimerase, uma fenda na qual o molde de DNA é transcrito em RNA. Ele interage fortemente com RPB8.
- RPB2 ( POLR2B ) - a segunda maior subunidade que em combinação com pelo menos duas outras subunidades de polimerase forma uma estrutura dentro da polimerase que mantém contato no sítio ativo da enzima entre o molde de DNA e o RNA recém-sintetizado.
- RPB3 ( POLR2C ) - a terceira maior subunidade. Existe como um heterodímero com outra subunidade de polimerase, POLR2J formando um subconjunto de núcleo. RPB3 interage fortemente com RPB1-5, 7, 10-12.
- A subunidade B4 da RNA polimerase II (RPB4) - codificada pelo gene POLR2Dé a quarta maior subunidade e pode ter um papel protetor de estresse.
- RPB5 - Em humanos é codificado pelo gene POLR2E . Duas moléculas desta subunidade estão presentes em cada RNA polimerase II. RPB5 interage fortemente com RPB1, RPB3 e RPB6.
- RPB6 ( POLR2F ) - forma uma estrutura com pelo menos duas outras subunidades que estabiliza a polimerase de transcrição no molde de DNA.
- RPB7 - codificado por POLR2G e pode desempenhar um papel na regulação da função da polimerase. RPB7 interage fortemente com RPB1 e RPB5.
- RPB8 ( POLR2H ) - interage com as subunidades RPB1-3, 5 e 7.
- RPB9 - O sulco no qual o molde de DNA é transcrito em RNA é composto por RPB9 ( POLR2I ) e RPB1.
- RPB10 - o produto do gene POLR2L . Ele interage com RPB1-3 e 5, e fortemente com RPB3.
- RPB11 - a subunidade RPB11 é ela própria composta de três subunidades em humanos: POLR2J (RPB11-a), POLR2J2 (RPB11-b) e POLR2J3 (RPB11-c).
- RPB12 - Também interage com RPB3 é RPB12 ( POLR2K ).
conjunto
RPB3 está envolvido na montagem de RNA polimerase II. Um subcomplex de RPB2 e RPB3 aparece logo após a síntese da subunidade. Este complexo posteriormente interage com RPB1. RPB3, RPB5 e RPB7 interagem entre si para formar homodímeros, e RPB3 e RPB5 juntos são capazes de contatar todas as outras subunidades RPB, exceto RPB9. Apenas RPB1 se liga fortemente a RPB5. A subunidade RPB1 também contata RPB7, RPB10 e mais fracamente, mas de forma mais eficiente com RPB8. Uma vez que RPB1 entra no complexo, outras subunidades como RPB5 e RPB7 podem entrar, onde RPB5 se liga a RPB6 e RPB8 e RPB3 traz RPB10, RPB 11 e RPB12. RPB4 e RPB9 podem entrar assim que a maior parte do complexo estiver montada. RPB4 forma um complexo com RPB7.
Cinética
As enzimas podem catalisar até vários milhões de reações por segundo. As taxas de enzimas dependem das condições da solução e da concentração de substrato. Como outras enzimas, POLR2 tem uma curva de saturação e uma velocidade máxima ( V max ). Tem um K m (concentração de substrato necessária para metade do V max ) e um k cat (o número de moléculas de substrato tratadas por um sítio ativo por segundo). A constante de especificidade é dada por k cat / K m . O máximo teórico para a constante de especificidade é o limite de difusão de cerca de 10 8 a 10 9 ( M −1 s −1 ), onde cada colisão da enzima com seu substrato resulta em catálise. Na levedura, a mutação no domínio Trigger-Loop da maior subunidade pode alterar a cinética da enzima.
A RNA polimerase bacteriana, um parente da RNA polimerase II, alterna entre os estados inativado e ativado por translocação para frente e para trás ao longo do DNA. As concentrações de [NTP] eq = 10 μM GTP, 10 μM UTP, 5 μM ATP e 2,5 μM CTP, produzem uma taxa média de alongamento, número de turnover, de ~ 1 bp (NTP) -1 para RNAP bacteriana, um parente da RNA polimerase II.
A RNA polimerase II sofre extensa pausa co-transcricional durante o alongamento da transcrição. Essa pausa é especialmente pronunciada nos nucleossomos e surge em parte pela entrada da polimerase em um estado de retrocesso transcricionalmente incompetente. A duração dessas pausas varia de segundos a minutos ou mais, e a saída de pausas de longa duração pode ser promovida por fatores de alongamento, como TFIIS. Por sua vez, a taxa de transcrição influencia se as histonas dos nucleossomos transcritos são removidas da cromatina ou reinseridas atrás da polimerase transcrita.
Alfa-Amanitina
A RNA polimerase II é inibida pela α-Amanitina e outras amatoxinas . α-Amanitina é uma substância altamente venenosa encontrada em muitos cogumelos. O veneno de cogumelo tem efeitos diferentes em cada uma das RNA polimerases: I, II, III. RNAP I é completamente indiferente à substância e funcionará normalmente, enquanto RNAP III tem uma sensibilidade moderada. A RNAP II, entretanto, é completamente inibida pela toxina. Alfa-Amanitina inibe RNAP II por fortes interações no "funil" da enzima, "fenda" e as regiões chave da "ponte α-hélice " da subunidade RPB-1.
Holoenzima
A holoenzima RNA polimerase II é uma forma de RNA polimerase II eucariótica que é recrutada para os promotores de genes que codificam proteínas em células vivas. Consiste em RNA polimerase II, um subconjunto de fatores gerais de transcrição e proteínas regulatórias conhecidas como proteínas SRB.
Parte da montagem da holoenzima é chamada de complexo de pré - iniciação , porque sua montagem ocorre no promotor do gene antes do início da transcrição . O complexo mediador atua como uma ponte entre a RNA polimerase II e os fatores de transcrição.
Controle pela estrutura da cromatina
Este é um esboço de um exemplo de mecanismo de células de levedura pelo qual a estrutura da cromatina e a modificação pós-tradução da histona ajudam a regular e registrar a transcrição de genes pela RNA polimerase II.
Este caminho dá exemplos de regulação nestes pontos de transcrição:
- Pré-iniciação (promoção por Bre1, modificação de histona)
- Iniciação (promoção por TFIIH, modificação de Pol II E promoção por COMPASS, modificação de histona)
- Alongamento (promoção por Set2, modificação de histona)
Isso se refere a vários estágios do processo como etapas regulatórias. Não foi provado que sejam usados para regulamentação, mas é muito provável que sejam.
Os promotores de alongamento de RNA Pol II podem ser resumidos em 3 classes.
- Fatores afetados por parada dependentes de drogas / sequência (várias proteínas interferentes)
- Fatores orientados para a estrutura da cromatina (modificadores pós-transcricionais de histona, por exemplo, histona metiltransferases)
- Fatores de melhoria da catálise de RNA Pol II (várias proteínas interferentes e cofatores de Pol II; ver RNA polimerase II).
Mecanismos de transcrição
-
Fatores orientados para a estrutura da cromatina:
(HMTs ( H istone M etil T ransferases)):
COMPASS§ † - ( COM plex de P roteínas AS associado a S et1) - Metila lisina 4 da histona H3: É responsável pela repressão / silenciamento da transcrição. Uma parte normal do crescimento celular e da regulação da transcrição dentro da RNAP II. - Set2 - Metila lisina 36 da histona H3: Set2 está envolvido na regulação do alongamento da transcrição por meio de seu contato direto com o CTD.
(exemplo irrelevante interessante: Dot1 * ‡ - Metila lisina 79 da histona H3.) - Bre1 - Ubiquina (adiciona ubiquitina a) lisina 123 de histona H2B. Associado à pré-iniciação e permitindo a ligação do RNA Pol II.
CTD de RNA polimerase
O terminal C de RPB1 é anexado para formar o domínio do terminal C (CTD). O domínio carboxi-terminal da RNA polimerase II consiste tipicamente em até 52 repetições da sequência Tyr-Ser-Pro-Thr-Ser-Pro-Ser. O domínio se estende desde o núcleo da enzima RNAPII até o canal de saída, este posicionamento é eficaz devido às suas induções de "reações de processamento de RNA, por meio de interações diretas ou indiretas com componentes da maquinaria de processamento de RNA". O domínio CTD não existe na RNA Polimerase I ou RNA Polimerase III. A RNA polimerase CTD foi descoberta primeiro no laboratório de CJIngles na Universidade de Toronto e também no laboratório de J Corden na Universidade Johns Hopkins durante os processos de sequenciamento do DNA que codifica a subunidade RPB1 da RNA polimerase de leveduras e camundongos, respectivamente. Outras proteínas freqüentemente se ligam ao domínio C-terminal da RNA polimerase para ativar a atividade da polimerase. É o domínio da proteína que está envolvido na iniciação da transcrição, no capeamento do transcrito do RNA e na ligação ao spliceossomo para o splicing do RNA .
Fosforilação do domínio CTD
A RNA polimerase II existe em duas formas não fosforilada e fosforilada, IIA e IIO, respectivamente. A transição entre as duas formas facilita funções diferentes para a transcrição. A fosforilação de CTD é catalisada por um dos seis fatores gerais de transcrição , TFIIH . O TFIIH tem dois propósitos: um é desenrolar o DNA no local de início da transcrição e o outro é fosforilar. A forma polimerase IIA junta-se ao complexo de pré-iniciação, isto é sugerido porque IIA se liga com maior afinidade ao TBP ( TATA-box binding protein ), a subunidade do fator de transcrição geral TFIID , do que a forma polimerase IIO. A forma polimerase IIO facilita o alongamento da cadeia de RNA. O método de iniciação do alongamento é feito pela fosforilação da Serina na posição 5 (Ser5), via TFIIH. O Ser5 recém fosforilado recruta enzimas para cobrir a extremidade 5 'do RNA recém-sintetizado e os "fatores de processamento 3' para os sítios poli (A) ". Uma vez que a segunda Serina é fosforilada, Ser2, o alongamento é ativado. Para terminar o alongamento, a desfosforilação deve ocorrer. Uma vez que o domínio é completamente desfosforilado, a enzima RNAP II é "reciclada" e catalisa o mesmo processo com outro local de iniciação.
Reparo recombinacional acoplado à transcrição
O dano oxidativo ao DNA pode bloquear a transcrição da RNA polimerase II e causar quebras de fita. Foi descrito um processo de recombinação associado à transcrição modelado por RNA que pode proteger contra danos ao DNA. Durante os estágios G1 / G0 do ciclo celular , as células exibem montagem de fatores de recombinação homóloga em quebras de fita dupla em regiões ativamente transcritas. Parece que a transcrição é acoplada ao reparo de quebras de fita dupla de DNA por recombinação homóloga modelada por RNA. Este processo de reparo reúne de forma eficiente e precisa quebras de fita dupla em genes sendo ativamente transcritos pela RNA polimerase II.
Veja também
- Transcrição eucariótica
- Modificação pós-transcricional
- RNA polimerase I
- Holoenzima de RNA polimerase II
- RNA polimerase III
- Transcrição (genética)
Referências
links externos
( Cópia do Wayback Machine )
- RNA + Polymerase + II na Biblioteca Nacional de Medicina dos Estados Unidos. Cabeçalhos de Assuntos Médicos (MeSH)