Ubiquitina - Ubiquitin
| Família Ubiquitina | |||||||||
|---|---|---|---|---|---|---|---|---|---|
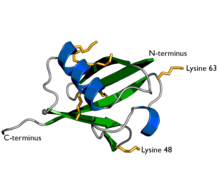
Um diagrama de ubiquitina . As sete cadeias laterais de lisina são mostradas em amarelo / laranja.
| |||||||||
| Identificadores | |||||||||
| Símbolo | ubiquitina | ||||||||
| Pfam | PF00240 | ||||||||
| InterPro | IPR000626 | ||||||||
| PRÓSITO | PDOC00271 | ||||||||
| SCOP2 | 1aar / SCOPe / SUPFAM | ||||||||
| |||||||||
Ubiquitina é uma pequena proteína reguladora (8,6 kDa ) encontrada na maioria dos tecidos de organismos eucarióticos , ou seja, é encontrada de forma ubíqua . Foi descoberto em 1975 por Gideon Goldstein e caracterizado ao longo do final dos anos 1970 e 1980. Quatro genes no genoma humano codificam para a ubiquitina: UBB , UBC , UBA52 e RPS27A .
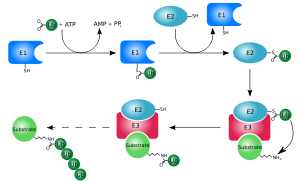
A adição de ubiquitina ao substrato uma proteína é chamado de ubiquitinação (ou, alternativamente, ubiquitinação ou ubiquitinação ). A ubiquitilação afeta as proteínas de várias maneiras: pode marcá-las para degradação por meio do proteassoma , alterar sua localização celular , afetar sua atividade e promover ou prevenir interações de proteínas . Ubiquitilação envolve três etapas principais: ativação, conjugação e ligação, realizada por enzimas de ativação de ubiquitina (E1s), enzimas de conjugação de ubiquitina (E2s) e ligases de ubiquitina (E3s), respectivamente. O resultado desta cascata sequencial é ligar a ubiquitina aos resíduos de lisina no substrato de proteína por meio de uma ligação isopeptídica , resíduos de cisteína por meio de uma ligação tioéster , resíduos de serina e treonina por meio de uma ligação éster ou o grupo amino do terminal N da proteína por meio de um ligação peptídica .
As modificações da proteína podem ser uma única proteína ubiquitina (monoubiquitilação) ou uma cadeia de ubiquitina (poliubiquitilação). As moléculas secundárias de ubiquitina estão sempre ligadas a um dos sete resíduos de lisina ou à metionina N-terminal da molécula de ubiquitina anterior. Estes resíduos de 'ligação' são representados por um "K" ou "M" (a notação de aminoácidos de uma letra de lisina e metionina, respectivamente) e um número, referindo-se à sua posição na molécula de ubiquitina como em K48, K29 ou M1 . A primeira molécula de ubiquitina é covalentemente ligada através de seu grupo carboxilato C-terminal a uma lisina, cisteína, serina, treonina ou terminal N particular da proteína alvo. A polubiquitilação ocorre quando o terminal C de outra ubiquitina está ligado a um dos sete resíduos de lisina ou à primeira metionina na molécula de ubiquitina previamente adicionada, criando uma cadeia. Esse processo se repete várias vezes, levando à adição de várias ubiquitinas. Apenas a poliubiquitilação em lisinas definidas, principalmente em K48 e K29, está relacionada à degradação pelo proteassoma (referido como o "beijo da morte molecular"), enquanto outras poliubiquitilações (por exemplo, em K63, K11, K6 e M1) e monoubiquitilações podem regular processos como tráfego endocítico , inflamação , tradução e reparo de DNA .
A descoberta de que as cadeias de ubiquitina direcionam proteínas para o proteassoma, que degrada e recicla proteínas, foi homenageado com o Prêmio Nobel de Química em 2004.
Identificação
Ubiquitina (originalmente, polipeptídeo imunopoiético ubíquo ) foi identificada pela primeira vez em 1975 como uma proteína de 8,6 kDa expressa em todas as células eucarióticas . As funções básicas da ubiquitina e os componentes do caminho da ubiquitina foram elucidados no início dos anos 1980 no Technion por Aaron Ciechanover , Avram Hershko e Irwin Rose, pelo qual o Prêmio Nobel de Química foi concedido em 2004.
O sistema de ubiquitilação foi inicialmente caracterizado como um sistema proteolítico dependente de ATP presente em extratos celulares. Um polipeptídeo estável ao calor presente nestes extratos, fator de proteólise dependente de ATP 1 (APF-1), foi encontrado para se tornar covalentemente ligado ao substrato de proteína modelo lisozima em um processo dependente de ATP e Mg 2+ . Múltiplas moléculas de APF-1 foram ligadas a uma única molécula de substrato por uma ligação isopeptídica e os conjugados foram degradados rapidamente com a liberação de APF-1 livre. Logo após a conjugação da proteína APF-1 ter sido caracterizada, o APF-1 foi identificado como ubiquitina. O grupo carboxila do resíduo de glicina C-terminal da ubiquitina (Gly76) foi identificado como a porção conjugada aos resíduos de lisina do substrato .
A proteína
| Número de resíduos | 76 |
|---|---|
| Massa molecular | 8564,8448 Da |
| Ponto isoelétrico (pI) | 6,79 |
| Nomes de genes | RPS27A (UBA80, UBCEP1), UBA52 (UBCEP2), UBB , UBC |
| Sequência ( letra única ) |
MQIFV K TLTG K TITLEVEPSDTIENV K A K IQD K EGIPPD
QQRLIFAG K QLEDGRTLSDYNIQ K ESTLHLVLRLRGG |
Ubiquitina é uma pequena proteína que existe em todas as células eucarióticas . Ele executa suas inúmeras funções por meio da conjugação de uma grande variedade de proteínas-alvo. Uma variedade de modificações diferentes pode ocorrer. A própria proteína ubiquitina consiste em 76 aminoácidos e tem uma massa molecular de cerca de 8,6 kDa. As principais características incluem sua cauda C-terminal e os 7 resíduos de lisina . É altamente conservado ao longo da evolução dos eucariotos; ubiquitina humana e de levedura compartilham 96% de identidade de sequência .
Genes
Ubiquitina é codificada em mamíferos por 4 genes diferentes. Os genes UBA52 e RPS27A codificam uma única cópia da ubiquitina fundida às proteínas ribossomais L40 e S27a, respectivamente. Os genes UBB e UBC codificam para proteínas precursoras de poliubiquitina.
Ubiquitilação
Ubiquitilação (também conhecida como ubiquitinação ou ubiquitinilação) é uma modificação enzimática pós-tradução na qual uma proteína ubiquitina é ligada a uma proteína substrato . Esse processo liga mais comumente o último aminoácido da ubiquitina ( glicina 76) a um resíduo de lisina no substrato. Uma ligação isopeptídica é formada entre o grupo carboxila (COO - ) da glicina da ubiquitina e o grupo épsilon- amino (ε- NH+
3) da lisina do substrato. A clivagem por tripsina de um substrato conjugado com ubiquitina deixa um "remanescente" de di-glicina que é usado para identificar o local da ubiquitilação. Ubiquitina também pode ser ligada a outros locais em uma proteína que são nucleófilos ricos em elétrons , denominado " ubiquitilação não canônica". Isso foi observado pela primeira vez com o grupo amina do N-terminal de uma proteína sendo usado para ubiquitilação, em vez de um resíduo de lisina, na proteína MyoD e foi observado desde 22 outras proteínas em várias espécies, incluindo a própria ubiquitina. Também há evidências crescentes de resíduos não lisina como alvos de ubiquitilação usando grupos não amina, como o grupo sulfidrila na cisteína e o grupo hidroxila na treonina e serina. O resultado final desse processo é a adição de uma molécula de ubiquitina (monoubiquitilação) ou uma cadeia de moléculas de ubiquitina (poliubiquitinação) à proteína substrato.
A ubiquitina requer três tipos de enzimas: enzimas de ativação de ubiquitina , enzimas de conjugação de ubiquitina e ligases de ubiquitina , conhecidas como E1s, E2s e E3s, respectivamente. O processo consiste em três etapas principais:
- Ativação: a ubiquitina é ativada em uma reação de duas etapas por uma enzima E1 de ativação da ubiquitina , que é dependente de ATP . A etapa inicial envolve a produção de um intermediário ubiquitina-adenilato. O E1 se liga tanto ao ATP quanto à ubiquitina e catalisa a acil-adenilação do terminal C da molécula de ubiquitina. A segunda etapa transfere a ubiquitina para um resíduo de cisteína do sítio ativo , com liberação de AMP . Esta etapa resulta em uma ligação tioéster entre o grupo carboxila C-terminal da ubiquitina e o grupo sulfidrila cisteína E1 . O genoma humano contém dois genes que produzem enzimas capazes de ativar a ubiquitina: UBA1 e UBA6 .
- Conjugação: as enzimas de conjugação de ubiquitina E2 catalisam a transferência de ubiquitina de E1 para o sítio ativo de cisteína de E2 por meio de uma reação de esterificação trans (tio). Para realizar essa reação, o E2 se liga à ubiquitina ativada e à enzima E1. Os humanos possuem 35 enzimas E2 diferentes, enquanto outros organismos eucarióticos têm entre 16 e 35. Eles são caracterizados por sua estrutura altamente conservada, conhecida como dobra catalítica de conjugação de ubiquitina (UBC).
- Ligadura: as ligases de ubiquitina E3 catalisam a etapa final da cascata de ubiquitinação. Mais comumente, eles criam uma ligação isopeptídica entre uma lisina da proteína alvo e a glicina C-terminal da ubiquitina. Em geral, esta etapa requer a atividade de uma das centenas de E3s. As enzimas E3 funcionam como módulos de reconhecimento de substrato do sistema e são capazes de interagir com E2 e substrato . Algumas enzimas E3 também ativam as enzimas E2. As enzimas E3 possuem um de dois domínios : o homólogo ao domínio do terminal carboxila E6-AP ( HECT ) e o domínio do gene novo realmente interessante ( RING ) (ou o domínio U-box intimamente relacionado). O domínio HECT E3s se liga transitoriamente à ubiquitina neste processo (um intermediário tioéster obrigatório é formado com a cisteína do sítio ativo de E3), enquanto o domínio RING E3s catalisa a transferência direta da enzima E2 para o substrato. O complexo promotor de anáfase (APC) e o complexo SCF (para o complexo de proteína Skp1-Cullin-F-box) são dois exemplos de E3s com múltiplas subunidades envolvidas no reconhecimento e ubiquitinação de proteínas alvo específicas para degradação pelo proteassoma .
Na cascata de ubiquitinação, E1 pode se ligar a muitos E2s, que podem se ligar a centenas de E3s de forma hierárquica. Ter níveis dentro da cascata permite uma regulamentação rígida do maquinário de ubiquitinação. Outras proteínas semelhantes à ubiquitina (UBLs) também são modificadas por meio da cascata E1-E2-E3, embora existam variações nesses sistemas.
As enzimas E4, ou fatores de alongamento da cadeia da ubiquitina, são capazes de adicionar cadeias de poliubiquitina pré-formadas às proteínas do substrato. Por exemplo, a monoubiquitilação múltipla do supressor de tumor p53 por Mdm2 pode ser seguida pela adição de uma cadeia de poliubiquitina usando p300 e CBP .
Tipos
A ubiquitinação afeta o processo celular regulando a degradação de proteínas (via proteassoma e lisossoma ), coordenando a localização celular de proteínas, ativando e inativando proteínas e modulando as interações proteína-proteína . Esses efeitos são mediados por diferentes tipos de ubiquitinação de substrato, por exemplo, a adição de uma única molécula de ubiquitina (monoubiquitinação) ou diferentes tipos de cadeias de ubiquitina (poliubiquitinação).
Monoubiquitinação
Monoubiquitinação é a adição de uma molécula de ubiquitina a um resíduo de proteína substrato. A multi-monoubiquitinação é a adição de uma molécula de ubiquitina a vários resíduos de substrato. A monoubiquitinação de uma proteína pode ter efeitos diferentes para a poliubiquitinação da mesma proteína. A adição de uma única molécula de ubiquitina é considerada necessária antes da formação das cadeias de poliubiquitina. A monoubiquitinação afeta os processos celulares, como tráfego de membrana , endocitose e brotamento viral .
Cadeias de poliubiquitina
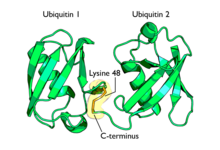
A poliubiquitinação é a formação de uma cadeia de ubiquitina em um único resíduo de lisina na proteína substrato. Após a adição de uma única porção de ubiquitina a um substrato de proteína, outras moléculas de ubiquitina podem ser adicionadas à primeira, produzindo uma cadeia de poliubiquitina. Essas cadeias são feitas ligando o resíduo de glicina de uma molécula de ubiquitina a uma lisina de ubiquitina ligada a um substrato. Ubiquitina tem sete resíduos de lisina e um N-terminal que serve como pontos de ubiquitinação; eles são K6, K11, K27, K29, K33, K48, K63 e M1, respectivamente. As cadeias ligadas à lisina 48 foram as primeiras identificadas e são o tipo de cadeia de ubiquitina mais bem caracterizado. As cadeias K63 também foram bem caracterizadas, enquanto a função de outras cadeias de lisina, cadeias mistas, cadeias ramificadas, cadeias lineares M1 e cadeias heterólogas (misturas de ubiquitina e outras proteínas semelhantes à ubiquitina) permanece mais obscura.
As cadeias de poliubiquitina ligadas à lisina 48 têm como alvo as proteínas para destruição, por um processo conhecido como proteólise . As cadeias de multi-ubiquitina com pelo menos quatro moléculas de ubiquitina de comprimento devem ser anexadas a um resíduo de lisina na proteína condenada para que seja reconhecida pelo proteassoma 26S . Esta é uma estrutura em forma de barril que compreende um núcleo proteolítico central feito de quatro estruturas em anel, flanqueadas por dois cilindros que permitem a entrada seletiva de proteínas ubiquitinadas. Uma vez lá dentro, as proteínas são rapidamente degradadas em pequenos peptídeos (geralmente com 3-25 resíduos de aminoácidos de comprimento). As moléculas de ubiquitina são clivadas da proteína imediatamente antes da destruição e são recicladas para uso posterior. Embora a maioria dos substratos proteicos seja ubiquitinada, há exemplos de proteínas não ubiquitinadas direcionadas ao proteassoma. As cadeias de poliubiquitina são reconhecidas por uma subunidade do proteassoma: S5a / Rpn10. Isso é conseguido por um motivo de interação de ubiquitina (UIM) encontrado em um patch hidrofóbico na região C-terminal da unidade S5a / Rpn10.
As cadeias ligadas à lisina 63 não estão associadas à degradação proteassomal da proteína substrato. Em vez disso, eles permitem a coordenação de outros processos, como tráfego endocítico , inflamação , tradução e reparo de DNA . Nas células, as cadeias ligadas à lisina 63 são ligadas pelo complexo ESCRT-0 , que impede sua ligação ao proteassoma. Este complexo contém duas proteínas, Hrs e STAM1, que contêm um UIM, que permite que ele se ligue a cadeias ligadas a 63 lisina.
Menos se sabe sobre as cadeias de ubiquitina atípicas (não ligadas à lisina 48), mas a pesquisa está começando a sugerir papéis para essas cadeias. Há evidências que sugerem que cadeias atípicas ligadas por lisina 6, 11, 27, 29 e metionina 1 podem induzir degradação proteassomal.
Podem ser formadas cadeias de ubiquitina ramificadas contendo vários tipos de ligação. A função dessas cadeias é desconhecida.
Estrutura
Cadeias com ligações diferentes têm efeitos específicos na proteína à qual estão ligadas, causados por diferenças nas conformações das cadeias de proteínas. As cadeias ligadas a K29-, K33-, K63- e M1 têm uma conformação bastante linear; eles são conhecidos como cadeias de conformação aberta. As cadeias ligadas a K6-, K11- e K48 formam conformações fechadas. As moléculas de ubiquitina em cadeias de conformação aberta não interagem umas com as outras, exceto pelas ligações isopeptídicas covalentes que as ligam. Em contraste, as cadeias de conformação fechadas têm interfaces com resíduos em interação. Alterar as conformações da cadeia expõe e oculta diferentes partes da proteína ubiquitina, e as diferentes ligações são reconhecidas por proteínas que são específicas para as topologias únicas que são intrínsecas à ligação. As proteínas podem se ligar especificamente à ubiquitina por meio dos domínios de ligação da ubiquitina (UBDs). As distâncias entre unidades individuais de ubiquitina em cadeias diferem entre as cadeias ligadas a lisina 63 e 48. Os UBDs exploram isso tendo pequenos espaçadores entre os motivos que interagem com a ubiquitina que se ligam às cadeias de 48 lisina (cadeias de ubiquitina compactas) e espaçadores maiores para as cadeias de 63 lisina. A maquinaria envolvida no reconhecimento de cadeias de poliubiquitina também pode diferenciar entre cadeias ligadas a K63 e cadeias ligadas a M1, demonstrado pelo fato de que as últimas podem induzir degradação proteassomal do substrato.
Função
O sistema de ubiquitinação funciona em uma ampla variedade de processos celulares, incluindo:
- Processamento de antígeno
- Apoptose
- Biogênese de organelas
- Ciclo celular e divisão
- Transcrição e reparo de DNA
- Diferenciação e desenvolvimento
- Resposta imunológica e inflamação
- Degeneração neural e muscular
- Manutenção da pluripotência
- Morfogênese de redes neurais
- Modulação de receptores de superfície celular, canais iônicos e via secretora
- Resposta ao estresse e moduladores extracelulares
- Biogênese ribossômica
- Infecção viral
Proteínas de membrana
A multi-monoubiquitinação pode marcar proteínas transmembrana (por exemplo, receptores ) para remoção das membranas (internalização) e cumprir vários papéis de sinalização dentro da célula. Quando as moléculas transmembrana da superfície celular são marcadas com ubiquitina, a localização subcelular da proteína é alterada, muitas vezes visando a proteína para destruição nos lisossomas. Isso funciona como um mecanismo de feedback negativo, porque muitas vezes a estimulação de receptores por ligantes aumenta sua taxa de ubiquitinação e internalização. Como a monoubiquitinação, as cadeias de poliubiquitina ligadas à lisina 63 também desempenham um papel no tráfego de algumas proteínas de membrana.
Sistema Fougaro
O sistema fougaro (grego; Fougaro, chaminé) é um sistema de sub-organela no núcleo que pode ser um mecanismo para reciclar ou remover moléculas da célula para o meio externo. As moléculas ou peptídeos são ubiquitinados antes de serem liberados do núcleo das células. As moléculas ubiquitinadas são liberadas de forma independente ou associadas a proteínas endossômicas como a Beclin.
Manutenção genômica
O antígeno nuclear de células em proliferação (PCNA) é uma proteína envolvida na síntese de DNA . Em condições fisiológicas normais, o PCNA é sumoilado (uma modificação pós-tradução semelhante à ubiquitinação). Quando o DNA é danificado por radiação ultravioleta ou produtos químicos, a molécula SUMO que está ligada a um resíduo de lisina é substituída por ubiquitina. O PCNA monoubiquitinado recruta polimerases que podem realizar a síntese de DNA com DNA danificado; mas isso é muito sujeito a erros, possivelmente resultando na síntese de DNA mutado. A poliubiquitinação ligada à lisina 63 de PCNA permite que ele execute um desvio de mutação menos sujeito a erros conhecido pela via de troca de modelo.
A ubiquitinação da histona H2AX está envolvida no reconhecimento de danos ao DNA de quebras de fita dupla de DNA. As cadeias de poliubiquitina ligadas à lisina 63 são formadas na histona H2AX pelo par de ligase E2 / E3 , Ubc13-Mms2 / RNF168. Esta cadeia K63 parece recrutar RAP80, que contém um UIM , e RAP80 ajuda a localizar BRCA1 . Esta via acabará por recrutar as proteínas necessárias para o reparo de recombinação homóloga .
Regulação transcricional
As histonas podem ser ubiquitinadas, geralmente na forma de monoubiquitinação (embora ocorram formas poliubiquitinadas). A ubiquitinação das histonas altera a estrutura da cromatina e permite o acesso das enzimas envolvidas na transcrição. Ubiquitina nas histonas também atua como um sítio de ligação para proteínas que ativam ou inibem a transcrição e também podem induzir modificações pós-tradução adicionais da proteína. Todos esses efeitos podem modular a transcrição de genes.
Desubiquitinação
As enzimas desubiquitinantes (DUBs) se opõem ao papel da ubiquinação removendo a ubiquitina das proteínas do substrato. Eles são proteases de cisteína que clivam a ligação amida entre as duas proteínas. Eles são altamente específicos, assim como as ligases E3 que fixam a ubiquitina, com apenas alguns substratos por enzima. Eles podem clivar as ligações isopeptídicas (entre a ubiquitina e a lisina) e as ligações peptídicas (entre a ubiquitina e o terminal N ). Além de remover a ubiquitina das proteínas do substrato, os DUBs têm muitas outras funções dentro da célula. Ubiquitina é expressa como múltiplas cópias unidas em uma cadeia (poliubiquitina) ou anexadas a subunidades ribossômicas. Os DUBs clivam essas proteínas para produzir ubiquitina ativa. Eles também reciclam a ubiquitina que foi ligada a pequenas moléculas nucleofílicas durante o processo de ubiquitinação. A monoubiquitina é formada por DUBs que clivam a ubiquitina das cadeias de poliubiquitina livres que foram previamente removidas das proteínas.
Domínios de ligação à ubiquitina
| Domínio | Número de Proteínas
no proteoma |
Comprimento
(aminoácidos) |
Ligação Ubiquitina
Afinidade |
|---|---|---|---|
| DEIXA |
S. cerevisiae : 7
H. sapiens : 21 |
42-43 | ~ 2–160 μM |
| GATII |
S. cerevisiae : 2
H. sapiens : 14 |
135 | ~ 180 μM |
| COLA |
S. cerevisiae :?
H. sapiens :? |
~ 135 | ~ 460 μM |
| NZF |
S. cerevisiae : 1
H. sapiens : 25 |
~ 35 | ~ 100-400 μM |
| PAZ |
S. cerevisiae : 5
H. sapiens : 16 |
~ 58 | Não conhecido |
| UBA |
S. cerevisiae : 10
H. sapiens : 98 |
45–55 | ~ 0,03–500 μM |
| UEV |
S. cerevisiae : 2
H. sapiens :? |
~ 145 | ~ 100–500 μM |
| UIM |
S. cerevisiae : 8
H. sapiens : 71 |
~ 20 | ~ 100-400 μM |
| VHS |
S. cerevisiae : 4
H. sapiens : 28 |
150 | Não conhecido |
Os domínios de ligação à ubiquitina (UBDs) são domínios modulares de proteínas que se ligam de forma não covalente à ubiquitina, esses motivos controlam vários eventos celulares. Estruturas moleculares detalhadas são conhecidas para uma série de UBDs, a especificidade de ligação determina seu mecanismo de ação e regulação e como regula proteínas e processos celulares.
Associações de doenças
Patogênese
A via da ubiquitina tem sido implicada na patogênese de uma ampla gama de doenças e distúrbios, incluindo:
- Neurodegeneração
- Infecção e imunidade
- Distúrbios genéticos
- Câncer
Neurodegeneração
Ubiquitina está implicado em doenças neurodegenerativas associadas com a disfunção Proteostase, incluindo doença de Alzheimer , doença do neurónio motor , doença de Huntington e doença de Parkinson . Variantes de transcrição que codificam diferentes isoformas de ubiquilina-1 são encontradas em lesões associadas à doença de Alzheimer e Parkinson . Demonstrou-se que níveis mais altos de ubiquilina no cérebro diminuem a malformação da proteína precursora da amilóide (APP) , que desempenha um papel fundamental no desencadeamento da doença de Alzheimer. Por outro lado, níveis mais baixos de ubiquilina-1 no cérebro foram associados ao aumento da malformação de APP . Uma mutação de frameshift na ubiquitina B pode resultar em um peptídeo truncado sem a glicina C-terminal . Este peptídeo anormal, conhecido como UBB + 1 , demonstrou se acumular seletivamente na doença de Alzheimer e outras tauopatias .
Infecção e imunidade
Ubiquitina e moléculas semelhantes a ubiquitina regulam extensivamente as vias de transdução de sinal imunológico em praticamente todos os estágios, incluindo repressão em estado estacionário, ativação durante a infecção e atenuação após eliminação. Sem essa regulação, a ativação imunológica contra patógenos pode ser defeituosa, resultando em doença crônica ou morte. Alternativamente, o sistema imunológico pode ficar hiperativado e órgãos e tecidos podem ser submetidos a danos autoimunes .
Por outro lado, os vírus devem bloquear ou redirecionar os processos da célula hospedeira, incluindo a imunidade, para se replicar com eficácia, embora muitos vírus relevantes para a doença tenham genomas limitados em termos de informação . Por causa de seu grande número de funções na célula, a manipulação do sistema de ubiquitina representa uma maneira eficiente de tais vírus bloquear, subverter ou redirecionar processos críticos da célula hospedeira para suportar sua própria replicação.
A proteína do gene I induzível por ácido retinóico ( RIG-I ) é um sensor primário do sistema imunológico para RNA viral e outro RNA invasivo em células humanas. A via de sinalização imunológica do receptor semelhante ao RIG-I ( RLR ) é uma das mais extensivamente estudadas em termos do papel da ubiquitina na regulação imunológica.
Distúrbios genéticos
- A síndrome de Angelman é causada por uma interrupção do UBE3A , que codifica uma enzima ubiquitina ligase (E3) denominada E6-AP.
- A síndrome de Von Hippel-Lindau envolve a interrupção de uma ubiquitina E3 ligase denominada supressor de tumor VHL, ou gene VHL .
- Anemia de Fanconi : Oito dos treze genes identificados cuja interrupção pode causar esta doença codificam proteínas que formam um grande complexo ubiquitina ligase (E3).
- A síndrome 3-M é um distúrbio de retardo de crescimento autossômico recessivo associado a mutações da ubiquitina ligase Cullin7 E3.
Uso diagnóstico
A imunohistoquímica usando anticorpos para ubiquitina pode identificar acúmulos anormais dessa proteína dentro das células, indicando um processo de doença. Esses acúmulos de proteínas são chamados de corpos de inclusão (que é um termo geral para qualquer coleção microscopicamente visível de material anormal em uma célula). Exemplos incluem:
- Emaranhados neurofibrilares na doença de Alzheimer
- Corpo de Lewy na doença de Parkinson
- Escolha corpos na doença de Pick
- Inclusões na doença do neurônio motor e doença de Huntington
- Corpos de Mallory na doença hepática alcoólica
- Fibras Rosenthal em astrócitos
Link para câncer
A modificação pós-tradução de proteínas é um mecanismo geralmente usado na sinalização de células eucarióticas . Ubiquitinação, ou conjugação de ubiquitina a proteínas , é um processo crucial para a progressão do ciclo celular e proliferação e desenvolvimento celular . Embora a ubiquitinação normalmente sirva como um sinal para a degradação de proteínas através do proteassoma 26S , ela também pode servir para outros processos celulares fundamentais, por exemplo, em endocitose , ativação enzimática e reparo de DNA. Além disso, uma vez que a ubiquitinação funciona para regular rigidamente o nível celular de ciclinas , é esperado que seu desregulamento tenha impactos severos. A primeira evidência da importância da via ubiquitina / proteassoma nos processos oncogênicos foi observada devido à alta atividade antitumoral dos inibidores de proteassoma. Vários estudos têm mostrado que defeitos ou alterações nos processos de ubiquitinação estão comumente associados ou presentes no carcinoma humano. As doenças malignas podem ser desenvolvidas por meio da perda de mutação de função diretamente no gene supressor de tumor , aumento da atividade de ubiquitinação e / ou atenuação indireta da ubiquitinação devido à mutação em proteínas relacionadas.
Perda direta de mutação de função da ubiquitina ligase E3
Carcinoma de células renais
O gene VHL ( Von Hippel – Lindau ) codifica um componente de uma ubiquitina ligase E3 . O complexo VHL tem como alvo o membro da família do fator de transcrição induzível por hipóxia (HIF) para degradação ao interagir com o domínio de destruição dependente de oxigênio sob condição normóxica. O HIF ativa alvos a jusante, como o fator de crescimento endotelial vascular (VEGF), promovendo a angiogênese . As mutações no VHL previnem a degradação do HIF e, portanto, levam à formação de lesões hipervasculares e tumores renais.
Câncer de mama
O gene BRCA1 é outro gene supressor de tumor em humanos que codifica a proteína BRCA1 que está envolvida na resposta a danos no DNA. A proteína contém um motivo RING com atividade E3 Ubiquitin Ligase. BRCA1 poderia formar dímero com outras moléculas, como BARD1 e BAP1 , por sua atividade de ubiquitinação. Mutações que afetam a função da ligase são freqüentemente encontradas e associadas a vários tipos de câncer.
Cyclin E
Como os processos na progressão do ciclo celular são os processos mais fundamentais para o crescimento e diferenciação celular, e são os mais comumente alterados em carcinomas humanos, espera-se que as proteínas reguladoras do ciclo celular estejam sob forte regulação. O nível de ciclinas, como o nome sugere, é alto apenas em determinado ponto do ciclo celular. Isso é alcançado pelo controle contínuo dos níveis de ciclinas / CDKs por meio de ubiquitinação e degradação. Quando a ciclina E é associada ao CDK2 e é fosforilada, uma proteína F-box Fbw7 associada a SCF reconhece o complexo e, portanto, o direciona para a degradação. Mutações no Fbw7 foram encontradas em mais de 30% dos tumores humanos, caracterizando-o como uma proteína supressora de tumor.
Aumento da atividade de ubiquitinação
Câncer cervical
Tipos oncogênicos do papilomavírus humano (HPV) são conhecidos por sequestrar a via celular ubiquitina- proteassoma para infecção viral e replicação. As proteínas E6 do HPV se ligam ao terminal N da ubiquitina ligase E6-AP E3 celular, redirecionando o complexo para se ligar ao p53 , um gene supressor de tumor bem conhecido cuja inativação é encontrada em muitos tipos de câncer. Assim, o p53 sofre ubiquitinação e degradação mediada por proteassoma. Enquanto isso, o E7, outro dos genes do HPV expressos precocemente, se ligará ao Rb , também um gene supressor de tumor, mediando sua degradação. A perda de p53 e Rb nas células permite que ocorra uma proliferação celular ilimitada.
regulamento p53
A amplificação do gene frequentemente ocorre em vários casos de tumor, incluindo de MDM2 , um gene que codifica para uma RING E3 Ubiquitina ligase responsável pela regulação negativa da atividade de p53. MDM2 tem como alvo p53 para ubiquitinação e degradação proteassomal, mantendo assim seu nível apropriado para a condição celular normal. A superexpressão de MDM2 causa perda de atividade de p53 e, portanto, permite que as células tenham um potencial replicativo ilimitado.
p27
Outro gene que é alvo da amplificação do gene é o SKP2 . SKP2 é uma proteína F-box que atua no reconhecimento do substrato para ubiquitinação e degradação. SKP2 tem como alvo p27 Kip-1 , um inibidor de quinases dependentes de ciclina ( CDKs ). As CDKs2 / 4 fazem parceria com as ciclinas E / D, respectivamente, família de reguladores do ciclo celular para controlar a progressão do ciclo celular através da fase G1. O baixo nível de proteína p27 Kip-1 é freqüentemente encontrado em vários cânceres e é devido à hiperativação da proteólise mediada por ubiquitina por meio da superexpressão de SKP2.
Efp
Efp , ou proteína dedo-RING induzível por estrogênio, é uma ubiquitina ligase E3 cuja superexpressão demonstrou ser a principal causa de câncer de mama independente de estrogênio . O substrato da Efp é a proteína 14-3-3 que regula negativamente o ciclo celular.
Evasão da Ubiquitinação
Câncer colorretal
O gene associado ao câncer colorretal é a polipose adenomatosa do colo (APC), que é um gene supressor de tumor clássico . O produto do gene APC tem como alvo a beta-catenina para degradação via ubiquitinação no terminal N , regulando assim seu nível celular. A maioria dos casos de câncer colorretal é encontrada com mutações no gene APC. No entanto, nos casos em que o gene APC não está mutado, as mutações são encontradas no terminal N da beta-catenina, o que a torna livre de ubiquitinação e, portanto, aumenta a atividade.
Glioblastoma
Por ser o câncer mais agressivo de origem cerebral, as mutações encontradas em pacientes com glioblastoma estão relacionadas à deleção de uma parte do domínio extracelular do receptor do fator de crescimento epidérmico (EGFR). Esta deleção faz com que a ligase CBL E3 seja incapaz de se ligar ao receptor para sua reciclagem e degradação por meio de uma via ubiquitina-lisossomal. Assim, o EGFR é constitutivamente ativo na membrana celular e ativa seus efetores a jusante que estão envolvidos na proliferação e migração celular.
Ubiquitinação dependente de fosforilação
A interação entre a ubiquitinação e a fosforilação tem sido um interesse contínuo de pesquisa, uma vez que a fosforilação freqüentemente serve como um marcador onde a ubiquitinação leva à degradação. Além disso, a ubiquitinação também pode atuar para ligar / desligar a atividade da quinase de uma proteína. O papel crítico da fosforilação é amplamente enfatizado na ativação e remoção da autoinibição na proteína Cbl . Cbl é uma ubiquitina ligase E3 com um domínio de dedo RING que interage com seu domínio de ligação de tirosina quinase (TKB) , evitando a interação do domínio RING com uma enzima E2 de conjugação de ubiquitina . Essa interação intramolecular é uma regulação de autoinibição que impede seu papel como regulador negativo de vários fatores de crescimento e da sinalização de tirosina quinase e ativação de células T. A fosforilação de Y363 alivia a autoinibição e aumenta a ligação a E2. Demonstrou-se que mutações que tornam a proteína Cbl disfuncional devido à perda de sua função ligase / supressora de tumor e manutenção de sua função oncogênica / sinalização positiva causam o desenvolvimento de câncer.
Como alvo de drogas
Triagem de substratos de ubiquitina ligase
A identificação de substratos da ligase E3 é crítica para entender sua implicação em doenças humanas, uma vez que a desregulação das interações do substrato E3 são frequentemente servidas como causa principal em muitos. Para superar a limitação do mecanismo usado para identificar os substratos da Ubiquitina Ligase E3, um sistema chamado 'Global Protein Stability (GPS) Profiling' foi desenvolvido em 2008. Este sistema de alto rendimento fez uso de proteínas repórter fundidas com milhares de potenciais substratos de forma independente. Por inibição da atividade da ligase (através da criação de Cul1 dominante negativo, portanto, torna a ubiquitinação para não ocorrer), a atividade de repórter aumentada mostra que os substratos identificados estão sendo acumulados. Essa abordagem adicionou um grande número de novos substratos à lista de substratos da ligase E3.
Possíveis aplicações terapêuticas
Bloqueio do reconhecimento de substrato específico pelas ligases E3, por exemplo, Bortezomib .
Desafio
Encontrar uma molécula específica que iniba seletivamente a atividade de uma determinada ligase E3 e / ou as interações proteína-proteína implicadas na doença permanece como uma área de pesquisa importante e em expansão. Além disso, como a ubiquitinação é um processo de várias etapas com vários participantes e formas intermediárias, a consideração das interações muito complexas entre os componentes deve ser levada em consideração ao projetar os inibidores de moléculas pequenas.
Proteínas semelhantes
Embora a ubiquitina seja o modificador pós-tradução mais conhecido, há uma família crescente de proteínas semelhantes à ubiquitina (UBLs) que modificam os alvos celulares em uma via que é paralela, mas distinta daquela da ubiquitina. UBLs conhecidos incluem: pequeno modificador semelhante à ubiquitina ( SUMO ), proteína de reatividade cruzada da ubiquitina (UCRP, também conhecida como gene 15 ISG15 estimulado por interferon ), modificador 1 relacionado à ubiquitina ( URM1 ), célula precursora neuronal expressa proteína-8 regulada para baixo no desenvolvimento ( NEDD8 , também chamada de Rub1 em S. cerevisiae ), antígeno leucocitário humano F-associado ( FAT10 ), autofagia-8 ( ATG8 ) e -12 ( ATG12 ), Poucas proteínas semelhantes à ubiquitina ( FUB1 ), MUB (UBL ancorado na membrana), modificador de dobra de ubiquitina-1 ( UFM1 ) e proteína semelhante à ubiquitina-5 ( UBL5 , que é conhecida como homóloga à ubiquitina-1 [Hub1] em S. pombe ). Embora essas proteínas compartilhem apenas uma modesta identidade de sequência primária com a ubiquitina, elas estão intimamente relacionadas tridimensionalmente. Por exemplo, SUMO compartilha apenas 18% de identidade de sequência, mas eles contêm a mesma dobra estrutural. Esta dobra é chamada de "dobra da ubiquitina". FAT10 e UCRP contêm dois. Esta prega beta globular compacta é encontrada na ubiquitina, UBLs e proteínas que compreendem um domínio semelhante à ubiquitina, por exemplo, a proteína de duplicação do corpo do polo do fuso S. cerevisiae , Dsk2, e a proteína NER, Rad23, ambas contêm domínios de ubiquitina N-terminais .
Essas moléculas relacionadas têm novas funções e influenciam diversos processos biológicos. Também há regulação cruzada entre as várias vias de conjugação, uma vez que algumas proteínas podem ser modificadas por mais de um UBL e, às vezes, até no mesmo resíduo de lisina. Por exemplo, a modificação SUMO freqüentemente atua de forma antagônica à ubiquitinação e serve para estabilizar substratos de proteínas. As proteínas conjugadas com UBLs normalmente não são direcionadas para degradação pelo proteassoma, mas funcionam em diversas atividades regulatórias. A ligação de UBLs pode alterar a conformação do substrato, afetar a afinidade por ligantes ou outras moléculas interagentes, alterar a localização do substrato e influenciar a estabilidade da proteína.
Os UBLs são estruturalmente semelhantes à ubiquitina e são processados, ativados, conjugados e liberados dos conjugados por etapas enzimáticas semelhantes aos mecanismos correspondentes da ubiquitina. UBLs também são traduzidos com extensões C-terminal que são processadas para expor o invariante C-terminal LRGG. Esses modificadores têm suas próprias enzimas específicas E1 (ativação), E2 (conjugação) e E3 (ligação) que conjugam os UBLs a alvos intracelulares. Esses conjugados podem ser revertidos por isopeptidases específicas de UBL que possuem mecanismos semelhantes aos das enzimas desubiquitinantes.
Em algumas espécies, o reconhecimento e a destruição das mitocôndrias dos espermatozoides por meio de um mecanismo que envolve a ubiquitina é responsável pela eliminação das mitocôndrias dos espermatozoides após a fertilização.
Origens procarióticas
Ubiquitina se acredita ter descido a partir de proteínas bacterianas semelhantes a ISSO ( O32583 ) ou moad ( P30748 ). Essas proteínas procarióticas, apesar de terem pouca identidade de sequência (ThiS tem 14% de identidade com a ubiquitina), compartilham a mesma dobra protéica. Essas proteínas também compartilham a química do enxofre com a ubiquitina. MoaD, que está envolvido na biossíntese de molibdopterina , interage com MoeB, que atua como uma enzima ativadora de ubiquitina E1 para MoaD, fortalecendo a ligação entre essas proteínas procarióticas e o sistema de ubiquitina. Um sistema semelhante existe para ThiS, com sua enzima semelhante a E1 ThiF . Também se acredita que a proteína Urm-1 da Saccharomyces cerevisiae , um modificador relacionado à ubiquitina, é um "fóssil molecular" que conecta a relação evolutiva com as moléculas procarióticas semelhantes à ubiquitina e a ubiquitina.
Archaea tem um homólogo funcionalmente mais próximo do sistema de modificação de ubiquitina, onde a "sampilação" com SAMPs (pequenas proteínas modificadoras de archaea) é realizada. O sistema de sampilação usa apenas E1 para guiar proteínas para o proteossomo . Proteoarchaeota , que está relacionado ao ancestral dos eucariotos, possui todas as enzimas E1, E2 e E3, além de um sistema Rpn11 regulado. Ao contrário do SAMP, que é mais semelhante ao ThiS ou MoaD, a ubiquitina do Proteoarchaeota é mais semelhante aos homólogos eucarióticos.
Proteína procariótica semelhante à ubiquitina (Pup) e ubiquitina bacteriana (UBact)
A proteína semelhante à ubiquitina procariótica (Pup) é um análogo funcional da ubiquitina que foi encontrada no filo bacteriano gram-positivo Actinobacteria . Ele tem a mesma função (direcionar proteínas para degradações), embora a enzimologia de ubiquitinação e pupilação seja diferente e as duas famílias não compartilhem homologia. Em contraste com a reação de três etapas de ubiquitinação, a pupilação requer duas etapas, portanto, apenas duas enzimas estão envolvidas na pupilação.
Em 2017, homólogos de Pup foram relatados em cinco filos de bactérias gram-negativas , em sete filos bacterianos candidatos e em um archaeon. As sequências dos homólogos de Pup são muito diferentes das sequências de Pup em bactérias gram-positivas e foram denominadas bacterianas Ubiquitina (UBact), embora a distinção ainda não tenha sido provada ser filogeneticamente apoiada por uma origem evolutiva separada e sem evidência experimental.
A descoberta do sistema Pup / UBact-proteassoma em bactérias gram-positivas e gram-negativas sugere que o sistema Pup / UBact-proteassoma evoluiu em bactérias antes da divisão em clados gram positivos e negativos há mais de 3000 milhões de anos ou, que esses sistemas foram adquiridos por diferentes linhagens bacterianas por meio de transferência (ões) horizontal (ões) de genes de um terceiro, ainda desconhecido, organismo. Em apoio à segunda possibilidade, dois loci UBact foram encontrados no genoma de um Archaeon metanotrófico anaeróbico não cultivado (ANME-1; locus CBH38808.1 e locus CBH39258.1 ).
Proteínas humanas contendo domínio de ubiquitina
Isso inclui proteínas do tipo ubiquitina.
ANUBL1 ; BAG1 ; BAT3 / BAG6 ; C1orf131 ; DDI1 ; DDI2 ; FAU ; HERPUD1 ; HERPUD2 ; HOPS ; IKBKB ; ISG15 ; LOC391257 ; MIDN ; NEDD8 ; OASL ; PARK2 ; RAD23A ; RAD23B ; RPS27A ; SACS ; 8U SF3A1 ; SUMO1 ; SUMO2 ; SUMO3 ; SUMO4 ; TMUB1 ; TMUB2 ; UBA52 ; UBB ; UBC ; UBD ; UBFD1 ; UBL4 ; UBL4A ; UBL4B ; UBL7 ; UBLCP1 ; UBQLN1 ; UBQLN2 ; UBQLN3 ; UBQLN4 ; UBQLNL ; UBTD1 ; UBTD2 ; UHRF1 ; UHRF2 ;
Proteínas relacionadas
Previsão de ubiquitinação
Os programas de previsão disponíveis atualmente são:
- UbiPred é um servidor de previsão baseado em SVM que usa 31 propriedades físico-químicas para prever locais de ubiquitinação.
- UbPred é um preditor aleatório baseado na floresta de potenciais locais de ubiquitinação em proteínas. Ele foi treinado em um conjunto combinado de 266 locais de ubiquitinação verificados experimentalmente não redundantes disponíveis em nossos experimentos e em dois estudos de proteômica em grande escala.
- CKSAAP_UbSite é uma previsão baseada em SVM que emprega a composição de pares de aminoácidos com espaçamento k em torno de um site de consulta (ou seja, qualquer lisina em uma sequência de consulta) como entrada, usa o mesmo conjunto de dados que UbPred.
Podcast
- Investigar o sistema ubiquitina proteassoma foi o foco de um Dementia Researcher Podcast, [1] . O podcast foi publicado em 16 de agosto de 2021, apresentado pela Professora Selina Wray da University College London.
Veja também
Referências
links externos
- GeneReviews / NCBI / NIH / UW entrada na síndrome de Angelman
- Entradas OMIM na síndrome de Angelman
- Entrada UniProt para ubiquitina
- "7.340 Ubiquitination: The Proteasome and Human Disease" . MIT OpenCourseWare . 2004. Notas do curso do MIT.
- Ubiquitina nos títulos de assuntos médicos da Biblioteca Nacional de Medicina dos EUA (MeSH)