Agregação de proteínas - Protein aggregation
Agregação de proteínas é um fenómeno biológico no qual as proteínas intrinsecamente desordenados ou mis-dobradas proteas agregada (isto é, se acumulam e se agregam) quer intra ou extracelularmente. Agregados de proteínas mal dobrados são freqüentemente correlacionados com doenças. Na verdade, os agregados de proteínas têm sido implicados em uma ampla variedade de doenças conhecidas como amiloidoses , incluindo ALS , Alzheimer , Parkinson e doença de príon .
Após a síntese, as proteínas normalmente se dobram em uma conformação tridimensional particular que é a mais termodinamicamente favorável: seu estado nativo. Esse processo de dobramento é impulsionado pelo efeito hidrofóbico : uma tendência de porções hidrofóbicas (temerosas de água) da proteína se protegerem do ambiente hidrofílico (que amam a água) da célula enterrando-se no interior da proteína. Assim, o exterior de uma proteína é tipicamente hidrofílico, enquanto o interior é tipicamente hidrofóbico.
As estruturas das proteínas são estabilizadas por interações não covalentes e ligações dissulfeto entre dois resíduos de cisteína . As interações não covalentes incluem interações iônicas e fracas interações de van der Waals . As interações iônicas se formam entre um ânion e um cátion e formam pontes de sal que ajudam a estabilizar a proteína. As interações de Van der Waals incluem interações não polares (isto é, força de dispersão de London ) e interações polares (isto é , ligações de hidrogênio , ligação dipolo-dipolo ). Estes desempenham um papel importante na estrutura secundária de uma proteína, como a formação de uma hélice alfa ou uma folha beta, e na estrutura terciária. As interações entre os resíduos de aminoácidos em uma proteína específica são muito importantes na estrutura final dessa proteína.
Quando há mudanças nas interações não covalentes, como pode acontecer com uma mudança na sequência de aminoácidos, a proteína é suscetível a dobramento incorreto ou desdobramento. Nestes casos, se a célula não ajudar a proteína a se dobrar novamente ou degradar a proteína não dobrada, a proteína não dobrada / dobrada incorretamente pode agregar, em que as porções hidrofóbicas expostas da proteína podem interagir com as manchas hidrofóbicas expostas de outras proteínas . Existem três tipos principais de agregados de proteínas que podem se formar: agregados amorfos, oligômeros e fibrilas amilóides .
Causas
A agregação de proteínas pode ocorrer devido a uma variedade de causas. Existem quatro classes nas quais essas causas podem ser categorizadas, que são detalhadas a seguir.
Mutações
Mutações que ocorrem na sequência de DNA podem ou não afetar a sequência de aminoácidos da proteína. Quando a sequência é afetada, um aminoácido diferente pode alterar as interações entre as cadeias laterais que afetam o enovelamento da proteína. Isso pode levar a regiões hidrofóbicas expostas da proteína que se agregam com a mesma proteína mal dobrada / não dobrada ou uma proteína diferente.
Além de mutações nas próprias proteínas afetadas, a agregação de proteínas também pode ser causada indiretamente por meio de mutações em proteínas em vias regulatórias, como a via de redobramento ( chaperones moleculares ) ou a via da ubiquitina-proteassoma (ubiquitina ligases). As acompanhantes ajudam no redobramento da proteína, proporcionando um ambiente seguro para a proteína se dobrar. As ubiquitinas ligases têm como alvo proteínas para degradação por meio da modificação da ubiquitina.
Problemas com a síntese de proteínas
A agregação de proteínas pode ser causada por problemas que ocorrem durante a transcrição ou tradução . Durante a transcrição, o DNA é copiado em mRNA, formando uma fita de pré-mRNA que sofre processamento de RNA para formar mRNA. Durante a tradução, os ribossomos e o tRNA ajudam a traduzir a sequência do mRNA em uma sequência de aminoácidos. Se surgirem problemas durante qualquer uma das etapas, criando uma fita de mRNA incorreta e / ou uma sequência de aminoácidos incorreta, isso pode fazer com que a proteína seja dobrada incorretamente, levando à agregação da proteína.
Estresses ambientais
Estresses ambientais como temperaturas extremas e pH ou estresse oxidativo também podem levar à agregação de proteínas. Uma dessas doenças é a crioglobulinemia .
As temperaturas extremas podem enfraquecer e desestabilizar as interações não covalentes entre os resíduos de aminoácidos. Os pHs fora da faixa de pH da proteína podem alterar o estado de protonação dos aminoácidos, o que pode aumentar ou diminuir as interações não covalentes. Isso também pode levar a interações menos estáveis e resultar no desdobramento de proteínas.
O estresse oxidativo pode ser causado por radicais como espécies reativas de oxigênio (ROS). Esses radicais instáveis podem atacar os resíduos de aminoácidos, levando à oxidação das cadeias laterais (por exemplo , cadeias laterais aromáticas , cadeias laterais de metionina ) e / ou clivagem das ligações polipeptídicas. Isso pode afetar as interações não covalentes que mantêm a proteína unida corretamente, o que pode causar a desestabilização da proteína e pode fazer com que a proteína se desdobre.
Envelhecimento
As células têm mecanismos que podem redobrar ou degradar agregados de proteínas. No entanto, à medida que as células envelhecem, esses mecanismos de controle são enfraquecidos e a célula é menos capaz de resolver os agregados.
A hipótese de que a agregação de proteínas é um processo causador do envelhecimento pode ser testada agora, uma vez que alguns modelos de envelhecimento retardado estão disponíveis. Se o desenvolvimento de agregados de proteína foi um processo independente do envelhecimento, retardar o envelhecimento não mostrará nenhum efeito na taxa de proteotoxicidade ao longo do tempo. No entanto, se o envelhecimento estiver associado ao declínio da atividade dos mecanismos de proteção contra a proteotoxicidade, os modelos de envelhecimento lento apresentariam agregação e proteotoxicidade reduzidas. Para resolver este problema, vários ensaios de toxicidade foram realizados em C. elegans . Esses estudos indicaram que a redução da atividade de sinalização de insulina / IGF (IIS), uma via regulatória proeminente do envelhecimento, protege da agregação de proteínas tóxicas ligadas à neurodegeneração. A validade desta abordagem foi testada e confirmada em mamíferos, pois a redução da atividade da via de sinalização do IGF-1 protegeu os camundongos modelo de Alzheimer das deficiências comportamentais e bioquímicas associadas à doença.
Localização agregada
Vários estudos têm mostrado que as respostas celulares à agregação de proteínas são bem reguladas e organizadas. Os agregados de proteínas localizam-se em áreas específicas da célula, e pesquisas foram feitas sobre essas localizações em procariotos (E. coli) e eucariotos (leveduras, células de mamíferos).
Bactérias
Os agregados das bactérias terminam assimetricamente em um dos pólos da célula, o "pólo mais antigo". Depois que a célula se divide, as células filhas com o pólo mais antigo obtêm o agregado de proteína e crescem mais lentamente do que as células filhas sem o agregado. Isso fornece um mecanismo de seleção natural para reduzir os agregados de proteínas na população bacteriana.
Fermento
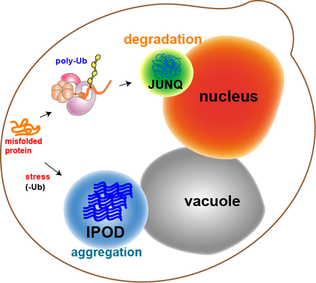
A maioria dos agregados de proteína nas células de levedura é redobrada por chaperones moleculares. No entanto, alguns agregados, como as proteínas danificadas por oxidação ou as proteínas marcadas para degradação, não podem ser redobradas. Em vez disso, existem dois compartimentos nos quais eles podem terminar. Os agregados de proteínas podem ser localizados no compartimento de controle de qualidade Justtanuclear ( JUNQ ), que está próximo à membrana nuclear, ou no depósito de proteína insolúvel ( IPOD ), próximo ao vacúolo em células de levedura. Os agregados de proteínas localizam-se na JUNQ quando são ubiquitinados e direcionados para degradação. As proteínas agregadas e insolúveis localizam-se no IPOD como uma deposição mais permanente. Há evidências de que as proteínas aqui podem ser removidas por autofagia. Essas duas vias trabalham juntas, pois as proteínas tendem a chegar ao IPOD quando a via do proteassoma está sendo sobrecarregada.
Células de mamífero
Em células de mamíferos, esses agregados de proteínas são denominados "agrossomos" e são formados quando a célula está doente. Isso ocorre porque os agregados tendem a se formar quando há proteínas heterólogas presentes na célula, o que pode surgir quando a célula sofre mutação. A ubiquitina ligase E3 é capaz de reconhecer proteínas mal dobradas e ubiquiná-las. HDAC6 pode então ligar-se à ubiquitina e à proteína motora dineína para trazer os agregados marcados para o centro de organização dos microtúbulos ( MTOC ). Lá, eles se agrupam em uma esfera que circunda o MTOC. Eles trazem acompanhantes e proteassomas e ativam a autofagia.
Eliminação
Existem dois sistemas principais de controle de qualidade de proteínas na célula que são responsáveis pela eliminação de agregados de proteínas. As proteínas mal dobradas podem ser redobradas pelo sistema bi-chaperone ou degradadas pelo sistema ubiquitina proteassoma ou autofagia.
Redobramento
O sistema bi-chaperone utiliza chaperonas Hsp70 (DnaK-DnaJ-GrpE em E. coli e Ssa1-Ydj1 / Sis1-Sse1 / Fe1 em levedura) e Hsp100 (ClpB em E. coli e Hsp104 em levedura) para desagregação e redobramento de proteínas .
Hsp70 interage com os agregados de proteína e recruta Hsp100. Hsp70 estabiliza um Hsp100 ativado. As proteínas Hsp100 têm loops de poros aromáticos que são usados para atividade de threading para separar polipeptídeos únicos. Esta atividade de segmentação pode ser iniciada no terminal N, terminal C ou no meio do polipeptídeo. O polipeptídeo é translocado através de Hsp100 em uma série de etapas, utilizando um ATP em cada etapa. O polipeptídeo se desdobra e pode então se redobrar sozinho ou com a ajuda de proteínas de choque térmico.
Degradação
As proteínas mal dobradas podem ser eliminadas através do sistema ubiquitina-proteassoma ( UPS ). Isso consiste em uma via E1-E2-E3 que ubiquina proteínas para marcá-las para degradação. Em eucariotos, as proteínas são degradadas pelo proteassoma 26S. Em células de mamíferos, a ligase E3, proteína de interação com Hsp70 do terminal carboxi (CHIP), tem como alvo proteínas ligadas a Hsp70. Na levedura, as ligases E3 Doa10 e Hrd1 têm funções semelhantes nas proteínas do retículo endoplasmático .
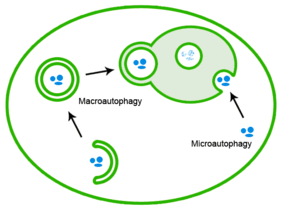
As proteínas mal dobradas também podem ser eliminadas por meio de autofagia, na qual os agregados de proteína são entregues ao lisossoma.
Toxicidade
Embora se tenha pensado que os próprios agregados de proteínas maduras são tóxicos, evidências recentes sugerem que são, de fato, os agregados de proteínas imaturos que são mais tóxicos. As manchas hidrofóbicas desses agregados podem interagir com outros componentes da célula e danificá-los. As hipóteses são de que a toxicidade dos agregados proteicos está relacionada a mecanismos de sequestro de componentes celulares, geração de espécies reativas de oxigênio e ligação a receptores específicos na membrana ou por rompimento de membranas. Um ensaio quantitativo foi usado para determinar que as espécies de alto peso molecular são responsáveis pela permeação da membrana. Sabe-se que agregados de proteínas in vitro podem desestabilizar bicamadas fosfolipídicas artificiais, levando à permeabilização da membrana.
Veja também
Referências
- ^ Aguzzi, A .; O'Connor, T. (março de 2010). "Doenças de agregação de proteínas: patogenicidade e perspectivas terapêuticas". Nature Reviews Drug Discovery . 9 (3): 237–48. doi : 10.1038 / nrd3050 . PMID 20190788 . S2CID 5756683 .
- ^ Stefani, M .; Dobson, CM. (Novembro de 2003). "Agregação de proteínas e toxicidade agregada: novos insights sobre o dobramento de proteínas, doenças de dobramento incorreto e evolução biológica". J Mol Med (Berl) . 81 (11): 678–99. doi : 10.1007 / s00109-003-0464-5 . PMID 12942175 . S2CID 23544974 .
- ^ De Felice, FG .; Vieira, MN .; Meirelles, MN .; Morozova-Roche, LA .; Dobson, CM .; Ferreira, ST. (Julho de 2004). "Formação de agregados amilóides a partir da lisozima humana e suas variantes associadas à doença usando pressão hidrostática". FASEB J . 18 (10): 1099–101. doi : 10.1096 / fj.03-1072fje . PMID 15155566 . S2CID 13647147 .
- ^ Tanzi, RE .; Bertram, L. (fevereiro de 2005). "Vinte anos da hipótese amilóide da doença de Alzheimer: uma perspectiva genética" . Cell . 120 (4): 545–55. doi : 10.1016 / j.cell.2005.02.008 . PMID 15734686 . S2CID 206559875 .
- ^ Brüning, Ansgar; Jückstock, Julia (01/01/2015). "Proteínas mal dobradas: de pequenos vilões a pequenos ajudantes na luta contra o câncer" . Fronteiras em Oncologia . 5 : 47. doi : 10.3389 / fonc.2015.00047 . PMC 4338749 . PMID 25759792 .
- ^ Gething, MJ .; Sambrook, J. (janeiro de 1992). "Enrolamento de proteínas na célula". Nature . 355 (6355): 33–45. Bibcode : 1992Natur.355 ... 33G . doi : 10.1038 / 355033a0 . PMID 1731198 . S2CID 4330003 .
- ^ Roberts, CJ. (Dezembro de 2007). "Cinética de agregação de proteína não nativa". Biotechnol Bioeng . 98 (5): 927–38. doi : 10.1002 / bit.21627 . PMID 17705294 . S2CID 21787377 .
- ^ Cox, David L .; Nelson, Michael M. (2013). Lehninger Principles of Biochemistry . Nova York: WH Freeman. p. 143. ISBN 978-1-4292-3414-6.
- ^ Berke, Sarah J Shoesmith; Paulson, Henry L (01/06/2003). "A agregação de proteínas e a via do proteassoma da ubiquitina: ganhando a mão UPPer na neurodegeneração". Opinião atual em Genética e Desenvolvimento . 13 (3): 253–261. doi : 10.1016 / S0959-437X (03) 00053-4 . PMID 12787787 .
- ^ a b Tecelão, Robert F. (2012). Molecular Biology . Nova York: McGraw-Hill. pp. 122–156, 523–600. ISBN 978-0-07-352532-7.
- ^ a b c d Tyedmers, Jens; Mogk, Axel; Bukau, Bernd (novembro de 2010). "Estratégias celulares para controlar a agregação de proteínas". Nature Reviews Molecular Cell Biology . 11 (11): 777–788. doi : 10.1038 / nrm2993 . PMID 20944667 . S2CID 22449895 .
- ^ Stadtman, ER; Levine, RL (29/07/2003). "Oxidação mediada por radicais livres de aminoácidos livres e resíduos de aminoácidos em proteínas". Aminoácidos . 25 (3–4): 207–218. doi : 10.1007 / s00726-003-0011-2 . ISSN 0939-4451 . PMID 14661084 . S2CID 26844881 .
- ^ Morley JF, Brignull HR, Weyers JJ, Morimoto RI (2002). "O limite para a agregação de proteínas de expansão de poliglutamina e toxicidade celular é dinâmico e influenciado pelo envelhecimento em Caenorhabditiselegans" . PNAS . 99 (16): 10417–10422. Bibcode : 2002PNAS ... 9910417M . doi : 10.1073 / pnas.152161099 . PMC 124929 . PMID 12122205 .
- ^ Bednarska, Natalia G .; Schymkowitz, Joost; Rousseau, Frederic; Van Eldere, Johan (01-01-2013). "Agregação de proteínas em bactérias: a fronteira tênue entre funcionalidade e toxicidade" . Microbiologia . 159 (9): 1795-1806. doi : 10.1099 / mic.0.069575-0 . PMID 23894132 .
- ^ a b Takalo, Mari; Salminen, Antero; Soininen, Hilkka; Hiltunen, Mikko; Haapasalo, Annakaisa (08/03/2013). "Mecanismos de agregação e degradação de proteínas em doenças neurodegenerativas" . American Journal of Neurodegenerative Disease . 2 (1): 1-14. ISSN 2165-591X . PMC 3601466 . PMID 23516262 .
- ^ Garcia-Mata, Rafael; Gao, Ya-Sheng; Sztul, Elizabeth (01/06/2002). "Aborrecimentos para levar o lixo para fora: Aggresomes Aggravating" . Traffic . 3 (6): 388–396. doi : 10.1034 / j.1600-0854.2002.30602.x . ISSN 1600-0854 . PMID 12010457 . S2CID 305786 .
- ^ Gregersen, Niels; Bolund, Lars; Bross, Peter (01/10/2005). "Proteína mal dobrada, agregação e degradação na doença". Molecular Biotechnology . 31 (2): 141-150. doi : 10,1385 / MB: 31: 2: 141 . ISSN 1073-6085 . PMID 16170215 . S2CID 36403914 .
- ^ a b Mogk, Axel; Kummer, Eva; Bukau, Bernd (01/01/2015). "Cooperação de máquinas chaperonas Hsp70 e Hsp100 na desagregação de proteínas" . Fronteiras em Biociências Moleculares . 2 : 22. doi : 10,3389 / fmolb.2015.00022 . ISSN 2296-889X . PMC 4436881 . PMID 26042222 .
- ^ Liberek, Krzysztof; Lewandowska, Agnieszka; Ziętkiewicz, Szymon (23/01/2008). "Chaperones no controle da desagregação de proteínas" . The EMBO Journal . 27 (2): 328–335. doi : 10.1038 / sj.emboj.7601970 . ISSN 0261-4189 . PMC 2234349 . PMID 18216875 .
- ^ a b Chen, Bryan; Retzlaff, Marco; Roos, Thomas; Frydman, Judith (01/08/2011). "Cellular Strategies of Protein Quality Control" . Cold Spring Harbor Perspectives in Biology . 3 (8): a004374. doi : 10.1101 / cshperspect.a004374 . ISSN 1943-0264 . PMC 3140689 . PMID 21746797 .
- ^ Zhu, YJ .; Lin, H .; Lal, R. (junho de 2000). "Proteína beta amilóide fresca e não fibrilar (1-40) induz degeneração celular rápida em fibroblastos humanos envelhecidos: evidência de toxicidade celular mediada pelo canal AbetaP". FASEB J . 14 (9): 1244–54. doi : 10.1096 / fasebj.14.9.1244 . PMID 10834946 . S2CID 42263619 .
- ^ Nilsberth, C .; Westlind-Danielsson, A .; Eckman, CB .; Condron, MM .; Axelman, K .; Forsell, C .; Stenh, C .; Luthman, J .; Teplow, DB .; et al. (Setembro de 2001). "A mutação 'Arctic' APP (E693G) causa a doença de Alzheimer pela formação de protofibrila Abeta aumentada" . Nat Neurosci . 4 (9): 887–93. doi : 10.1038 / nn0901-887 . PMID 11528419 . S2CID 13516479 .
- ^ Soto C (2003). "Desdobrando o papel da proteína misfolding em doenças neurodegenerativas". Nat. Rev. Neurosci . 4 (1): 49–60. doi : 10.1038 / nrn1007 . PMID 12511861 . S2CID 205499427 .
- ^ Flagmeier P, De S, Wirthensohn DC, Lee SF, Vincke C, Muyldermans S, Knowles TPJ, Gandhi S, Dobson CM, Klenerman D (2017). "Medição ultrassensível do influxo de Ca2 + em vesículas lipídicas induzidas por agregados de proteína" . Angew. Chem. Int. Ed. Engl . 56 (27): 7750–7754. doi : 10.1002 / anie.201700966 . PMC 5615231 . PMID 28474754 .