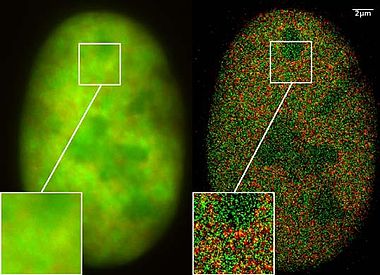
Iluminação modulada espacialmente Vertico - Vertico spatially modulated illumination
A iluminação modulada espacialmente da Vertico ( Vertico-SMI ) é o microscópio óptico mais rápido para a análise 3D de células completas na faixa nanométrica . É baseado em duas tecnologias desenvolvidas em 1996, SMI (iluminação modulada espacialmente) e SPDM (microscopia de distância de precisão espectral). A resolução óptica efetiva deste nanoscópio óptico atingiu a vizinhança de 5 nm em 2D e 40 nm em 3D, ultrapassando em muito o limite de resolução λ / 2 (cerca de 200 nm para luz azul) aplicado à microscopia padrão usando transmissão ou reflexão de luz natural (em oposição à iluminação estruturada ou fluorescência ) de acordo com o limite de resolução de Abbe Esse limite (também conhecido como limite de Rayleigh ) foi determinado por Ernst Abbe em 1873 e rege o limite de resolução alcançável de microscópios usando técnicas convencionais.
O microscópio Vertico-SMI foi desenvolvido por uma equipe liderada por Christoph Cremer , emérito da Universidade de Heidelberg , e é baseado na combinação de técnicas óticas de luz de microscopia de localização (SPDM, microscopia de distância de precisão espectral ) e iluminação estruturada (SMI, iluminação modulada espacialmente )
Desde março de 2008, muitos corantes fluorescentes padrão, como corantes fluorescentes GFP e Alexa, podem ser usados com esta microscopia de localização SPDMphymod (fluoróforos fisicamente modificáveis), para a qual apenas um único comprimento de onda de laser de intensidade adequada é suficiente para nanoimagem.
Configuração
SMI representa um tipo especial de iluminação óptica a laser (iluminação modulada espacialmente ) e Vertico reflete o arranjo vertical do eixo do microscópio que torna possível a análise de células fixas , mas também de células vivas com uma resolução óptica abaixo de 10 nanômetros (1 nanômetro = 1 nm = 1 × 10 −9 m).
Uma particularidade dessa tecnologia em comparação com as técnicas de focalização, como a microscopia 4Pi , são as exposições de amplo campo que permitem que células inteiras sejam representadas em escala nano. Essa exposição 3D de uma célula inteira com um tamanho de objeto típico de 20 µm x 20 µm requer apenas 2 minutos. Grandes exposições de campo significam que todo o objeto é iluminado e detectado simultaneamente.
Iluminação modulada espacialmente
A microscopia SMI é um processo óptico de luz da chamada função de espalhamento de ponto - engenharia. Estes são processos que modificam a função de propagação de ponto (PSF) de um microscópio de uma maneira adequada para aumentar a resolução óptica, para maximizar a precisão das medições de distância de objetos fluorescentes que são pequenos em relação ao comprimento de onda da luz iluminante, ou para extrair outros parâmetros estruturais na faixa nanométrica.
O microscópio SMI sendo desenvolvido no Instituto Kirchhoff de Física da Universidade de Heidelberg consegue isso da seguinte maneira: A intensidade de iluminação dentro do alcance do objeto não é uniforme, ao contrário dos microscópios convencionais de fluorescência de campo amplo, mas é espacialmente modulada de maneira precisa pelo uso de dois feixes de laser de interferência opostos ao longo do eixo. O princípio do campo de ondas espacialmente modulado foi desenvolvido em 1993 por Bailey et al. A abordagem de microscopia SMI usada no aplicativo Heidelberg move o objeto em etapas de alta precisão através do campo de onda, ou o próprio campo de onda é movido em relação ao objeto por mudança de fase. Isso resulta em um tamanho axial e resolução de distância aprimorados.
O SMI pode ser combinado com outras tecnologias de super resolução, por exemplo, com 3D LIMON ou LSI- TIRF como um interferômetro de reflexão interna total com iluminação estruturada lateralmente. Esta técnica SMI permitiu adquirir imagens ópticas de luz de distribuições de autofluoróforo nas seções de tecido do olho humano com resolução óptica antes incomparável. O uso de três diferentes comprimentos de onda de excitação (488, 568 e 647 nm), permite reunir informações espectrais sobre o sinal de autofluorescência. Isso tem sido usado para tratar o tecido do olho humano afetado pela degeneração macular AMD.
SPDM: microscopia de localização
Uma única e minúscula fonte de luz pode ser localizada muito melhor do que a resolução de um microscópio: embora a luz produza um ponto borrado, algoritmos de computador podem ser usados para calcular com precisão o centro do ponto borrado, levando em consideração a função de propagação do ponto do microscópio, as propriedades de ruído do detector e assim por diante. No entanto, essa abordagem não funciona quando há muitas fontes próximas umas das outras: Todas as fontes desfocam juntas.
SPDM (microscopia de distância de precisão espectral) é uma família de técnicas em microscopia de fluorescência que contorna este problema medindo apenas algumas fontes de cada vez, de modo que cada fonte é "opticamente isolada" das outras (ou seja, separada por mais do que o resolução do microscópio, normalmente ~ 200-250 nm). Então, a técnica acima (encontrar o centro de cada ponto desfocado) pode ser usada.
Se as moléculas têm uma variedade de espectros diferentes (espectros de absorção e / ou espectros de emissão), então é possível olhar para a luz de apenas algumas moléculas por vez, usando as fontes de luz e filtros apropriados. As moléculas também podem ser distinguidas de maneiras mais sutis com base no tempo de vida fluorescente e outras técnicas.
A resolução estrutural alcançável usando SPDM pode ser expressa em termos da menor distância mensurável entre dois em sua posição espacial determinada partícula puntiforme de diferentes características espectrais ("resolução topológica"). A modelagem mostrou que em condições adequadas em relação à precisão de localização, densidade de partículas, etc., a "resolução topológica" corresponde a uma " frequência espacial " que, em termos da definição clássica, é equivalente a uma resolução óptica muito melhorada.
SPDM é uma microscopia de localização que atinge uma resolução ótica efetiva várias vezes melhor do que a resolução ótica convencional (aprox. 200-250 nm), representada pela meia largura do máximo principal da função de imagem de ponto efetivo. Ao aplicar processos de precisão óptica a laser adequados, a posição e as distâncias significativamente menores do que a meia largura da função de propagação de ponto (convencionalmente 200-250 nm) podem ser medidas com precisão nanométrica entre alvos com diferentes assinaturas espectrais. Uma importante área de aplicação é a pesquisa do genoma (estudo da organização funcional do genoma ). Outra área importante de uso é a pesquisa sobre a estrutura das membranas.
Um dos fundamentos mais importantes da microscopia de localização em geral é o primeiro trabalho experimental para a localização de objetos fluorescentes em nanoescala (3D) em 1996 e prova teórica e experimental para uma precisão de localização usando luz visível na faixa de 1 nm - a base para microscopia de localização melhor do que 1/100 do comprimento de onda.
SPDMphymod: corantes fluorescentes padrão no modo piscando como GFP
Apenas nos últimos dois anos as moléculas foram usadas em estudos nanoscópicos que emitem a mesma frequência de luz espectral (mas com assinaturas espectrais diferentes com base nas características do flash), mas que podem ser ligadas e desligadas por meio da luz conforme necessário para a precisão espectral microscopia de distância. Ao combinar milhares de imagens da mesma célula, foi possível usar medições de precisão óptica a laser para gravar imagens de localização com resolução óptica significativamente melhorada. A aplicação desses novos processos de nanoscopia parecia até recentemente muito difícil porque se presumia que apenas moléculas especialmente fabricadas poderiam ser ligadas e desligadas de maneira adequada usando luz.
Em março de 2008 , o laboratório de Christoph Cremer descobriu que isso também era possível para muitos corantes fluorescentes padrão como GFP , corantes Alexa e moléculas de fluoresceína, desde que certas condições fotofísicas estejam presentes. Usando essa tecnologia chamada SPDMphymod (fluoróforos fisicamente modificáveis), um único comprimento de onda de laser de intensidade adequada é suficiente para a nanoimagem. Em contraste, outras microscopias de localização precisam de dois comprimentos de onda de laser quando moléculas especiais de fluorescência foto-comutáveis / fotoativáveis são usadas.
O gene GFP foi introduzido e expresso em muitas células procarióticas e eucarióticas e o Prêmio Nobel de Química 2008 foi concedido a Martin Chalfie , Osamu Shimomura e Roger Y. Tsien por sua descoberta e desenvolvimento da proteína verde fluorescente. A descoberta de que essas moléculas fluorescentes padrão podem ser usadas estende a aplicabilidade do método SPMD a vários campos de pesquisa em biofísica , biologia celular e medicina .
Corantes fluorescentes padrão já usados com sucesso com a tecnologia SPDMphymod: GFP, RFP, YFP, Alexa 488, Alexa 568, Alexa 647, Cy2, Cy3, Atto 488 e fluoresceína.
LIMON: microscopia de super resolução 3D
LIMON (microscopia de nanosizing microscópica leve) foi inventado em 2001 na Universidade de Heidelberg e combina microscopia de localização e iluminação espacialmente modulada à microscopia de super resolução 3D.
As imagens 3D usando Vertico-SMI são possíveis pela combinação de SMI e SPDM, onde primeiro o SMI e depois o processo SPDM é aplicado. O processo SMI determina o centro das partículas e sua propagação na direção do eixo do microscópio. Enquanto o centro das partículas / moléculas pode ser determinado com uma precisão de 1-2 nm, a propagação em torno deste ponto pode ser determinada até um diâmetro axial de aprox. 30-40 nm.
Posteriormente, a posição lateral das partículas / moléculas individuais é determinada usando SPDM, alcançando uma precisão de alguns nanômetros. Atualmente, o SPDM atinge 16 quadros / s com uma resolução efetiva de 10 nm em 2D (plano do objeto); aproximadamente 2.000 desses quadros são combinados com dados SMI (tempo de aquisição de ca. 10 seg) para atingir uma imagem tridimensional de resolução mais alta (resolução 3D ótica efetiva ca. 40-50 nm). Com uma câmera mais rápida , pode-se esperar taxas ainda mais altas (até várias centenas de quadros / seg, em desenvolvimento). Usando tintas adequadas, resoluções óticas 3D eficazes ainda mais altas devem ser possíveis
Combinando SPDMphymod com SMI (ambos inventados no laboratório de Christoph Cremer em 1996), uma reconstrução 3D de duas cores dos arranjos espaciais dos clusters Her2 / neu e Her3 foi alcançada. As posições em todas as três direções dos aglomerados de proteínas podem ser determinadas com uma precisão de cerca de 25 nm.
Uso de microscopia de super resolução na indústria
Apesar de seu uso em laboratórios biomédicos, as tecnologias de super resolução podem servir como ferramentas importantes na pesquisa farmacêutica. Eles podem ser especialmente úteis na identificação e avaliação de alvos. Por exemplo, as máquinas biomoleculares (BMM) são nanoestruturas altamente complexas que consistem em várias moléculas grandes e que são responsáveis por funções básicas nas células do corpo. Dependendo de seu status funcional, eles têm uma estrutura 3D definida. Exemplos de máquinas biomoleculares são os nucleossomos que permitem ao DNA, um transportador de informação genética com dois metros de comprimento, se dobrar nas células do corpo em um espaço de apenas alguns milionésimos de milímetro de diâmetro. Portanto, o DNA pode servir como um centro de informação e controle.
Ao usar o LIMON 3D em combinação com a marcação do complexo LIMON, é possível, pela primeira vez, tornar visíveis proteínas ou ácidos nucléicos ocultos de um complexo de moléculas 3D das chamadas máquinas biomoleculares sem destruir o complexo. Até agora, o problema na maioria dos casos era que o complexo tinha que ser destruído para uma análise detalhada das macromoléculas individuais nele. Alternativamente, modelos de simulação de computador virtual ou métodos caros de ressonância magnética nuclear foram usados para visualizar a estrutura tridimensional de tais complexos.
Literatura
links externos
- Superresolução GFP
- Lista de publicação de nanoscopia óptica
- comunicado de imprensa da Universidade de Heidelberg