Predição do gene - Gene prediction

Em biologia computacional , a predição ou descoberta de genes se refere ao processo de identificação das regiões do DNA genômico que codificam os genes . Isso inclui genes que codificam proteínas , bem como genes de RNA , mas também pode incluir a previsão de outros elementos funcionais, como regiões regulatórias . A descoberta do gene é uma das primeiras e mais importantes etapas na compreensão do genoma de uma espécie, uma vez que ela foi sequenciada .
Em seus primeiros dias, a "descoberta de genes" baseava-se em experimentos meticulosos em células e organismos vivos. A análise estatística das taxas de recombinação homóloga de vários genes diferentes poderia determinar sua ordem em um determinado cromossomo , e as informações de muitos desses experimentos poderiam ser combinadas para criar um mapa genético especificando a localização aproximada de genes conhecidos em relação uns aos outros. Hoje, com a sequência do genoma abrangente e poderosos recursos computacionais à disposição da comunidade de pesquisa, a descoberta de genes foi redefinida como um problema amplamente computacional.
A determinação de que uma sequência é funcional deve ser diferenciada da determinação da função do gene ou de seu produto. Prever a função de um gene e confirmar que a predição do gene é precisa ainda exige experimentação in vivo por meio do gene knockout e outros ensaios, embora as fronteiras da pesquisa de bioinformática estejam tornando cada vez mais possível prever a função de um gene com base apenas em sua sequência.
A previsão do gene é uma das etapas principais na anotação do genoma , após a montagem da sequência , a filtragem de regiões não codificantes e o mascaramento de repetição.
A predição do gene está intimamente relacionada ao chamado 'problema de busca de alvos', investigando como as proteínas de ligação ao DNA ( fatores de transcrição ) localizam locais de ligação específicos dentro do genoma . Muitos aspectos da predição de genes estruturais são baseados na compreensão atual dos processos bioquímicos subjacentes na célula , como transcrição de genes , tradução , interações proteína-proteína e processos de regulação , que são objeto de pesquisa ativa em vários campos ômicos , como transcriptômica , proteômica , metabolômica e, mais geralmente , genômica estrutural e funcional .
Métodos empíricos
Em sistemas empíricos (similaridade, homologia ou baseados em evidências) de localização de genes, o genoma alvo é pesquisado por sequências que são semelhantes à evidência extrínseca na forma de etiquetas de sequência expressas conhecidas , RNA mensageiro (mRNA), produtos de proteína e produtos homólogos ou sequências ortólogas. Dada uma sequência de mRNA, é trivial derivar uma sequência única de DNA genômico a partir da qual ela deve ter sido transcrita . Dada uma sequência de proteína, uma família de possíveis sequências de DNA codificantes pode ser derivada por tradução reversa do código genético . Uma vez que as sequências de DNA candidatas foram determinadas, é um problema algorítmico relativamente simples pesquisar com eficiência um genoma alvo para correspondências, completas ou parciais, e exatas ou inexatas. Dada uma sequência, algoritmos de alinhamento local, como BLAST , FASTA e Smith-Waterman, procuram regiões de similaridade entre a sequência alvo e possíveis correspondências candidatas. As correspondências podem ser completas ou parciais e exatas ou inexatas. O sucesso dessa abordagem é limitado pelo conteúdo e pela precisão do banco de dados de sequência.
Um alto grau de similaridade com um conhecido RNA mensageiro ou produto proteico é uma forte evidência de que uma região de um genoma alvo é um gene codificador de proteína. No entanto, para aplicar esta abordagem sistemicamente, é necessário um sequenciamento extensivo de mRNA e produtos proteicos. Isso não é apenas caro, mas em organismos complexos, apenas um subconjunto de todos os genes no genoma do organismo é expresso em um determinado momento, o que significa que a evidência extrínseca para muitos genes não está prontamente acessível em qualquer cultura de célula única. Assim, coletar evidências extrínsecas para a maioria ou todos os genes em um organismo complexo requer o estudo de muitas centenas ou milhares de tipos de células , o que apresenta dificuldades adicionais. Por exemplo, alguns genes humanos podem ser expressos apenas durante o desenvolvimento como embrião ou feto, o que pode ser difícil de estudar por razões éticas.
Apesar dessas dificuldades, extensas bases de dados de transcrições e sequências de proteínas foram geradas para humanos e também para outros organismos modelo importantes em biologia, como camundongos e leveduras. Por exemplo, o banco de dados RefSeq contém transcrições e sequências de proteínas de muitas espécies diferentes, e o sistema Ensembl mapeia de forma abrangente essas evidências para humanos e vários outros genomas. No entanto, é provável que esses bancos de dados estejam incompletos e contenham pequenas, mas significativas quantidades de dados errôneos.
Novas tecnologias de sequenciamento de transcriptoma de alto rendimento , como sequenciamento de RNA-Seq e ChIP, abrem oportunidades para incorporar evidências extrínsecas adicionais na previsão e validação de genes e permitem alternativas estruturalmente ricas e mais precisas aos métodos anteriores de medição da expressão do gene , como etiqueta de sequência expressa ou Microarray de DNA .
Os principais desafios envolvidos na predição de genes envolvem lidar com erros de sequenciamento em dados brutos de DNA, dependência da qualidade da montagem da sequência , manipulação de leituras curtas, mutações de frameshift , genes sobrepostos e genes incompletos.
Em procariotos, é essencial considerar a transferência horizontal de genes ao pesquisar homologia de sequência de genes. Um fator adicional importante subutilizado nas ferramentas atuais de detecção de genes é a existência de agrupamentos de genes - operons (que são unidades funcionais de DNA contendo um agrupamento de genes sob o controle de um único promotor ) em procariotos e eucariotos. Os detectores de genes mais populares tratam cada gene isoladamente, independentemente dos outros, o que não é biologicamente preciso.
Métodos ab initio
A predição do gene Ab Initio é um método intrínseco baseado no conteúdo do gene e na detecção do sinal. Por causa do custo inerente e da dificuldade na obtenção de evidências extrínsecas para muitos genes, também é necessário recorrer à descoberta de genes ab initio , em que a sequência de DNA genômico sozinha é sistematicamente pesquisada em busca de certos sinais reveladores de genes codificadores de proteínas. Esses sinais podem ser amplamente categorizados como sinais , sequências específicas que indicam a presença de um gene próximo, ou conteúdo , propriedades estatísticas da própria sequência codificadora da proteína. O achado do gene ab initio pode ser caracterizado com mais precisão como previsão do gene , uma vez que a evidência extrínseca é geralmente necessária para estabelecer de forma conclusiva que um gene putativo é funcional.
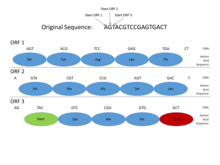
Nos genomas de procariotos , os genes têm sequências promotoras (sinais) específicas e relativamente bem compreendidas , como a caixa de Pribnow e locais de ligação a fatores de transcrição , que são fáceis de identificar sistematicamente. Além disso, a sequência que codifica para uma proteína ocorre como uma estrutura de leitura aberta contígua (ORF), que normalmente tem centenas ou milhares de pares de bases de comprimento. As estatísticas de códons de parada são tais que até mesmo encontrar um quadro de leitura aberto desse comprimento é um sinal bastante informativo. (Uma vez que 3 dos 64 códons possíveis no código genético são códons de parada, seria de se esperar um códon de parada aproximadamente a cada 20-25 códons, ou 60-75 pares de bases, em uma sequência aleatória .) Além disso, o DNA codificador de proteínas tem certos periodicidades e outras propriedades estatísticas que são fáceis de detectar em uma sequência desse comprimento. Essas características tornam a localização de genes procarióticos relativamente simples, e sistemas bem projetados são capazes de atingir altos níveis de precisão.
A descoberta do gene ab initio em eucariotos , especialmente em organismos complexos como os humanos, é consideravelmente mais desafiadora por várias razões. Em primeiro lugar, o promotor e outros sinais regulatórios nesses genomas são mais complexos e menos bem compreendidos do que nos procariotos, tornando-os mais difíceis de reconhecer de forma confiável. Dois exemplos clássicos de sinais identificados por localizadores de genes eucarióticos são ilhas CpG e locais de ligação para uma cauda poli (A) .
Em segundo lugar, os mecanismos de splicing empregados pelas células eucarióticas significam que uma sequência particular de codificação de proteína no genoma é dividida em várias partes ( exões ), separadas por sequências não codificantes ( íntrons ). (Os próprios locais de splice são outro sinal que os localizadores de genes eucarióticos costumam ser projetados para identificar.) Um gene codificador de proteína típico em humanos pode ser dividido em uma dúzia de exons, cada um com menos de duzentos pares de bases de comprimento, e alguns com até vinte a trinta. Portanto, é muito mais difícil detectar periodicidades e outras propriedades de conteúdo conhecidas de DNA codificador de proteínas em eucariotos.
Localizadores avançados de genes para genomas procarióticos e eucarióticos normalmente usam modelos probabilísticos complexos , como modelos de Markov ocultos (HMMs) para combinar informações de uma variedade de diferentes medições de sinal e conteúdo. O sistema GLIMMER é um localizador de genes amplamente utilizado e altamente preciso para procariotos. GeneMark é outra abordagem popular. Os localizadores de genes ab initio eucarióticos , em comparação, alcançaram apenas um sucesso limitado; exemplos notáveis são os programas GENSCAN e geneid . O localizador de genes SNAP é baseado em HMM como o Genscan, e tenta ser mais adaptável a diferentes organismos, abordando problemas relacionados ao uso de um localizador de genes em uma sequência do genoma contra o qual não foi treinado. Algumas abordagens recentes, como mSplicer, CONTRAST ou mGene, também usam técnicas de aprendizado de máquina , como máquinas de vetores de suporte para a previsão de genes com sucesso. Eles constroem um modelo discriminativo usando máquinas de vetores de suporte de Markov ocultos ou campos aleatórios condicionais para aprender uma função de pontuação de predição de gene precisa.
Os métodos Ab Initio foram avaliados, com alguns se aproximando de 100% de sensibilidade, no entanto, à medida que a sensibilidade aumenta, a precisão sofre como resultado do aumento de falsos positivos .
Outros sinais
Entre os sinais derivados usados para a predição estão as estatísticas resultantes das estatísticas de subseqüência , como estatísticas k-mer , Isochore (genética) ou composição / uniformidade / entropia de GC do domínio de composição, sequência e comprimento do quadro, Íntron / Exon / Doador / Aceitador / Promotor e vocabulário do sítio de ligação ribossomal , dimensão fractal , transformada de Fourier de um DNA codificado por pseudo-número, parâmetros da curva Z e certas características de execução.
Foi sugerido que sinais diferentes daqueles diretamente detectáveis nas sequências podem melhorar a previsão do gene. Por exemplo, foi relatado o papel da estrutura secundária na identificação de motivos regulatórios. Além disso, foi sugerido que a previsão da estrutura secundária do RNA ajuda a previsão do local de splice.
Redes neurais
Redes neurais artificiais são modelos computacionais que se destacam em aprendizado de máquina e reconhecimento de padrões . As redes neurais devem ser treinadas com dados de exemplo antes de serem capazes de generalizar para dados experimentais e testadas contra dados de benchmark. As redes neurais são capazes de apresentar soluções aproximadas para problemas que são difíceis de resolver algoritmicamente, desde que haja dados de treinamento suficientes. Quando aplicadas à predição de genes, as redes neurais podem ser usadas junto com outros métodos ab initio para prever ou identificar características biológicas, como locais de união. Uma abordagem envolve o uso de uma janela deslizante, que atravessa os dados da sequência de maneira sobreposta. A saída em cada posição é uma pontuação baseada em se a rede pensa que a janela contém um site de emenda doador ou um site de emenda do aceitador. Janelas maiores oferecem mais precisão, mas também requerem mais poder computacional. Uma rede neural é um exemplo de sensor de sinal, pois seu objetivo é identificar um local funcional no genoma.
Abordagens combinadas
Programas como o Maker combinam abordagens extrínsecas e ab initio mapeando dados de proteínas e EST para o genoma para validar as previsões ab initio . Augustus , que pode ser usado como parte do canal Maker, também pode incorporar dicas na forma de alinhamentos EST ou perfis de proteínas para aumentar a precisão da previsão do gene.
Abordagens de genômica comparada
Como os genomas inteiros de muitas espécies diferentes são sequenciados, uma direção promissora na pesquisa atual sobre a descoberta de genes é uma abordagem de genômica comparativa .
Isso se baseia no princípio de que as forças da seleção natural fazem com que os genes e outros elementos funcionais sofram mutações a uma taxa mais lenta do que o resto do genoma, uma vez que as mutações em elementos funcionais têm maior probabilidade de impactar negativamente o organismo do que as mutações em outros lugares. Os genes podem, portanto, ser detectados comparando os genomas de espécies relacionadas para detectar essa pressão evolutiva para a conservação. Esta abordagem foi aplicada pela primeira vez aos genomas de camundongos e humanos, usando programas como SLAM, SGP e TWINSCAN / N-SCAN e CONTRAST.
Múltiplos informantes
O TWINSCAN examinou apenas a sintenia humano-camundongo para procurar genes ortólogos. Programas como N-SCAN e CONTRAST permitiram a incorporação de alinhamentos de múltiplos organismos ou, no caso do N-SCAN, um único organismo alternativo ao alvo. O uso de vários informantes pode levar a melhorias significativas na precisão.
CONTRAST é composto por dois elementos. O primeiro é um classificador menor, identificando os locais de emenda do doador e locais de emenda do aceitador, bem como códons de início e parada. O segundo elemento envolve a construção de um modelo completo usando aprendizado de máquina. Dividir o problema em dois significa que conjuntos de dados direcionados menores podem ser usados para treinar os classificadores, e esse classificador pode operar independentemente e ser treinado com janelas menores. O modelo completo pode usar o classificador independente e não precisa desperdiçar tempo computacional ou complexidade do modelo reclassificando os limites de íntron-exon. O artigo em que CONTRAST é apresentado propõe que seu método (e os do TWINSCAN, etc.) sejam classificados como de montagem de genes de novo , usando genomas alternativos, e identificando-os como distintos de ab initio , que usam genomas 'informantes' alvo.
A descoberta comparativa de genes também pode ser usada para projetar anotações de alta qualidade de um genoma para outro. Exemplos notáveis incluem Projector, GeneWise, GeneMapper e GeMoMa. Essas técnicas agora desempenham um papel central na anotação de todos os genomas.
Previsão de pseudogene
Pseudogenes são parentes próximos de genes, compartilhando homologia de sequência muito alta, mas sendo incapazes de codificar para o mesmo produto de proteína . Embora antes relegados como subprodutos do sequenciamento de genes , cada vez mais, conforme as funções regulatórias estão sendo descobertas, eles estão se tornando alvos preditivos por si próprios. A predição de pseudogene utiliza similaridade de sequência existente e métodos ab initio, enquanto adiciona filtragem adicional e métodos de identificação de características de pseudogene.
Métodos de similaridade de sequência podem ser personalizados para previsão de pseudogenes usando filtragem adicional para encontrar pseudogenes candidatos. Isso poderia usar a detecção de desativação, que procura por mutações sem sentido ou frameshift que truncariam ou colapsariam uma sequência de codificação funcional. Além disso, traduzir DNA em sequências de proteínas pode ser mais eficaz do que apenas homologia direta de DNA.
Os sensores de conteúdo podem ser filtrados de acordo com as diferenças nas propriedades estatísticas entre pseudogenes e genes, como uma contagem reduzida de ilhas CpG em pseudogenes ou as diferenças no conteúdo de GC entre pseudogenes e seus vizinhos. Os sensores de sinal também podem ser afiados para pseudogenes, procurando a ausência de íntrons ou caudas de poliadenina.
Predição do gene metagenômico
Metagenômica é o estudo do material genético recuperado do meio ambiente, resultando em informações sequenciais de um pool de organismos. Predizer genes é útil para metagenômica comparativa .
As ferramentas de metagenômica também se enquadram nas categorias básicas de uso de abordagens de similaridade de sequência (MEGAN4) e técnicas ab initio (GLIMMER-MG).
Glimmer-MG é uma extensão do GLIMMER que se baseia principalmente em uma abordagem ab initio para encontrar genes e usando conjuntos de treinamento de organismos relacionados. A estratégia de predição é aumentada pela classificação e agrupamento de conjuntos de dados de genes antes de aplicar métodos de predição de genes ab initio. Os dados são agrupados por espécie. Este método de classificação utiliza técnicas de classificação filogenética metagenômica. Um exemplo de software para esse propósito é o Phymm, que usa modelos markov interpolados - e PhymmBL, que integra o BLAST nas rotinas de classificação.
MEGAN4 usa uma abordagem de similaridade de sequência, usando alinhamento local contra bancos de dados de sequências conhecidas, mas também tenta classificar usando informações adicionais sobre papéis funcionais, vias biológicas e enzimas. Como na predição de genes de um único organismo, as abordagens de similaridade de sequência são limitadas pelo tamanho do banco de dados.
FragGeneScan e MetaGeneAnnotator são programas populares de predição de genes baseados no modelo Hidden Markov . Esses preditores são responsáveis por erros de sequenciamento, genes parciais e funcionam para leituras curtas.
Outra ferramenta rápida e precisa para predição de genes em metagenomas é o MetaGeneMark. Esta ferramenta é usada pelo DOE Joint Genome Institute para anotar IMG / M, a maior coleção de metagenoma até hoje.
Veja também
- Lista de software de previsão de genes
- Mineração de sequência
- Previsão da função da proteína
- Pegada filogenética
- Semelhança de sequência (homologia)
links externos
- Augusto
- FGENESH
- GeMoMa - Predição de genes baseada em homologia baseada em aminoácidos e conservação da posição do íntron, bem como dados de RNA-Seq
- geneid , SGP2
- Glimmer , GlimmerHMM
- GenomeThreader
- ChemGenome
- GeneMark
- Gismo
- mGene
- StarORF - Uma ferramenta multi-plataforma e web para prever ORFs e obter sequência de complemento reverso
- Maker - um pipeline de anotação de genoma portátil e facilmente configurável
