Receptor tipo RIG-I - RIG-I-like receptor
Os receptores do tipo RIG ( receptores do tipo gene I indutíveis pelo ácido retinóico, RLRs ) são um tipo de receptor de reconhecimento de padrão intracelular envolvido no reconhecimento de vírus pelo sistema imune inato . RIG-I (gene indutível de ácido retinóico ou DDX58) é o receptor mais bem caracterizado dentro da família de receptores semelhantes a RIG-I (RLR). Junto com MDA5 (associado à diferenciação de melanoma 5) e LGP2 (laboratório de genética e fisiologia 2), esta família de receptores de reconhecimento de padrão citoplasmático (PRRs) são sentinelas para o RNA viral intracelular que é um produto de infecção viral. Os receptores RLR fornecem defesa de primeira linha contra infecções virais na maioria dos tecidos.
Ligantes RLR
O receptor RIG-I prefere se ligar a RNA de fita simples ou dupla curto (<2000 bp) carregando um trifosfato 5 'não coberto e motivos adicionais, como motivos de RNA ricos em poliuridina. RIG-I desencadeia uma resposta imune a vírus de RNA de várias famílias, incluindo os paramixovírus (por exemplo, sarampo), rabdovírus (por exemplo, vírus da estomatite vesicular) e ortomixovírus (por exemplo, influenza A ). Os ligantes de MDA5 são mal caracterizados, mas a preferência é por RNA de fita dupla longa (> 2.000 bp), como a forma replicativa de RNA de picornavírus que é encontrada em células infectadas por picornavírus. LGP2 liga-se ao RNA de fita dupla de extremidades cegas de comprimento variável e também ao MDA5 ligado ao RNA para regular a formação de filamentos . Este último está ligado ao reconhecimento de picornavírus por LGP2 (por exemplo, vírus da encefalomiocardite), de acordo com MDA5.
Características estruturais
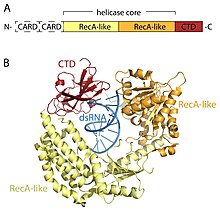
Os receptores RLR são membros da família de helicase DEAD-box (SF2) (apesar de conter um motivo DExD / H, em vez do motivo DEAD característico da família) e compartilham uma arquitetura de domínio comum. Todos contêm um núcleo helicase catalítico constituído por dois domínios semelhantes a RecA . O núcleo da helicase catalítica contém pelo menos 9 motivos de sequência altamente conservados que coordenam a ligação de ATP e RNA e a hidrólise de ATP para desenrolar o RNA. Um domínio C-terminal (CTD; InterPro : IPR021673 ) segue o núcleo da helicase e este domínio também se liga ao RNA viral. Anéis de ligação de RNA distintos dentro do CTD dos três RLRs ditam o tipo de RNA que eles podem se ligar. Além do núcleo da helicase e CTD, RIG-I e MDA5 têm dois CARD N-terminal ( domínios de recrutamento ativo de caspase ) que são essenciais para o início da sinalização a jusante. LGP2 é diferente de RIG-I e MDA5, pois não tem os domínios de sinalização CARD e, em vez disso, está implicado como um regulador positivo e negativo de RIG-I e MDA5.
Ativação de sinalização
Em células não infectadas que estão ausentes de RNA viral, RIG-I existe em uma conformação inativa na qual os domínios CARD são mascarados devido à sua interação com o CTD. Após a ligação do RNA, RIG-I muda para uma conformação na qual os domínios CARD são expostos e 'disponíveis' para sinalização. Por outro lado, os MDA5 CARDs são desimpedidos na ausência de RNA viral. Como uma salvaguarda para a ativação de RLR, os CARDs RIG-I e MDA5 expostos podem sofrer modificações pós-tradução (por exemplo , ubiquitinação , fosforilação ) que regulam positivamente ou negativamente a sinalização a jusante.
Sinalização antiviral RIG-I
No estado ativado, os domínios RIG-I CARD expostos interagem com os domínios CARD de MAVS (proteína de sinalização antiviral mitocondrial, também conhecida como IPS-1, VISA ou Cardif) que fica na superfície externa da mitocôndria . Este evento de ligação é essencial para a sinalização, pois faz com que o MAVS forme grandes agregados funcionais nos quais o TRAF3 (fator 3 associado ao receptor de TNF) e, subsequentemente, o complexo IKKε / TBK1 (I-kappa-B quinase-epsilon / TANK-binding quinase 1) são recrutados. O complexo IKKε / TBK1 leva à ativação dos fatores de transcrição do fator regulador do interferon 3 (IRF3) e IRF7 que induzem interferons do tipo I (incluindo IFNα e IFNβ ) e do tipo III (IFN). Os IFNs tipo I se ligam a receptores de IFNs tipo I na superfície da célula que os produziu, e também a outros tipos de células que expressam o receptor, para ativar a sinalização JAK-STAT (Janus quinase / transdutores de sinal e ativadores de transcrição). Isso leva à indução de centenas de genes estimulados por interferon (ISGs) que amplificam a resposta de IFN. Em geral, isso causa a morte das células infectadas, a proteção das células circundantes e a ativação da resposta imune antiviral específica do antígeno. Coletivamente, essa resposta imune antiviral coordenada controla a infecção viral.
Regulamento
Como a produção prolongada de IFN está ligada à sinalização de RLR de doenças humanas deve ser rigidamente regulada. Uma das várias maneiras de se conseguir isso é pela modificação pós-tradução, ou marcação, das proteínas de sinalização RLR do hospedeiro com fosfato (conhecido como fosforilação ) ou ubiquitina (conhecida como ubiquitinação). Essas tags também podem ser removidas, o que adiciona uma camada regulatória adicional à sinalização RLR. Essas modificações pós-traducionais e sua remoção são prevalentes na sinalização de RLR e até regulam o próprio receptor RIG-I. Mais notoriamente, o domínio RIG-I CARD é fosforilado pela proteína quinase C -α (PKC-α) e PKC-β no estado de repouso para regular negativamente a sinalização. Após a infecção viral RIG-I é desfosforilado por PP1α e PP1γ , permitindo a ubiquitinação do domínio CARD RIG-I pela ligase E3 TRIM25 para activar a resposta imunitária antiviral mediada por RLR. Dadas as modificações pós-traducionais são tão pertinentes à ativação da sinalização de RLR, não é surpreendente que elas sejam direta ou indiretamente direcionadas por vírus como influenza A e sarampo, respectivamente, para suprimir a sinalização.
Seqüestro viral de sinalização RLR
Os vírus desenvolveram maneiras de subverter a sinalização RLR para aumentar sua sobrevivência. Por exemplo, o vírus influenza A e o vírus do Nilo Ocidental (WNV) usam suas proteínas NS1 (proteína não estrutural 1) para bloquear a ubiquitinação de RIG-I por TRIM25 ou causar degradação de RIG-I, respectivamente, que por sua vez inibe a produção de IFN. Este resultado também é alcançado pela proteína NS3 / 4A da hepatite C (HCV) pela clivagem de uma parte do MAVS e da protease líder do vírus da febre aftosa (FMDV) (Lpro) que cliva o LGP2. Da mesma forma, o vírus da dengue (DENV) usa suas proteínas NS2B3, NS2A e NS4B para se ligar a IKKε e prevenir a fosforilação de IRF3 e sua proteína NS4A, de acordo com o vírus zika , para ligar MAVS para bloquear a ligação do receptor RLR. Outro exemplo proeminente é o das proteínas V de paramixovírus, que se ligam diretamente a vários RLR ou proteínas de sinalização a jusante, incluindo MDA5, LGP2 e STAT , ou proteínas como PP1α e PP1γ que regulam negativamente a sinalização de RLR.
Veja também
Referências
![]() Este artigo foi adaptado da seguinte fonte sob uma licença CC BY 4.0 ( 2019 ) ( relatórios dos revisores ):
Natalie Borg (2019). "RIG-I like receptors" (PDF) . WikiJournal of Science . 2 (1): 1. doi : 10.15347 / WJS / 2019.001 . ISSN 2470-6345 . Wikidata Q62604415 .
Este artigo foi adaptado da seguinte fonte sob uma licença CC BY 4.0 ( 2019 ) ( relatórios dos revisores ):
Natalie Borg (2019). "RIG-I like receptors" (PDF) . WikiJournal of Science . 2 (1): 1. doi : 10.15347 / WJS / 2019.001 . ISSN 2470-6345 . Wikidata Q62604415 .
links externos
- PTHR14074 : Helicase com Relacionado ao Domínio da Morte ( filtro para humanos )