Gene essencial - Essential gene
Genes essenciais são indispensáveis genes de organismos para crescer e se reproduzir descendentes sob determinado ambiente. No entanto, ser essencial depende muito das circunstâncias em que o organismo vive. Por exemplo, um gene necessário para digerir o amido só é essencial se o amido for a única fonte de energia. Recentemente, tentativas sistemáticas têm sido feitas para identificar os genes que são absolutamente necessários para manter a vida, desde que todos os nutrientes estejam disponíveis. Esses experimentos levaram à conclusão de que o número absolutamente necessário de genes para bactérias é da ordem de 250-300. Genes essenciais de organismos unicelulares codificam proteínas para três funções básicas, incluindo processamento de informações genéticas, envelopes celulares e produção de energia. Essas funções genéticas são usadas para manter um metabolismo central , replicar DNA , traduzir genes em proteínas , manter uma estrutura celular básica e mediar processos de transporte para dentro e para fora da célula. Comparados com os organismos unicelulares, os organismos multicelulares têm genes mais essenciais relacionados à comunicação e ao desenvolvimento. A maioria dos genes essenciais em vírus está relacionada ao processamento e manutenção da informação genética. Em contraste com a maioria dos organismos unicelulares, os vírus não possuem muitos genes essenciais para o metabolismo, o que os força a sequestrar o metabolismo do hospedeiro. A maioria dos genes não são essenciais, mas transmitem vantagens seletivas e aumento da aptidão . Conseqüentemente, a grande maioria dos genes não é essencial e muitos podem ser excluídos sem consequências, pelo menos na maioria das circunstâncias.
Bactérias: estudos de todo o genoma
Duas estratégias principais têm sido empregadas para identificar genes essenciais em todo o genoma: deleção direcionada de genes e mutagênese aleatória usando transposons . No primeiro caso, genes individuais anotados (ou ORFs ) são completamente deletados do genoma de forma sistemática. Na mutagênese mediada por transposons, os transposons são inseridos aleatoriamente em tantas posições em um genoma quanto possível, com o objetivo de interromper a função dos genes-alvo (ver figura abaixo). Mutantes de inserção que ainda são capazes de sobreviver ou crescer sugerem o transposon inserido em um gene que não é essencial para a sobrevivência. A localização das inserções do transposon pode ser determinada através da hibridização em microarrays ou através do sequenciamento do transposon . Um resumo dessas telas é mostrado na tabela.
| Organismo | Mutagênese | Método | Leia | ORFs | Não essencial. | Essencial | % Ess. | Notas | Ref. |
|---|---|---|---|---|---|---|---|---|---|
| Mycoplasma genitalium / pneumoniae | Aleatória | População | Sequenciamento | 482 | 130 | 265-350 | 55-73% | --- | |
| Mycoplasma genitalium | Aleatória | Clones | Sequenciamento | 482 | 100 | 382 | 79% | b, c | |
| Staphylococcus aureus WCUH29 | Aleatória | Clones | Sequenciamento | 2.600 | n / D | 168 | n / D | b, c | |
| Staphylococcus aureus RN4220 | Aleatória | Clones | Sequenciamento | 2.892 | n / D | 658 | 23% | --- | |
| Haemophilus influenzae Rd | Aleatória | População | Footprint-PCR | 1.657 | 602 | 670 | 40% | --- | |
| Streptococcus pneumoniae Rx-1 | Visadas | Clones | Formação de colônia | 2.043 | 234 | 113 | n / D | c | |
| Streptococcus pneumoniae D39 | Visadas | Clones | Formação de colônia | 2.043 | 560 | 133 | n / D | c | |
| Streptococcus pyogenes 5448 | Aleatória | Transposon | Tn-seq | 1.865 | ? | 227 | 12% | --- | |
| Streptococcus pyogenes NZ131 | Aleatória | Transposon | Tn-seq | 1.700 | ? | 241 | 14% | --- | |
| Streptococcus sanguinis SK36 | Visadas | Clones | Formação de colônia | 2.270 | 2.052 | 218 | 10% | a, j | |
| Mycobacterium tuberculosis H37Rv | Aleatória | População | Microarray | 3.989 | 2.567 | 614 | 15% | --- | |
| Mycobacterium tuberculosis | Aleatória | Transposon | ? | 3.989 | ? | 401 | 10% | --- | |
| Mycobacterium tuberculosis H37Rv | Aleatória | Transposon | Sequenciamento NG | 3.989 | ? | 774 | 19% | --- | |
| Mycobacterium tuberculosis H37Rv | Aleatória | Transposon | Sequenciamento NG | 3.989 | 3.364 | 625 | 16% | Oi | |
| Mycobacterium tuberculosis | --- | Computacional | Computacional | 3.989 | ? | 283 | 7% | --- | |
| Bacillus subtilis 168 | Visadas | Clones | Formação de colônia | 4.105 | 3.830 | 261 | 7% | a, d, g | |
| Escherichia coli K-12 MG1655 | Aleatória | População | Footprint-PCR | 4.308 | 3.126 | 620 | 14% | --- | |
| Escherichia coli K-12 MG1655 | Visadas | Clones | Formação de colônia | 4.308 | 2.001 | n / D | n / D | a, e | |
| Escherichia coli K-12 BW25113 | Visadas | Clones | Formação de colônia | 4.390 | 3.985 | 303 | 7% | uma | |
| Pseudomonas aeruginosa PAO1 | Aleatória | Clones | Sequenciamento | 5.570 | 4.783 | 678 | 12% | uma | |
| Porphyromonas gingivalis | Aleatória | Transposon | Sequenciamento | 1.990 | 1.527 | 463 | 23% | --- | |
| Pseudomonas aeruginosa PA14 | Aleatória | Clones | Sequenciamento | 5.688 | 4.469 | 335 | 6% | a, f | |
| Salmonella typhimurium | Aleatória | Clones | Sequenciamento | 4.425 | n / D | 257 | ~ 11% | b, c | |
| Helicobacter pylori G27 | Aleatória | População | Microarray | 1.576 | 1.178 | 344 | 22% | --- | |
| Corynebacterium glutamicum | Aleatória | População | ? | 3.002 | 2.352 | 650 | 22% | --- | |
| Francisella Novicida | Aleatória | Transposon | ? | 1.719 | 1.327 | 392 | 23% | --- | |
| Mycoplasma pulmonis UAB CTIP | Aleatória | Transposon | ? | 782 | 472 | 310 | 40% | --- | |
| Vibrio cholerae N16961 | Aleatória | Transposon | ? | 3.890 | ? | 779 | 20% | --- | |
| Salmonella Typhi | Aleatória | Transposon | ? | 4.646 | ? | 353 | 8% | --- | |
| Staphylococcus aureus | Aleatória | Transposon | ? | ~ 2.600 | ? | 351 | 14% | --- | |
| Caulobacter crescentus | Aleatória | Transposon | Tn-Seq | 3.876 | 3.240 | 480 | 12,2% | --- | |
| Neisseria meningitidis | Aleatória | Transposon | ? | 2.158 | ? | 585 | 27% | --- | |
| Desulfovibrio alaskensis | Aleatória | Transposon | Sequenciamento | 3.258 | 2.871 | 387 | 12% | --- |
Tabela 1. Genes essenciais em bactérias . Mutagênese : mutantes direcionados são exclusões de genes; mutantes aleatórios são inserções de transposon . Métodos : Os clones indicam deleções de um único gene, a população indica mutagênese de toda a população, por exemplo, usando transposons. Genes essenciais de triagens populacionais incluem genes essenciais para adequação (ver texto). ORFs : número de todos os quadros de leitura abertos nesse genoma. Notas : (a) coleção mutante disponível; (b) método de triagem de essencialidade direta (por exemplo, por meio de RNA antisense) que não fornece informações sobre genes não essenciais. (c) Apenas conjunto de dados parcial disponível. (d) Inclui essencialidade do gene previsto e compilação de dados de estudos publicados de essencialidade de um único gene. (e) Projeto em andamento. (f) Deduzido por comparação dos dois conjuntos de dados de essencialidade do gene obtidos independentemente nas cepas de P. aeruginosa PA14 e PAO1. (g) O resultado original de 271 genes essenciais foi corrigido para 261, com 31 genes considerados essenciais sendo na verdade não essenciais, enquanto 20 novos genes essenciais foram descritos desde então. (h) Contar genes com domínios essenciais e aqueles que levam a defeitos de crescimento quando interrompidos como essenciais, e aqueles que levam a vantagem de crescimento quando interrompidos como não essenciais. (i) Envolveu uma biblioteca mutante totalmente saturada de 14 réplicas, com 84,3% dos locais de inserção possíveis com pelo menos uma inserção de transposon. (j) Cada gene essencial foi confirmado independentemente pelo menos cinco vezes.

Com base em estudos experimentais de todo o genoma e análise de biologia de sistemas, um banco de dados de genes essenciais foi desenvolvido por Kong et al. (2019) para prever> 4000 espécies bacterianas.
Eucariotos
Em Saccharomyces cerevisiae (levedura de brotamento) 15-20% de todos os genes são essenciais. Em Schizosaccharomyces pombe (levedura de fissão) foram construídas 4.836 deleções heterozigóticas cobrindo 98,4% das 4.914 estruturas de leitura aberta que codificam para proteínas. 1.260 dessas exclusões acabaram sendo essenciais.
Rastreios semelhantes são mais difíceis de realizar em outros organismos multicelulares, incluindo mamíferos (como modelo para humanos), devido a razões técnicas, e os seus resultados são menos claros. No entanto, vários métodos foram desenvolvidos para o verme nematóide C. elegans , a mosca da fruta e o peixe-zebra (ver tabela). Um estudo recente de 900 genes de camundongos concluiu que 42% deles eram essenciais, embora os genes selecionados não fossem representativos.
Experimentos de nocaute de genes não são possíveis ou pelo menos não éticos em humanos. No entanto, as mutações naturais levaram à identificação de mutações que levam à morte embrionária precoce ou posterior. Observe que muitos genes em humanos não são absolutamente essenciais para a sobrevivência, mas podem causar doenças graves quando sofrem mutação. Essas mutações são catalogadas no banco de dados Online Mendelian Inheritance in Man (OMIM). Em uma análise computacional da variação genética e mutações em 2.472 ortólogos humanos de genes essenciais conhecidos no camundongo, Georgi et al. encontraram uma seleção forte e purificadora e níveis comparativamente reduzidos de variação de sequência, indicando que esses genes humanos também são essenciais.
Embora possa ser difícil provar que um gene é essencial em humanos, pode-se demonstrar que um gene não é essencial ou nem mesmo causa doenças. Por exemplo, o sequenciamento dos genomas de 2.636 cidadãos islandeses e a genotipagem de 101.584 indivíduos adicionais encontraram 8.041 indivíduos que tinham 1 gene completamente desativado (ou seja, essas pessoas eram homozigotas para um gene não funcional). Dos 8.041 indivíduos com nocaute completo, 6.885 foram estimados como homozigotos , 1.249 foram estimados como heterozigotos compostos (ou seja, eles tinham ambos os alelos de um gene nocauteado, mas os dois alelos tinham mutações diferentes). Nestes indivíduos, um total de 1.171 dos 19.135 genes humanos ( RefSeq ) (6,1%) foram completamente eliminados. Concluiu-se que esses 1.171 genes não são essenciais em humanos - pelo menos nenhuma doença associada foi relatada. Da mesma forma, as sequências de exoma de 3.222 adultos com herança do Paquistão britânico com alta relação parental revelaram 1.111 genótipos homozigotos de variantes raras com perda prevista da função do gene (LOF = nocautes) em 781 genes. Este estudo encontrou uma média de 140 genótipos LOF previstos (por sujeito), incluindo 16 heterozigotos raros ( frequência de alelos menores <1%), 0,34 homozigotos raros, 83,2 heterozigotos comuns e 40,6 homozigotos comuns. Quase todos os genótipos LOF homozigotos raros foram encontrados em segmentos autozigotos (94,9%). Mesmo que a maioria desses indivíduos não tivesse nenhum problema de saúde óbvio decorrente de seus genes defeituosos, é possível que problemas menores de saúde possam ser encontrados em exames mais detalhados.
Um resumo das telas de essencialidade é mostrado na tabela abaixo (principalmente com base no Banco de Dados de Genes Essenciais.
| Organismo | Método | Genes essenciais | Ref. |
| Arabidopsis thaliana | Inserção de T-DNA | 777 | |
| Caenorhabditis elegans (verme) | Interferência de RNA | 294 | |
| Danio rerio (peixe-zebra) | Mutagênese de inserção | 288 | |
| Drosophila melanogaster (mosca da fruta) | Mutagênese de inserção de elemento P | 339 | |
| Homo sapiens (humano) | Procura literária | 118 | |
| Homo sapiens (humano) | Tela baseada em CRISPR / Cas9 | 1.878 | |
| Homo sapiens (humano) | Triagem de armadilha de gene haplóide | ~ 2.000 | |
| Homo sapiens (humano) | ortólogos de rato | 2.472 | |
| Mus musculus (rato) | Procura literária | 2114 | |
| Saccharomyces cerevisiae (levedura) | Deleções de um único gene | 878 | |
| Saccharomyces cerevisiae (levedura) | Deleções de um único gene | 1.105 | |
| Schizosaccharomyces pombe (levedura) | Deleções de um único gene | 1.260 |
Vírus
Os vírus não possuem muitos genes necessários para o metabolismo, forçando-os a sequestrar o metabolismo do hospedeiro. Rastreios de genes essenciais foram realizados em alguns vírus. Por exemplo, o citomegalovírus humano (CMV) apresentou 41 ORFs essenciais, 88 não essenciais e 27 aumentantes (150 ORFs no total). A maioria dos genes essenciais e aumentadores está localizada na região central, e os genes não essenciais geralmente se agrupam perto das extremidades do genoma viral.
Tscharke e Dobson (2015) compilou uma pesquisa abrangente de genes essenciais no vírus Vaccinia e atribuiu papéis a cada uma das 223 ORFs da cepa Western Reserve (WR) e 207 ORFs da cepa Copenhagen, avaliando seu papel na replicação em cultura de células. De acordo com sua definição, um gene é considerado essencial (ou seja, tem um papel na cultura de células) se sua deleção resultar em uma diminuição no título do vírus de mais de 10 vezes em uma curva de crescimento de etapa única ou múltipla. Todos os genes envolvidos na produção do vírion envolvido, formação da cauda de actina e liberação do vírion extracelular também foram considerados essenciais. Os genes que influenciam o tamanho da placa, mas não a replicação foram definidos como não essenciais. Por esta definição 93 genes são necessários para a replicação do vírus Vaccinia em cultura de células, enquanto 108 e 94 ORFs, de WR e Copenhagen, respectivamente, não são essenciais. Os vírus vaccinia com deleções em qualquer uma das extremidades do genoma se comportaram como esperado, exibindo apenas defeitos leves ou de gama do hospedeiro. Em contraste, a combinação de deleções em ambas as extremidades do genoma para a cepa WR de VACV causou um defeito de crescimento devastador em todas as linhas de células testadas. Isso demonstra que as deleções de um único gene não são suficientes para avaliar a essencialidade dos genes e que mais genes são essenciais no vírus Vaccinia do que se pensava originalmente.
Um dos bacteriófagos selecionados para genes essenciais inclui o micobacteriófago Giles. Pelo menos 35 dos 78 genes Giles previstos (45%) não são essenciais para o crescimento lítico. 20 genes foram considerados essenciais. Um grande problema com os genes de fago é que a maioria de seus genes permanece funcionalmente desconhecida, portanto, seu papel é difícil de avaliar. Uma triagem do fago Salmonella enterica SPN3US revelou 13 genes essenciais, embora permaneça um pouco obscuro quantos genes foram realmente testados.
Análise quantitativa da essencialidade do gene
Em teoria, os genes essenciais são qualitativos. No entanto, dependendo do ambiente circundante, certos mutantes de genes essenciais podem mostrar funções parciais, que podem ser determinadas quantitativamente em alguns estudos. Por exemplo, a deleção de um gene particular pode reduzir a taxa de crescimento (ou taxa de fertilidade ou outros caracteres) para 90% do tipo selvagem. Se houver isozimas ou caminhos alternativos para os genes essenciais, eles podem ser excluídos completamente.
Letalidade sintética
Dois genes são sintéticos letais se nenhum deles for essencial, mas quando ambos são mutados, o duplo mutante é letal. Alguns estudos estimam que o número de genes letais sintéticos pode ser da ordem de 45% de todos os genes.
Genes condicionalmente essenciais

Muitos genes são essenciais apenas em certas circunstâncias. Por exemplo, se o aminoácido lisina é fornecido a uma célula, qualquer gene necessário para fazer a lisina não é essencial. No entanto, quando não há fornecimento de lisina, os genes que codificam enzimas para a biossíntese de lisina tornam-se essenciais, uma vez que nenhuma síntese de proteínas é possível sem lisina.
Streptococcus pneumoniae parece exigir 147 genes para crescimento e sobrevivência na saliva , mais do que 113-133 que foram encontrados em estudos anteriores.
A deleção de um gene pode resultar em morte ou no bloqueio da divisão celular . Embora o último caso possa implicar "sobrevivência" por algum tempo, sem divisão celular, a célula pode morrer eventualmente. Da mesma forma, em vez de divisão celular bloqueada, uma célula pode ter crescimento reduzido ou metabolismo variando de quase indetectável a quase normal. Assim, há gradiente de "essencial" a completamente não essencial, novamente dependendo da condição. Alguns autores, portanto, distinguiram entre genes " essenciais para a sobrevivência " e " essenciais para a boa forma ".
O papel do fundo genético . Semelhante às condições ambientais, o background genético pode determinar a essencialidade de um gene: um gene pode ser essencial em um indivíduo, mas não em outro, dado seu background genético. As duplicações de genes são uma explicação possível (veja abaixo).
Dependência metabólica . Os genes envolvidos em certas vias biossintéticas, como a síntese de aminoácidos , podem se tornar não essenciais se um ou mais aminoácidos forem fornecidos pelo meio de cultura ou por outro organismo. Esta é a principal razão pela qual muitos parasitas (por exemplo, Cryptosporidium hominis ) ou bactérias endossimbiônicas perderam muitos genes (por exemplo, Chlamydia ). Esses genes podem ser essenciais, mas apenas presentes no organismo hospedeiro. Por exemplo, Chlamydia trachomatis não pode sintetizar nucleotídeos de purina e pirimidina de novo , então essas bactérias são dependentes dos genes biossintéticos de nucleotídeos do hospedeiro.
Outro tipo de dependência metabólica, não relacionado a interações entre espécies, pode ser encontrado quando as bactérias são cultivadas sob condições nutricionais específicas . Por exemplo, mais de 100 genes tornam-se essenciais quando Escherichia coli é cultivada em meio com limitação de nutrientes. Especificamente, a isocitrato desidrogenase (icd) e a citrato sintase (gltA) são duas enzimas que fazem parte do ciclo do ácido tricarboxílico (TCA) . Ambos os genes são essenciais na mídia mínima M9 (que fornece apenas os nutrientes mais básicos). No entanto, quando a mídia está suplementando com 2-oxoglutarato ou glutamato , esses genes não são mais essenciais.
Duplicações de genes e vias metabólicas alternativas
Muitos genes são duplicados dentro de um genoma e muitos organismos têm diferentes vias metabólicas (via metabólica alternativa) para sintetizar os mesmos produtos. Essas duplicações ( parálogos ) e vias metabólicas alternativas geralmente tornam os genes essenciais não essenciais porque a duplicata pode substituir a cópia original. Por exemplo, o gene que codifica a enzima aspartoquinase é essencial em E. coli . Em contraste, o genoma do Bacillus subtilis contém três cópias desse gene, nenhuma das quais é essencial por si só. No entanto, uma deleção tripla de todos os três genes é letal. Nesses casos, a essencialidade de um gene ou grupo de parálogos pode muitas vezes ser prevista com base na essencialidade de um único gene essencial em uma espécie diferente. Na levedura, poucos dos genes essenciais são duplicados no genoma: 8,5% dos genes não essenciais, mas apenas 1% dos genes essenciais têm um homólogo no genoma da levedura.
No verme C. elegans , os genes não essenciais são altamente super-representados entre as duplicatas, possivelmente porque a duplicação de genes essenciais causa a superexpressão desses genes. Woods et al. descobriram que os genes não essenciais são duplicados (fixados) e perdidos com mais frequência em comparação com os genes essenciais. Em contraste, os genes essenciais são duplicados com menos frequência, mas após a duplicação bem-sucedida são mantidos por períodos mais longos.
Conservação
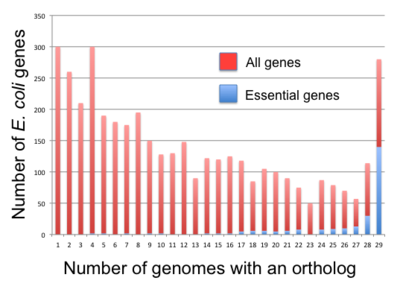
Nas bactérias , os genes essenciais parecem ser mais conservados do que os genes não essenciais, mas a correlação não é muito forte. Por exemplo, apenas 34% dos genes essenciais de B. subtilis possuem ortólogos confiáveis em todos os Firmicutes e 61% dos genes essenciais de E. coli possuem ortólogos confiáveis em todas as gama-proteobactérias . Fang et al. (2005) definiram genes persistentes como os genes presentes em mais de 85% dos genomas do clado. Eles encontraram 475 e 611 desses genes para B. subtilis e E. coli , respectivamente. Além disso, eles classificaram os genes em cinco classes de acordo com a persistência e a essencialidade: genes persistentes, genes essenciais, genes não essenciais persistentes (PNE) (276 em B. subtilis , 409 em E. coli ), genes não persistentes essenciais (ENP) (73 em B . subtilis , 33 em E. coli ), e genes não essenciais não persistentes (NPNE) (3.558 em B. subtilis , 3.525 em E. coli ). Fang et al. encontraram 257 genes persistentes, que existem tanto em B. subtilis (para Firmicutes) e E. coli (para Gamma-proteobactérias). Entre estes, 144 (respectivamente 139) foram previamente identificados como essenciais em B. subtilis (respectivamente E. coli ) e 25 (respectivamente 18) dos 257 genes não estão presentes no B. subtilis 475 (respectivamente 611 E. coli) persistente genes. Todos os outros membros do pool são genes PNE.
Em eucariotos , 83% dos ortólogos um-para-um entre Schizosaccharomyces pombe e Saccharomyces cerevisiae conservaram a essencialidade, ou seja, não são essenciais em ambas as espécies ou são essenciais em ambas as espécies. Os 17% restantes dos genes não são essenciais em uma espécie e são essenciais na outra. Isso é bastante notável, dado que S. pombe está separado de S. cerevisiae por aproximadamente 400 milhões de anos de evolução.
Em geral, genes altamente conservados e, portanto, mais antigos (ou seja, genes com origem filogenética anterior) têm maior probabilidade de ser essenciais do que genes mais jovens - mesmo que tenham sido duplicados.
Estude
O estudo experimental de genes essenciais é limitado pelo fato de que, por definição, a inativação de um gene essencial é letal para o organismo. Portanto, eles não podem ser simplesmente excluídos ou transformados para analisar os fenótipos resultantes (uma técnica comum em genética ).
Existem, no entanto, algumas circunstâncias em que genes essenciais podem ser manipulados. Em organismos diplóides , apenas uma única cópia funcional de alguns genes essenciais pode ser necessária ( haplosuficiência ), com o heterozigoto exibindo um fenótipo instrutivo. Alguns genes essenciais podem tolerar mutações que são deletérias, mas não totalmente letais, uma vez que não abolem completamente a função do gene.
A análise computacional pode revelar muitas propriedades das proteínas sem analisá-las experimentalmente, por exemplo, olhando para proteínas homólogas , função, estrutura etc. (veja também abaixo, Predição de genes essenciais ). Os produtos de genes essenciais também podem ser estudados quando expressos em outros organismos , ou quando purificados e estudados in vitro .
Genes condicionalmente essenciais são mais fáceis de estudar. Foram identificadas variantes sensíveis à temperatura de genes essenciais que codificam produtos que perdem função em altas temperaturas e, portanto, só mostram um fenótipo em temperatura elevada.
Reprodutibilidade
Se as triagens de genes essenciais forem repetidas em laboratórios independentes, muitas vezes resultam em listas de genes diferentes. Por exemplo, as telas em E. coli produziram de ~ 300 a ~ 600 genes essenciais (ver Tabela 1 ). Essas diferenças são ainda mais pronunciadas quando diferentes cepas bacterianas são usadas (ver Figura 2 ). Uma explicação comum é que as condições experimentais são diferentes ou que a natureza da mutação pode ser diferente (por exemplo, uma deleção completa do gene vs. um transposon mutante). Os rastreios de transposões, em particular, são difíceis de reproduzir, dado que um transposão pode inserir-se em muitas posições dentro de um gene. As inserções em direção à extremidade 3 'de um gene essencial podem não ter um fenótipo letal (ou nenhum fenótipo) e, portanto, podem não ser reconhecidas como tal. Isso pode levar a anotações errôneas (aqui: falsos negativos).
Comparação de telas CRISPR / cas9 e RNAi . Telas para identificar genes essenciais na linha celular de leucemia mielóide crônica humana K562 com esses dois métodos mostraram apenas sobreposição limitada. A uma taxa de falsos positivos de 10%, havia ~ 4.500 genes identificados na triagem Cas9 contra ~ 3.100 na triagem shRNA , com apenas ~ 1.200 genes identificados em ambos.
Diferentes genes essenciais em diferentes organismos
Organismos diferentes podem ter genes essenciais diferentes. Por exemplo, Bacillus subtilis tem 271 genes essenciais. Cerca de metade (150) dos genes ortólogos em E. coli também são essenciais. Outros 67 genes que são essenciais em E. coli não são essenciais em B. subtilis , enquanto 86 genes essenciais de E. coli não têm nenhum ortólogo de B. subtilis . Em Mycoplasma genitalium, pelo menos 18 genes são essenciais que não são essenciais em M. bovis. Muitos desses diferentes genes essenciais são causados por parálogos ou vias metabólicas alternativas.
Esses diferentes genes essenciais em bactérias podem ser usados para desenvolver terapias antibacterianas direcionadas contra certos patógenos específicos para reduzir a resistência aos antibióticos na era do microbioma. Stone et al (2015) usaram a diferença em genes essenciais em bactérias para desenvolver drogas seletivas contra o patógeno oral Porphyromonas gingivalis , ao invés da bactéria benéfica Streptococcus sanguis .
Predição
Genes essenciais podem ser previstos computacionalmente. No entanto, a maioria dos métodos usa dados experimentais ("conjuntos de treinamento") até certo ponto. Chen et al. determinou quatro critérios para selecionar conjuntos de treinamento para tais previsões: (1) genes essenciais no conjunto de treinamento selecionado devem ser confiáveis; (2) as condições de crescimento nas quais os genes essenciais são definidos devem ser consistentes nos conjuntos de treinamento e previsão; (3) as espécies usadas como conjunto de treinamento devem estar intimamente relacionadas ao organismo-alvo; e (4) organismos usados como conjuntos de treinamento e predição devem exibir fenótipos ou estilos de vida semelhantes. Eles também descobriram que o tamanho do conjunto de treinamento deve ser de pelo menos 10% do total de genes para produzir previsões precisas. Algumas abordagens para prever genes essenciais são:
Genômica comparada . Logo após os primeiros genomas (de Haemophilus influenzae e Mycoplasma genitalium ) se tornarem disponíveis, Mushegian et al. tentou prever o número de genes essenciais com base em genes comuns nessas duas espécies. Supôs-se que apenas genes essenciais deveriam ser conservados ao longo da longa distância evolutiva que separava as duas bactérias. Este estudo identificou aproximadamente 250 genes essenciais candidatos. À medida que mais genomas se tornavam disponíveis, o número de genes essenciais previstos continuava diminuindo porque mais genomas compartilhavam cada vez menos genes. Como consequência, concluiu-se que o núcleo conservado universal consiste em menos de 40 genes. No entanto, esse conjunto de genes conservados não é idêntico ao conjunto de genes essenciais, pois diferentes espécies dependem de diferentes genes essenciais.
Uma abordagem semelhante foi usada para inferir genes essenciais do pan-genoma de espécies de Brucella . 42 genomas completos de Brucella e um total de 132.143 genes codificadores de proteínas foram usados para prever 1.252 genes essenciais potenciais, derivados do genoma central, em comparação com um banco de dados procariotos de genes essenciais.
Análise de rede . Depois que as primeiras redes de interação de proteínas de levedura foram publicadas, descobriu-se que proteínas altamente conectadas (por exemplo, por interações proteína-proteína ) têm maior probabilidade de ser essenciais. No entanto, proteínas altamente conectadas podem ser artefatos experimentais e alta conectividade pode representar pleiotropia em vez de essencialidade. No entanto, os métodos de rede foram aprimorados com a adição de outros critérios e, portanto, têm algum valor na previsão de genes essenciais.
Aprendizado de máquina . Hua et al. usou o aprendizado de máquina para prever genes essenciais em 25 espécies bacterianas .
Índice de Hurst . Liu et al. (2015) usaram o expoente de Hurst , um parâmetro característico para descrever a correlação de longo alcance no DNA para prever genes essenciais. Em 31 de 33 genomas bacterianos, os níveis de significância dos expoentes de Hurst dos genes essenciais foram significativamente maiores do que para o conjunto de genes completo correspondente, enquanto os níveis de significância dos expoentes de Hurst dos genes não essenciais permaneceram inalterados ou aumentaram apenas ligeiramente.
Genomas mínimos . Também se pensava que genes essenciais poderiam ser inferidos de genomas mínimos que supostamente contêm apenas genes essenciais. O problema aqui é que os menores genomas pertencem a espécies parasitas (ou simbiônicas) que podem sobreviver com um conjunto de genes reduzido à medida que obtêm muitos nutrientes de seus hospedeiros. Por exemplo, um dos menores genomas é o de Hodgkinia cicadicola , um simbionte das cigarras, contendo apenas 144 Kb de DNA que codifica apenas 188 genes. Como outros simbiontes, Hodgkinia recebe muitos de seus nutrientes de seu hospedeiro, então seus genes não precisam ser essenciais.
Modelagem metabólica . Genes essenciais também podem ser previstos em genomas completamente sequenciados por reconstrução metabólica , isto é, reconstruindo o metabolismo completo a partir do conteúdo do gene e, em seguida, identificando os genes e vias que foram consideradas essenciais em outras espécies. No entanto, esse método pode ser comprometido por proteínas de função desconhecida. Além disso, muitos organismos têm vias de apoio ou alternativas que devem ser levadas em consideração (ver figura 1). A modelagem metabólica também foi usada por Basler (2015) para desenvolver um método para prever genes metabólicos essenciais. A análise de equilíbrio de fluxo , um método de modelagem metabólica, foi recentemente usada para prever genes essenciais no metabolismo do carcinoma de células renais de células claras.
Genes de função desconhecida . Surpreendentemente, um número significativo de genes essenciais não tem função conhecida. Por exemplo, entre os 385 candidatos essenciais em M. genitalium , nenhuma função pode ser atribuída a 95 genes, embora esse número tenha sido reduzido para 75 em 2011. A maioria dos genes funcionalmente essenciais desconhecidos têm funções biológicas potenciais relacionadas a um dos três fundamentais funções.
ZUPLS . Song et al. apresentou um novo método para prever genes essenciais que usa apenas a curva Z e outros recursos baseados em sequência. Tais características podem ser calculadas prontamente a partir das sequências de DNA / aminoácidos. No entanto, a confiabilidade desse método permanece um pouco obscura.
Servidores essenciais de previsão de genes . Guo et al. (2015) desenvolveram três serviços online para prever genes essenciais em genomas bacterianos. Essas ferramentas disponíveis gratuitamente são aplicáveis para sequências de genes únicos sem funções anotadas, genes únicos com nomes definidos e genomas completos de cepas bacterianas. Kong et al. (2019) desenvolveram o banco de dados ePath , que pode ser usado para pesquisar mais de 4000 espécies bacterianas para predizer genes essenciais.
Domínios de proteínas essenciais
Embora a maioria dos genes essenciais codifique proteínas, muitas proteínas essenciais consistem em um único domínio. Este fato tem sido usado para identificar domínios de proteínas essenciais. Goodacre et al. identificaram centenas de domínios essenciais de função desconhecida (eDUFs). Lu et al. apresentou uma abordagem semelhante e identificou 3.450 domínios que são essenciais em pelo menos uma espécie microbiana.
Veja também
- Aminoácido essencial
- Proteínas essenciais em complexos de proteínas
- Gene
- Genoma
- Genoma mínimo
- Mutação
Referências
Leitura adicional
- Gao F, Luo H, Zhang CT, Zhang R (2015). "Análise de essencialidade de genes baseada em DEG 10, um banco de dados atualizado de genes essenciais". Gene Essentiality . Methods in Molecular Biology. 1279 . pp. 219–33. doi : 10.1007 / 978-1-4939-2398-4_14 . ISBN 978-1-4939-2397-7. PMID 25636622 .
- Long JL, ed. (2015). Gene Essentiality - Springer Methods and Protocols . Methods in Molecular Biology. 1279 . Humana Press. p. 248. doi : 10.1007 / 978-1-4939-2398-4 . ISBN 978-1-4939-2397-7. S2CID 27547825 .
links externos
- Banco de dados de genes essenciais
- OGEE: banco de dados de essencialidade online
- Banco de dados de EGGS (Essential Genes on Genome Scale)
- Banco de dados ePath (genes essenciais no caminho)
- Genes essenciais em E. coli (EcoliWiki )
- Genes essenciais em E. coli (Ecogene)
- Benjamin Lewin 's Essential Genes (livro didático), Pearson / Prentice-Hall .
