POLD1 - POLD1
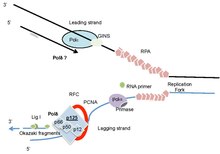
O gene polimerase delta 1 ( POLD1 ) codifica a grande subunidade catalítica POLD1 / p125 do complexo DNA polimerase delta (Polδ). A enzima Polδ é responsável por sintetizar a fita atrasada de DNA e também foi implicada em algumas atividades na fita principal (Figura 1). A subunidade POLD1 / p125 codifica os domínios de polimerização de DNA e exonuclease , que fornecem à proteína uma segunda função importante na revisão para garantir a precisão da replicação durante a síntese de DNA e em vários tipos de reparo de DNA ligado à replicação após dano ao DNA. Mutações da linha germinativa que prejudicam a atividade de POLD1 foram implicadas em vários tipos de câncer hereditário, em alguns cânceres esporádicos e em uma síndrome de desenvolvimento de envelhecimento prematuro, hipoplasia mandibular , surdez e características progeróides e lipodistrofia ( síndrome MDPL / MDP ). Estudos de POLD1 enfatizam a importância de manter a estabilidade genômica para limitar a tumorigênese . Atualmente não está claro se a tumorigênese aumentada associada a defeitos de POLD1 é o resultado de substituições de bases aumentadas ou devido ao colapso do garfo e produção de quebras de fita dupla de DNA (DSBs). Revisões recentes abordaram funções importantes de POLD1 e Polδ.
Descoberta
A primeira DNA polimerase, DNA polimerase I , foi descoberta por Arthur Kornberg e seus colegas em 1956, revisada em. Em 1976, Byrnes et al. descobriram uma terceira atividade da DNA polimerase em células de mamíferos, chamada de polimerase delta (δ). Foi purificado a partir de medula óssea hiperplásica eritróide de coelho e descrito como uma DNA polimerase que possuía uma atividade de exonuclease intrínseca 3 'a 5'. Uma função de revisão de exonuclease 3'-5 'para DNA polimerases ( E. coli ) foi descrita pela primeira vez 4 anos antes por Kornberg e Brutlag, revisado em. O DNA Polδ humano é um heterotetrâmero . As quatro subunidades são: (POLD1 / p125), ( POLD3 / p66), ( POLD2 / p50 ) e ( POLD4 / p12), com os nomes alternativos refletindo os pesos moleculares expressos em quilodaltons (kDa). A subunidade catalítica da polimerase foi identificada como o polipeptídeo de 125 kDa por coloração de atividade em 1991. Vários grupos clonaram independentemente os cDNAs de POLD1 humano e murino. Após sua purificação de várias fontes, incluindo timo de bezerro, placenta humana e células HeLa, sua atividade foi implicada no reparo do DNA.
Gene
Polimerase (DNA) delta 1, subunidade catalítica e POLD1 são o nome e o símbolo do gene aprovados pelo Comitê de Nomenclatura Genética (HGNC) da Organização do Genoma Humano (HUGO). POLD1 também é conhecido como CDC2, MDPL, POLD e CRCS10), tem ~ 34 kb de comprimento e sua localização citogenética é o cromossomo 19 q13.33. A localização precisa, na montagem GRCh38.p2, é do par de bases 50.384.290 ao par de bases 50.418.018 no cromossomo 19. O ortólogo de camundongo mapeia para o cromossomo 7. Em humanos, o principal transcrito POLD1 (NM_002691.3) contém 27 exons e se traduz nos 1107 aminoácidos da subunidade p125 ou A. Uma isoforma mais longa foi relatada com uma inserção in-frame de 26 aminoácidos após o aminoácido 592 (NP_001295561.1). Um pseudogene (LOC100422453) foi relatado no braço longo do cromossomo 6. A Tabela 1 fornece nomes de genes e localizações cromossômicas para as várias subunidades de Polδ em humanos, camundongos, levedura de brotamento ( S. cerevisiae ) e levedura de fissão ( S. pombe ) .
O promotor do gene POLD1 é regulado através da maquinaria do ciclo celular e a expressão de mRNA de POLD1 atinge um pico no final da fase G1 / S durante a replicação do DNA. O promotor POLD1 é rico em G / C e não possui TATA box . A transcrição deste promotor contendo a caixa GC é regulada por fatores de transcrição relacionados a Sp1 e Sp1, como Sp3 , com sua ligação mediada por sequências de ligação repetidas de 11 pb. O promotor POLD1 contém uma sequência semelhante a E2F localizada perto do principal local de início da transcrição . Outro elemento regulador, a região de homologia de elementos do ciclo celular / genes do ciclo celular (CDE / CHR), localizada a jusante do local de início, é importante para a transcrição de POLD1 na fase G2 / M pelas proteínas E2F1 e p21 . P53 regula a transcrição de POLD1 por ativação indireta dependente de p21 de uma via p53-p21-DREAM-CDE / CHR. Um estudo relatou que a proteína supressora de tumor p53 compete com Sp1 pela ligação ao promotor POLD1 . Um microARN (MIR), o miR-155 , regula negativamente POLD1 indirectamente pela supressão do factor de transcrição FOXO3a , que tem sios de ligao putativos na POLD1 promotor (RTMAAYA; elemento de resposta).
Proteína
POLD1 / p125 tem uma dobra comum da família B, semelhante a outras DNA polimerases (Polα e ε). POLD1 / p125 humano tem um sinal de localização nuclear putativo na extremidade N-terminal (resíduos 4-19). Os resíduos 304-533 contêm o domínio de exonuclease (Figura 2), enquanto os resíduos 579-974 contêm o domínio de polimerase. O domínio de exonuclease é um domínio semelhante a DnaQ do tipo DEDDy comum à família da B-DNA polimerase . Este domínio tem uma estrutura em grampo beta que ajuda a alternar entre os locais ativos da polimerase e da exonuclease em caso de incorporação incorreta de nucleotídeos.
Motivos A e C, que são os mais conservados do domínio da polimerase. Estes têm 2 aspartatos catalíticos , no motivo A (DXXLYPS, D602) e no motivo C (DTDS, D757) que se ligam ao cálcio no sítio ativo. O motivo A tem 11 aminoácidos que são importantes na incorporação de nucleotídeos e na formação da ligação fosfodiéster .
A tirosina Y701 funciona de forma semelhante à tirosina Y567 no bacteriófago ortólogo RB69 como a porta estérica de açúcar que impede a incorporação de ribonucleotídeos. Um motivo LXCXE (711 a 715) medeia a ligação a pRB durante a fase G1 do ciclo celular. O domínio da polimerase também tem um motivo KKRY altamente conservado (resíduos 806 a 809) que é importante para a função de ligação e catalítica. POLD1 pode ser direcionado ao nucléolo após acidificação por meio de um motivo de sequência de detenção nucleolar (NoDS) representado por pequenos motivos de sequência dispersos por toda a região de codificação da proteína. O domínio C-terminal tem dois motivos conservados de ligação a metal ricos em cisteína (CysA e CysB) (de 1012 e 1083) necessários para a ligação do Antígeno Nuclear Celular em Proliferação (PCNA) e recrutamento de subunidades acessórias, respectivamente. CysB coordena um cluster [4Fe-4S] adicionado por meio do Conjunto de proteína Citosólica Ferro-enxofre (CIA), que requer a função da máquina de montagem do Cluster de Enxofre de Ferro (ISC) mitocondrial. O processo de maturação é mediado pelo complexo de alvejamento central CIA1-CIA2B / FAM96B- MMS19 , que interage com a apoproteína para garantir a inserção específica do cluster Fe-S.

Estudos de ligação e associação demonstraram que POLD2 está estreitamente associado a POLD1; POLD3 e POLD2 interagem entre si e POLD4 interage com POLD1 e POLD2. Heterotetrâmero Polδ reconstituído por coexpressão de subunidades em células Sf9 tinha propriedades semelhantes às Polδ purificado do timo de bezerro, e a holoenzima completa foi fortemente estimulada por PCNA. Numerosos estudos demonstraram que, embora POLD1 possua a polimerase e a atividade de revisão da exonuclease 3'-5 ', as outras subunidades aumentam essas atividades, habilidades de ligação ao DNA e interações funcionalmente importantes com PCNA e seu Fator de Replicação C (RFC). A holoenzima DNA Polδ é frequentemente considerada como incluindo PCNA e RFC, bem como as quatro subunidades do complexo de polimerase (Figura 1).
Uma série de outros estudos e triagens identificaram parceiros de interação adicionais relevantes para funções na replicação e reparo do DNA. A Figura 3 mostra uma matriz de interações estabelecidas e putativas durante a replicação e o reparo, que podem ser acessadas posteriormente por meio de e. Um site da Vanderbilt University fornece interação adicional sobre a importante estrutura da proteína POLD1 e várias classes de interação de genes e proteínas, com base em critérios como coocorrência em uma interação física direta complexa, relacionamento regulatório e coexpressão.
| Subunidades delta da polimerase |
Nome da proteína em humanos |
Homo sapiens | Mus musculus | Saccharomyces cerevisiae | Schizosaccharomyces pombe |
|---|---|---|---|---|---|
| A (catalítico) | p125 | POLD1-Chr 19q13.3 | Pold1-Chr 7B4 | POL3-Chr IV | cdc6-Chr II |
| B (acessório) | p50 | POLD2-Chr 7p13 | Pold2-Chr 11A2 | POL31-Chr X | cdc1-Chr I |
| C (acessório) | p66 | POLD3-Chr 11q14 | Pold3-Chr 7F1 | POL32-Chr X | cdc27-Chr II |
| D (acessório) | p12 | POLD4-Chr 11q13 | Pold4-Chr 19A | - | cdm1-Chr II |
| Tabela 1: Nomes de genes e localizações cromossômicas para as várias subunidades da polimerase delta em levedura humana, de camundongo, de brotamento e de fissão. | |||||
Expressão e regulação

A proteína POLD1 / P125 é expressa de forma ubíqua em um painel de tecidos humanos com altos níveis nos tecidos do coração e pulmão. A localização subcelular de POLD1 / p125 é predominantemente no núcleo e no nucleoplasma .
Uma redução em POLD1 / p125 foi observada em fibroblastos de pele humana senescentes e em linfócitos de uma população idosa. A expressão de POLD1 / p125 é regulada epigeneticamente em resposta a danos no DNA. Outros estudos também mostraram que a expressão de POLD1 / p125 é regulada por miR-155, p53 e pelo RNA não codificante longo , PVT1 . Na presença de dano ao DNA ou estresse de replicação ( luz ultravioleta , metanossulfonato de metila , hidroxiureia ou afidicolina ), a subunidade POLD4 / p12 é rapidamente degradada. As atividades catalíticas de p125 são diferentes quer esteja no heterotetrâmero (Polδ4, com p12) ou no heterotrímero (Polδ3, sem p12). A produção do heterotrímero depende da degradação de p12 pela ligase E3 RNF8 , uma proteína envolvida no reparo de DSBs e possivelmente na recombinação homóloga (HR). Além disso, a E3 ligase CRL4 Cdt2 pode degradar POLD4 / p12 durante a replicação normal do DNA e na presença de danos ao DNA. POLD4 / p12 também pode ser degradado pela protease µ-calpaína, que está envolvida na apoptose desencadeada pelo cálcio .
POLD1 / p125 tem um domínio NoDS que regula o transporte para o nucléolo em resposta à acidose. O transporte nucleolar requer uma interação direta entre a subunidade p50 e a proteína WRN . Durante a resposta ao dano ao DNA, o WRN se move para fora do nucléolo e, assim, libera Polδ. POLD1 / p125 também demonstrou interagir com PDIP46 / SKAR e LMO2 .
Função
Replicação de DNA
A replicação do DNA é um processo altamente organizado que envolve muitas enzimas e proteínas, incluindo várias DNA polimerases. A principal atividade replicativa na fase S do ciclo celular depende de três DNA polimerases - Polimerase alfa (Polα), Polimerase delta (Polδ) e Polimerase epsilon (Polε). Após o início da síntese de DNA por Polα, Polδ ou Polε executam a síntese de cadeia principal e retardada, respectivamente. Essas polimerases mantêm uma fidelidade muito alta, que é garantida pelo emparelhamento de bases de Watson-Crick e pela atividade 3'-exonuclease (ou revisão). Estudos recentes afirmam que Polδ pode sintetizar a fita principal. Como essas polimerases funcionam, em relação a outros fatores envolvidos na replicação, é de grande interesse, pois provavelmente explica a paisagem mutacional que elas produzem quando defeituosas. A manutenção da fidelidade de replicação é um equilíbrio fino entre os erros únicos das polimerases δ e ε, o equilíbrio entre a revisão e MMR e a distinção no processamento de ribonucleotídeos entre as duas fitas. Estudos extensivos em modelos de levedura mostraram que mutações no domínio de exonuclease de Polδ e homólogos de Polε podem causar um fenótipo mutador , revisado em. O DNA de fita simples (ss) sintetizado durante a síntese de fita retardada pode ser direcionado por agentes danificadores de ss-DNA também como é um alvo seletivo para mutações APOBEC . Proteínas de ligação de DNA que rapidamente se reassociam pós-replicação evitam que Polδ repare os erros produzidos por Polα na fita atrasada madura. Estudos de levedura mostraram que Polδ pode corrigir erros de Polε na fita principal.
Reparo de DNA
A atividade POLD1 contribui para vários processos de reparo de DNA evolutivamente conservados , incluindo reparo de incompatibilidade (MMR), síntese de translesão (TLS), reparo de excisão de base (BER), reparo de excisão de nucleotídeo (NER) e reparo de quebra de fita dupla (DSB). POLD1 medeia as etapas pós-incisão em BER, NER e MMR. Polδ interage com a maquinaria MMR para dar suporte à revisão pós-replicação do DNA recém-sintetizado, com células portadoras de mutações que inativam os componentes POLD1 e MMR experimentando taxas de mutação elevadas. Como observado acima, um heterotrímero Polδ (Polδ3) torna-se a forma oligomérica dominante de POLD1 e é ativo durante a presença de dano ao DNA. Polδ3 é menos sujeito a erros do que (Polδ4) e pode discriminar melhor entre pares incompatíveis, associado a uma melhor atividade de revisão: no entanto, tem capacidade reduzida de contornar algumas lesões de base. Em vez disso, a troca da polimerase Polδ para a polimerase zeta especializada (Polζ) é importante para TLS, pois a substituição de p125 pela subunidade catalítica Polζ, p353, permite uma melhor atividade de desvio. Neste processo, o domínio C-terminal altamente conservado (CTD) de POLD1 / p125 interage com o domínio CTD de Polζ, e os aglomerados de ferro dentro de cada CTD medeiam interações envolvendo a ligação a POLD2 que permite a troca de polimerase durante TLS. Alguns estudos recentes sugerem que uma mudança de Polδ para Pol lambda (λ) também apóia o TLS e o reparo de danos oxidativos ao DNA, como lesões de 7,8-Diidro-8-oxoguanina .
A depleção de POLD1 pode interromper o ciclo celular nas fases G1 e G2 / M em células humanas. O bloqueio do ciclo celular nessas fases normalmente indica a presença de danos ao DNA e a ativação de pontos de verificação de danos ao DNA. As células depletadas de POLD1 são sensíveis à inibição das quinases de ponto de verificação de dano ao DNA ATR e CHK1 . Em S. pombe , os mecanismos de HR podem reiniciar bifurcações de replicação paralisadas utilizando a atividade de síntese de fita Polδ, mas tal reinicialização mediada por HR não paralela é muito propensa a erros, podendo levar a um aumento da instabilidade genômica. Polδ interage estrutural e funcionalmente com a proteína WRN, e WRN recruta Polδ para o nucléolo. O gene WRN sofre mutação na síndrome de Werner (um distúrbio autossômico recessivo ), levando ao envelhecimento acelerado e ao aumento da instabilidade genética. A interação com WRN aumenta a processabilidade de Polδ de uma maneira independente do PCNA. Por meio dessas interações, o WRN impacta diretamente a replicação-reparo do DNA e auxilia na síntese mediada por Polδ.
Significado clínico
Câncer
Demonstrou-se que as proteínas de reparo do DNA são importantes em doenças humanas, incluindo o câncer. Por exemplo, mutações da linha germinativa em proteínas de reparo de DNA envolvidas em MMR (MSH2, MLH1, MSH6 e PMS2) foram descritas na síndrome de Lynch (LS), que é caracterizada pela presença de instabilidade de microssatélites (MSI). Mais recentemente, mutações da linha germinativa foram relatadas nos domínios de exonuclease de POLD1 e POLE , a subunidade catalítica de Polε. Essas mutações estão associadas a polipose oligo-adenomatosa, câncer colorretal de início precoce (CRC), câncer endometrial (EDMC), câncer de mama e tumores cerebrais . ( Revisado em) A maioria das mutações POLD1 relatadas ligadas ao câncer estão presentes na exonuclease domínio. Em contraste com LS, os tumores com mutação POLD1 são microssatélites estáveis. Alguns dados sugerem a ideia de que os tumores POLD1 estão associados a mutações condutoras em genes, incluindo APC e KRAS . A mutação missense POLD1 p. S478N, no domínio da exonuclease, foi validado como prejudicial e patogênico. Outras variantes de POLD1 foram clinicamente identificadas que foram previstas como prejudiciais e estão atualmente sob investigação adicional (por exemplo, p. D316H, p. D316G, p. R409W, p. L474P e p. P327L).
Em pacientes pediátricos, mutações de duplo acerto em POLD1 ou PÓLO e deficiência de reparo de incompatibilidade bialélica (bMMRD) levam a fenótipos de tumor ultra-hipermutado. Fenótipos como ultra-hipermutação em tumores podem indicar melhor resposta a novas terapêuticas contra o câncer em desenvolvimento, embora isso precise de avaliação direta para POLD1 . Bouffet et al. relatam dois irmãos com bMMRD- glioblastoma multiforme que têm mutações somáticas no PÓLO (P436H em um, S461P no outro), e mostrou uma resposta duradoura a um ensaio clínico com o anti- morte programada-1 inibidor nivolumab . Mutações POLD1 foram estudadas em linhas celulares e modelos de camundongos. Por exemplo, uma mutação Polδ homozigótica em camundongos que interrompe a função enzimática leva a uma incidência de câncer altamente elevada.
MDPL
Mutações prejudiciais em POLD1 também foram observadas em pacientes com uma síndrome conhecida como hipoplasia mandibular, surdez e características progeróides com síndrome de lipodistrofia (MDPL / MDP) (# 615381 no banco de dados Online Mendelian Inheritance in Man (OMIM)). Esta é uma síndrome muito rara e poucos estudos descrevendo mutações foram relatados. As mutações que foram observadas estão nas regiões que afetam o domínio da exonuclease e os domínios da polimerase. Cinco casos de novo não relacionados foram descritos com a mesma variante heterozigótica , c.1812_1814delCTC p.Ser605del (rs398122386). S605 está no motivo A altamente conservado do sítio ativo da polimerase. Esta variante não inibe a atividade de ligação ao DNA, mas afeta a catálise. Outra variante foi relatada em um paciente separado (p.R507C). Esta variante está localizada no domínio ExoIII altamente conservado e ainda não foi completamente caracterizada.
Variantes de POLD1 Ser605del e R507C também foram identificadas em um subconjunto de pacientes com síndrome de Werner atípica (AWS). Após o teste molecular, esses pacientes foram reclassificados como pacientes com MDPL / MDP. MDPL / MDP, AWS e síndrome de Werner, todos apresentam progéria . Um primeiro exemplo de transmissão germinativa foi observado em uma mãe e um filho com a mutação Ser605del. Recentemente, dois estudos independentes identificaram pacientes com a mesma variante de splice homozigótica em POLE1 , a subunidade catalítica de Polε. Um apresentava um fenótipo de dismorfismo facial , imunodeficiência , livedo e baixa estatura (também conhecido como síndrome FILS). O segundo apresentou sintomas mais graves. Esses casos se juntam a um número crescente de defeitos de desenvolvimento associados a mutações hereditárias que visam a função dos genes da polimerase.
Foi observada regulação negativa de POLD1 dependente da idade . embora nenhum significado clínico tenha sido associado a esse fenótipo até o momento. Também estão em andamento estudos para entender se há relação entre essas patologias ou essas mutações e uma predisposição ao câncer. Os mecanismos atualmente propostos pelos quais os defeitos de POLD1 são patogênicos focalizam a ideia de defeitos de replicação levando à instabilidade genômica e ativação de checkpoint, levando à morte celular ou senescência celular. Alternativamente, Polδ está associado a laminas e ao envelope nuclear durante a parada G1 / S ou fase S inicial; mutações em laminas causam lipodistrofias relacionadas ao envelope nuclear com fenótipos semelhantes a MDPL / MDP e síndrome de Werner.
Avaliação de risco de câncer e testes comerciais
Os cânceres colorretais hereditários (CRCs) associados a mutações na capacidade de revisão de POLD1 e POLE são às vezes denominados como " polipose associada à revisão de polipose " (PPAP), (embora pelo menos um estudo tenha identificado mutações de POLD1 associadas a CRC sem polipose). Mutações POLD1 também foram associadas a um aumento da predisposição de câncer de endométrio . Um estudo recente sugeriu diretrizes para testes genéticos para mutações POLD1, que incluem: 1) Ocorrência de 20-100 adenomas e 2) História familiar que atenda aos critérios Amsterdam II para câncer colorretal e endometrial. As diretrizes de testes clínicos atuais para famílias com mutações em POLD1 / PÓLO incluem colonoscopias (a cada 1–2 anos), gastroduodenoscopias (a cada 3 anos) começando cedo (20-25), possibilidade de tumores cerebrais e rastreamento de câncer endometrial (começando aos 40 para mulheres operadoras). Atualmente, estudos estão em andamento para determinar o risco exato de câncer de mutações específicas de POLD1 . Os dados atuais sugerem que as mutações neste gene são altamente penetrantes . Outro estudo recente mostrou que as mutações que afetam as mutações Polδ e Polε podem co-ocorrer junto com as mutações MMR. Isso sugere que o teste do gene do painel deve incluir os genes MMR e Pol, mesmo em pacientes com MSI.
Existem várias opções de teste de diagnóstico comercial para mutações em POLD1. O teste genético inclui tipicamente exons codificadores de POLD1 (26) e pelo menos 20 bases nas regiões não codificantes adjacentes. Para famílias com mutações conhecidas, o teste de local único também está disponível para confirmar a presença de uma mutação. A disponibilidade desses testes genéticos abriu novas possibilidades para cânceres anteriormente classificados como câncer colorretal geneticamente indefinido ou câncer colorretal tipo “X”. Recursos para testes clínicos para MDPL / MDP também foram desenvolvidos.
Notas





