Ribonuclease H - Ribonuclease H
| ribonuclease H | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura cristalográfica de E. coli RNase HI.
| |||||||||
| Identificadores | |||||||||
| EC nº | 3.1.26.4 | ||||||||
| CAS no. | 9050-76-4 | ||||||||
| Bancos de dados | |||||||||
| IntEnz | Vista IntEnz | ||||||||
| BRENDA | Entrada BRENDA | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | Entrada KEGG | ||||||||
| MetaCyc | via metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBsum | ||||||||
| Ontologia Genética | AmiGO / QuickGO | ||||||||
| |||||||||
| ribonuclease H retroviral | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| EC nº | 3.1.26.13 | ||||||||
| Bancos de dados | |||||||||
| IntEnz | Vista IntEnz | ||||||||
| BRENDA | Entrada BRENDA | ||||||||
| ExPASy | NiceZyme view | ||||||||
| KEGG | Entrada KEGG | ||||||||
| MetaCyc | via metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBsum | ||||||||
| |||||||||
A ribonuclease H (abreviada como RNase H ou RNH ) é uma família de enzimas endonucleases não específicas de sequência que catalisam a clivagem de RNA em um substrato de RNA / DNA por meio de um mecanismo hidrolítico . Membros da família RNase H podem ser encontrados em quase todos os organismos, de bactérias a arqueas e eucariotos .
A família é dividida em grupos evolutivamente relacionados com preferências de substrato ligeiramente diferentes , amplamente designados por ribonuclease H1 e H2. O genoma humano codifica H1 e H2. A ribonuclease H2 humana é um complexo heterotrimérico composto por três subunidades, mutações em qualquer uma das quais estão entre as causas genéticas de uma doença rara conhecida como síndrome de Aicardi-Goutières . Um terceiro tipo, intimamente relacionado com H2, é encontrado apenas em alguns procariotos , enquanto H1 e H2 ocorrem em todos os domínios da vida . Além disso, os domínios retrovirais da ribonuclease H do tipo RNase H1 ocorrem em proteínas de transcriptase reversa de múltiplos domínios , que são codificadas por retrovírus como o HIV e são necessárias para a replicação viral.
Em eucariotos, a ribonuclease H1 está envolvida na replicação do DNA do genoma mitocondrial . Tanto H1 quanto H2 estão envolvidos em tarefas de manutenção do genoma, como processamento de estruturas R-loop .
Classificação e nomenclatura
A ribonuclease H é uma família de enzimas endonucleases com uma especificidade de substrato compartilhada para a fita de RNA de duplexes de RNA - DNA . Por definição, as RNases H clivam as ligações fosfodiéster do esqueleto do RNA para deixar um grupo hidroxila 3 ' e um grupo fosfato 5' . RNases H foram propostas como membros de uma superfamília evolutivamente relacionada que abrange outras nucleases e enzimas de processamento de ácido nucleico, como integrases retrovirais , transposases de DNA , resolvases de junção de Holliday , proteínas Piwi e Argonauta , várias exonucleases e a proteína spliceosomal Prp8 .
RNases H podem ser amplamente divididos em dois subtipos, H1 e H2, que por razões históricas recebem designações de algarismos arábicos em eucariotos e designações de algarismos romanos em procariotos . Assim, a Escherichia coli RNase HI é um homólogo do Homo sapiens RNase H1. Em E. coli e em muitos outros procariotos, o gene rnhA codifica HI e o gene rnhB codifica HII. Uma terceira classe relacionada, chamada HIII, ocorre em algumas bactérias e arqueas ; está intimamente relacionado com as enzimas HII procarióticas.
Estrutura
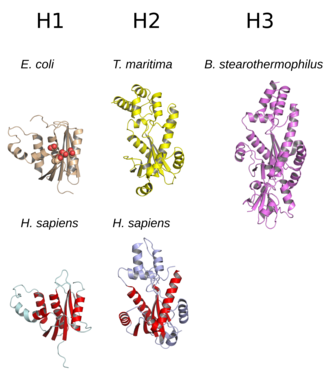
A estrutura da RNase H geralmente consiste em uma folha β de 5 cadeias rodeada por uma distribuição de hélices α . Todas as RNases H têm um sítio ativo centrado em um motivo de sequência conservado composto de resíduos de aspartato e glutamato , frequentemente referido como o motivo DEDD. Esses resíduos interagem com íons de magnésio necessários cataliticamente .
RNases H2 são maiores do que H1 e geralmente têm hélices adicionais. A organização do domínio das enzimas varia; alguns membros procarióticos e a maioria dos eucarióticos do grupo H1 têm um pequeno domínio adicional no terminal N conhecido como "domínio de ligação híbrido", que facilita a ligação ao RNA: duplexes híbridos de DNA e, às vezes, confere processividade aumentada . Enquanto todos os membros do grupo H1 e os membros procarióticos do grupo H2 funcionam como monômeros, as enzimas H2 eucarióticas são heterotrímeros obrigatórios . As enzimas HIII procarióticas são membros do grupo H2 mais amplo e compartilham a maioria das características estruturais com H2, com a adição de um domínio de ligação da caixa TATA N-terminal . Os domínios retrovirais RNase H que ocorrem em proteínas de transcriptase reversa de múltiplos domínios têm estruturas que se assemelham muito ao grupo H1.
As RNases H1 foram amplamente estudadas para explorar as relações entre a estrutura e a atividade enzimática. Eles também são usados, especialmente o homólogo de E. coli , como sistemas modelo para estudar o enovelamento de proteínas . Dentro do grupo H1, foi identificada uma relação entre a maior afinidade de ligação ao substrato e a presença de elementos estruturais que consistem em uma hélice e uma alça flexível fornecendo uma superfície de ligação ao substrato maior e mais básica . A C-hélice tem uma distribuição taxonômica espalhada; está presente nos homólogos de E. coli e RNase H1 humana e ausente no domínio HIV RNase H, mas existem exemplos de domínios retrovirais com hélices C.
Função
As enzimas da ribonuclease H clivam as ligações fosfodiéster do RNA em um híbrido de RNA de fita dupla: DNA, deixando um grupo 3 ' hidroxila e um grupo 5' fosfato em cada extremidade do local de corte com um mecanismo de catálise de íons de dois metais, em que dois cátions divalentes, como Mg2 + e Mn2 +, participam diretamente da função catalítica. Dependendo das diferenças em suas sequências de aminoácidos, essas RNases H são classificadas em RNases H do tipo 1 e 2 RNases H do tipo 1 têm RNases H1 procarióticas e eucarióticas e RNase H. retroviral. As RNases H do tipo 2 têm RNases H2 procarióticas e eucarióticas e RNase H3 bacteriana. Estas RNases H existem na forma monomérica, exceto para as RNases H2 eucarióticas, que existem na forma heterotrimérica. RNase H1 e H2 têm preferências de substrato distintas e funções distintas, mas sobrepostas na célula. Em procariotos e eucariotos inferiores, nenhuma das enzimas é essencial , enquanto ambas são consideradas essenciais em eucariotos superiores. A atividade combinada das enzimas H1 e H2 está associada à manutenção da estabilidade do genoma devido à degradação das enzimas do componente de RNA dos R-loops .
Ribonuclease H1
| Identificadores | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Símbolo | RNase H | ||||||||
| Pfam | PF00075 | ||||||||
| Clã Pfam | CL0219 | ||||||||
| InterPro | IPR002156 | ||||||||
| PRÓSITO | PS50879 | ||||||||
| |||||||||
As enzimas ribonuclease H1 requerem pelo menos quatro pares de bases contendo ribonucleotídeos em um substrato e não podem remover um único ribonucleotídeo de uma fita que é composta de desoxirribonucleotídeos. Por esse motivo, é considerado improvável que as enzimas RNase H1 estejam envolvidas no processamento de primers de RNA de fragmentos de Okazaki durante a replicação do DNA . RNase H1 não é essencial em organismos unicelulares onde foi investigada; em E. coli , nocautes RNase H1 conferem um fenótipo sensível à temperatura, e em S. cerevisiae , eles produzem defeitos na resposta ao estresse.
Em muitos eucariotos, incluindo mamíferos , os genes RNase H1 incluem uma sequência de direcionamento mitocondrial , levando à expressão de isoformas com e sem o MTS presente. Como resultado, a RNase H1 está localizada tanto na mitocôndria quanto no núcleo . Em modelos de camundongos knockout , os mutantes nulos de RNase H1 são letais durante a embriogênese devido a defeitos na replicação do DNA mitocondrial . Os defeitos na replicação do DNA mitocondrial induzidos pela perda de RNase H1 são provavelmente devidos a defeitos no processamento do R-loop .
Ribonuclease H2
| Identificadores | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Símbolo | RNase HII | ||||||||
| Pfam | PF01351 | ||||||||
| Clã Pfam | CL0219 | ||||||||
| InterPro | IPR024567 | ||||||||
| |||||||||
Em procariotos, a RNase H2 é enzimaticamente ativa como uma proteína monomérica. Em eucariotos, é um heterotrímero obrigatório composto por uma subunidade catalítica A e subunidades estruturais B e C. Embora a subunidade A seja homóloga à RNase H2 procariótica, as subunidades B e C não têm homólogos aparentes em procariotos e são mal conservadas em o nível de sequência mesmo entre eucariotos. A subunidade B medeia as interações proteína-proteína entre o complexo H2 e PCNA , que localiza H2 em focos de replicação .
As enzimas H2 procarióticas e eucarióticas podem clivar ribonucleotídeos únicos em uma fita. no entanto, eles têm padrões de clivagem e preferências de substrato ligeiramente diferentes: as enzimas procarióticas têm menor processabilidade e hidrolisam ribonucleotídeos sucessivos de forma mais eficiente do que ribonucleotídeos com um desoxirribonucleotídeo 5 ' , enquanto as enzimas eucarióticas são mais processuais e hidrolisam ambos os tipos de substrato com eficiência semelhante. A especificidade do substrato de RNase H2 confere-lhe um papel no reparo de excisão de ribonucleotídeo , removendo ribonucleotídeos incorporados incorretamente do DNA, além do processamento de R-loop . Embora H1 e H2 estejam presentes no núcleo da célula de mamífero , H2 é a fonte dominante de atividade de RNase H lá e é importante para manter a estabilidade do genoma.
Alguns procariotos possuem um gene adicional do tipo H2 designado RNase HIII na nomenclatura de numeração romana usada para os genes procarióticos. As proteínas HIII estão mais relacionadas ao grupo H2 por identidade de sequência e semelhança estrutural, mas têm preferências de substrato que se assemelham mais a H1. Ao contrário de HI e HII, que são amplamente distribuídos entre procariontes, HIII é encontrado em apenas alguns organismos com uma distribuição taxonômica espalhada; é um pouco mais comum em arquéias e raramente ou nunca é encontrado no mesmo genoma procariótico que HI.
Mecanismo
O sítio ativo de quase todas as RNases H contém quatro resíduos de aminoácidos carregados negativamente, conhecidos como motivo DEDD; frequentemente uma histidina, por exemplo, no HIV-1, humana ou E. coli também está presente.
Os resíduos carregados ligam dois íons metálicos que são necessários para a catálise; sob condições fisiológicas, esses são íons de magnésio , mas o manganês também geralmente suporta a atividade enzimática, enquanto o cálcio ou a alta concentração de Mg2 + inibe a atividade.
Com base em evidências experimentais e simulações de computador, a enzima ativa uma molécula de água ligada a um dos íons metálicos com a histidina conservada. O estado de transição é de natureza associativa e forma um intermediário com o fosfato protonado e o grupo de saída do alcóxido desprotonado. O grupo de saída é protonado através do glutamato, que tem um pKa elevado e é provável que seja protonado. O mecanismo é semelhante à RNase T e à subunidade RuvC na enzima Cas9, que também usam um mecanismo de histidina e de íon de dois metais.
O mecanismo de liberação do produto clivado ainda não foi resolvido. Evidências experimentais de cristalografia resolvida no tempo e nucleases semelhantes apontam para um papel de um terceiro íon na reação recrutada para o sítio ativo.
Em biologia humana
O genoma humano contém quatro genes que codificam RNase H:
- RNASEH1 , um exemplo do subtipo H1 (monomérico)
- RNASEH2A , a subunidade catalítica do complexo trimérico H2
- RNASEH2B , uma subunidade estrutural do complexo trimérico H2
- RNASEH2C , uma subunidade estrutural do complexo trimérico H2
Além disso, o material genético de origem retroviral aparece com frequência no genoma, refletindo a integração dos genomas dos retrovírus endógenos humanos . Tais eventos de integração resultam na presença de genes que codificam a transcriptase reversa retroviral , que inclui um domínio RNase H. Um exemplo é ERVK6 . Repetições terminais longas (LTR) e repita o terminal não-long (non-LTR) retrotransposons também são comuns no genoma e muitas vezes incluem seus próprios domínios RNase H, com uma história evolutiva complexa.
Papel na doença

Em pequenos estudos, as mutações na RNase H1 humana foram associadas à oftalmoplegia externa progressiva crônica , uma característica comum da doença mitocondrial .
As mutações em qualquer uma das três subunidades RNase H2 estão bem estabelecidas como causas de uma doença genética rara conhecida como síndrome de Aicardi-Goutières (AGS), que se manifesta como sintomas neurológicos e dermatológicos em uma idade precoce. Os sintomas da AGS se assemelham aos da infecção viral congênita e estão associados com a regulação positiva inadequado de tipo I interferon . AGS também pode ser causado por mutações em outros genes: TREX1 , SAMHD1 , ADAR e MDA5 / IFIH1, todos os quais estão envolvidos no processamento de ácido nucleico. A caracterização da distribuição mutacional em uma população de pacientes AGS encontrou 5% de todas as mutações AGS em RNASEH2A, 36% em 2B e 12% em 2C. Mutações em 2B foram associadas a comprometimento neurológico um pouco mais brando e com ausência de suprarregulação do gene induzida por interferon que pode ser detectada em pacientes com outros genótipos associados a AGS.
Em vírus
Dois grupos de vírus usam a transcrição reversa como parte de seus ciclos de vida: retrovírus , que codificam seus genomas em RNA de fita simples e se replicam por meio de um intermediário de DNA de fita dupla; e vírus dsDNA-RT , que replicam seus genomas de DNA de fita dupla por meio de um intermediário "pré-genoma" de RNA. Os exemplos patogênicos incluem o vírus da imunodeficiência humana e o vírus da hepatite B , respectivamente. Ambos codificam grandes proteínas multifuncionais de transcriptase reversa (RT) contendo domínios RNase H.
As proteínas retrovirais RT do HIV-1 e o vírus da leucemia murina são os membros da família mais bem estudados. A RT retroviral é responsável por converter o genoma de RNA de fita simples do vírus em DNA de fita dupla. Este processo requer três etapas: primeiro, a atividade da DNA polimerase dependente de RNA produz DNA de fita negativa a partir do modelo de RNA de fita positiva, gerando um intermediário híbrido de RNA: DNA; segundo, a fita de RNA é destruída; e terceiro, a atividade da DNA polimerase dependente de DNA sintetiza DNA de fita mais, gerando DNA de fita dupla como produto final. A segunda etapa deste processo é realizada por um domínio RNase H localizado no terminal C da proteína RT.
A RNase H executa três tipos de ações de clivagem: degradação não específica do genoma do RNA da fita positiva, remoção específica do primer do tRNA da fita negativa e remoção do primer do trato de polipurina rico em purina (PPT) da fita positiva. RNase H desempenha um papel na iniciação da fita positiva, mas não no método convencional de sintetizar uma nova sequência de iniciador. Em vez disso, RNase H cria um "primer" do PPT que é resistente à clivagem de RNase H. Ao remover todas as bases, exceto o PPT, o PPT é usado como um marcador para o final da região U3 de sua longa repetição terminal .
Como a atividade da RNase H é necessária para a proliferação viral, este domínio tem sido considerado um alvo de drogas para o desenvolvimento de drogas anti- retrovirais usadas no tratamento de HIV / AIDS e outras condições causadas por retrovírus. Foram identificados inibidores da RNase H retroviral de vários quimiotipos diferentes , muitos dos quais têm um mecanismo de ação baseado na quelação dos cátions do sítio ativo. Os inibidores da transcriptase reversa que inibem especificamente a função da polimerase da RT estão em uso clínico generalizado, mas não os inibidores da função da RNase H; é a única função enzimática codificada pelo HIV que ainda não é alvo de drogas em uso clínico.
Evolução
As RNases H são amplamente distribuídas e ocorrem em todos os domínios da vida . A família pertence a uma grande superfamília de enzimas de nuclease e é considerada evolutivamente antiga. Em genomas procarióticos, vários genes RNase H estão frequentemente presentes, mas há pouca correlação entre a ocorrência dos genes HI, HII e HIII e as relações filogenéticas gerais , sugerindo que a transferência horizontal de genes pode ter desempenhado um papel no estabelecimento da distribuição dessas enzimas. RNase HI e HIII raramente ou nunca aparecem no mesmo genoma procariótico. Quando o genoma de um organismo contém mais de um gene RNase H, eles às vezes apresentam diferenças significativas no nível de atividade. Essas observações foram sugeridas para refletir um padrão evolutivo que minimiza a redundância funcional entre os genes RNase H. A RNase HIII, que é exclusiva dos procariotos, tem uma distribuição taxonômica espalhada e é encontrada tanto em bactérias quanto em arquéias ; acredita-se que tenha divergido do HII bastante cedo.
A trajetória evolutiva de RNase H2 em eucariotos, especialmente o mecanismo pelo qual homólogos eucarióticos se tornaram heterotrímeros obrigatórios, não é clara; as subunidades B e C não têm homólogos aparentes em procariotos.
Formulários
Como a RNase H degrada especificamente apenas o RNA em híbridos de RNA: DNA de fita dupla, ela é comumente usada como reagente de laboratório em biologia molecular . As preparações purificadas de E. coli RNase HI e HII estão disponíveis comercialmente. A RNase HI é freqüentemente usada para destruir o molde de RNA após a síntese de DNA complementar da primeira fita (cDNA) por transcrição reversa . Também pode ser usado para clivar sequências de RNA específicas na presença de segmentos curtos de DNA complementares. Técnicas altamente sensíveis, como ressonância de plasmão de superfície, podem ser usadas para detecção. A RNase HII pode ser usada para degradar o componente primer de RNA de um fragmento de Okazaki ou para introduzir cortes de fita simples em posições contendo um ribonucleotídeo. Uma variante da PCR de início a quente , conhecida como PCR dependente de RNase H ou rhPCR, foi descrita usando uma RNase HII termoestável do archaeon hipertermofílico Pyrococcus abyssi . Digno de nota, a proteína inibidora da ribonuclease comumente usada como reagente não é eficaz na inibição da atividade de HI ou HII.
História
As ribonucleases H foram descobertas no laboratório de Peter Hausen quando os pesquisadores descobriram a atividade da endonuclease híbrida de RNA: DNA no timo de bezerros em 1969 e deram a ela o nome de "ribonuclease H " para designar sua especificidade de híbrido . A atividade da RNase H foi subsequentemente descoberta em E. coli e em uma amostra de oncovírus com genomas de RNA durante os primeiros estudos de transcrição reversa viral . Mais tarde ficou claro que o extrato de timo de bezerro continha mais de uma proteína com atividade RNase H e que E. coli continha dois genes RNase H. Originalmente, a enzima agora conhecida como RNase H2 em eucariotos foi designada H1 e vice-versa, mas os nomes das enzimas eucarióticas foram trocados para corresponder aos de E. coli para facilitar a análise comparativa, produzindo a nomenclatura moderna em que as enzimas procarióticas são designadas com algarismos romanos e as enzimas eucarióticas com algarismos arábicos. A RNase HIII procariótica, relatada em 1999, foi o último subtipo de RNase H a ser identificado.
A caracterização de RNase H2 eucariótica foi historicamente um desafio, em parte devido à sua baixa abundância. Esforços cuidadosos na purificação da enzima sugeriram que, ao contrário da RNase H2 de E. coli , a enzima eucariótica tinha várias subunidades. O homólogo de S. cerevisiae da proteína de E. coli (isto é, a subunidade H2A) foi facilmente identificável por bioinformática quando o genoma da levedura foi sequenciado, mas a proteína correspondente não apresentou atividade enzimática isolada. Eventualmente, as subunidades B e C da levedura foram isoladas por co-purificação e consideradas necessárias para a atividade enzimática. No entanto, as subunidades B e C da levedura têm uma identidade de sequência muito baixa com seus homólogos em outros organismos, e as proteínas humanas correspondentes foram identificadas de forma conclusiva apenas depois que mutações em todas as três causaram a síndrome de Aicardi-Goutières .
Referências
links externos
- Artigo GeneReviews / NCBI / NIH / UW sobre a Síndrome de Aicardi-Goutières
- RNase + H nos títulos de assuntos médicos da Biblioteca Nacional de Medicina dos EUA (MeSH)


